QSER1
| QSER1 | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifikatori | |||||||||||||||||||||||||
| Aliasi | QSER1 | ||||||||||||||||||||||||
| Vanjski ID-jevi | MGI: 2138986 HomoloGene: 11710 GeneCards: QSER1 | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| Ortolozi | |||||||||||||||||||||||||
| Vrste | Čovjek | Miš | |||||||||||||||||||||||
| Entrez | |||||||||||||||||||||||||
| Ensembl | |||||||||||||||||||||||||
| UniProt | |||||||||||||||||||||||||
| RefSeq (mRNK) | |||||||||||||||||||||||||
| RefSeq (bjelančevina) | |||||||||||||||||||||||||
| Lokacija (UCSC) | Chr 11: 32.89 – 32.99 Mb | Chr 2: 104.59 – 104.65 Mb | |||||||||||||||||||||||
| PubMed pretraga | [3] | [4] | |||||||||||||||||||||||
| Wikipodaci | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
Glutaminom i serinom bogati protein 1 ili QSER1 je protein koji je kodiran genom QSER1.[5]
Protein QSER1 je regulator metilacije DNK.[6] QSER1 ima jednog alijasa, FLJ21924.[5]
Gen
[uredi | uredi izvor]Lokacija
[uredi | uredi izvor]Gen QSER1 nalazi se na kratkom kraku hromosoma 11, sekvenca (11p13), gdje počinje sa 32,914.792. bp i završava na 33,001.816. bp. Dužina je 87.024 bp. Nalazi se između gena DEPDC7 i PRRG4 i 500.000 bp nizvodno od gena za Wilmsov tumor 1 (WT1), koji je upleten u više patoloških promjena.[5][7]
Homologija
[uredi | uredi izvor]Ortolozi
[uredi | uredi izvor]QSER1 je visoko konzerviran u većini vrsta kladusa Chordata. Ortolozi pronađeni su kod primata, ptica, gmizavaca, vodozemaca i riba još u celakantu, koji su se razišli prije 414,9 miliona godina.[5][7]
Paralozi
[uredi | uredi izvor]QSER1 ima jedan paralog kod ljudi, protein 12 bogat prolinom ili PRR12. PRR12 se nalazi na hromosomu 9, na pozicili 9q13.33, a nema poznatu funkciju. PRR12 se nalazi u većini vrsta hordata još u celakantu.[8] Događaj dupliranja vjerovatno se dogodio negdje u lozi hordata blizu divergencije celakanta. I PRR12 i QSER1 sadrže konzervirani domen DUF4211, blizu 3’ krajeva gena.
iRNK
[uredi | uredi izvor]Promotor i transkripcijski faktori
[uredi | uredi izvor]Promotorska regija za QSER1 je duga 683 bp i nalazi se na hromosomu 11 između 32,914.224 bp i 32,914.906 bp. Postoji određeno preklapanje između promotorske regije i 5’ UTR QSER1. Predviđeni transkripcijski faktori sa konzervacijom uključuju (ali nisu ograničeni na) EGR1, p53, E2F3, E2F4, PLAG1, NeuroD2, Myf5, IKZF1, SMAD 3, KRAB, MZF1 i c-Myb.[9]
Ekspresija
[uredi | uredi izvor]Normalna ekspresija
[uredi | uredi izvor]Ekspresija QSER1 je viđena na nivoima nižim od 50% u mnogim tkivima. Međutim, zapažena je u skeletnim mišićima, slijepom crijevu, trigeminusnoj gangliji, stablu malog mozga, ponsu, kičmenoj moždini, cilijarnoj gangliji, globus pallidus, subtalamusnom jezgru, leđnom ganglijskom korijenu, fetusnoj jetri, nadbubrežnim žlijezdama, jajnicima, korpusu maternice, srčanim miocitima pretkomorskokomprskom čvoru, koži, hipofizi, jeziku, ranim eritroidnim progenitorima i krajnicima.[10][11]

Diferencijalna ekspresija
[uredi | uredi izvor]Primijećeno je značajno smanjenje ekspresije QSER1 u bubrežnim mezangijnim ćelijama, kao odgovor na liječenje s 25 mM glukoze. Ovo stanje proučavano je jer je u tim ćelijama zabilježena različita ekspresija gena uključenih u regulaciju ćelijskog ciklusa, kao odgovor na visoke razine glukoze zabilježene kod dijabetes melitus.[12][13]
Druga studija je primijetila prekomjernu ekspresiju QSER1 u patološkoj kardiomiopatiji. Ovo stanje povezano je s promijenjenom ekspresijom gena uključenih u imunoske odgovore, signalizaciju, rast i proliferaciju, kao i infiltraciju B-limfocita.[14][15]
Diferencijalna ekspresija QSER1 se vidi u više stanja raka. Prekomjerna ekspresija QSER1 zabilježena je u Burkittovom limfomu.[10] Ekspresija SER1 se takođe povećava sa povećanjem Gleasonovog skora (naprednije faze) raka prostate.[16] U studiji o odgovoru raka dojke na paklitaksel i fluorouracil-doksorubicin-ciklofosfamidnu hemoterapiju primijećeno je da su linije raka dojke s višim nivoom QSER1 imale veću vjerovatnoću da će odgovoriti na liječenje od onih sa nedovoljno izraženom QSER1.[17]
Veća ekspresija QSER1 također je zabilježena u epitelnim ćelijama dojke imortaliziranih ćelijskih linija, nego u epitelnim ćelijama dojke iz ćelijskih linija s konačnim životnim vijekom.[18]
3’ UTR
[uredi | uredi izvor]Predviđeno je preko 20 petlji peteljke u 3 ’UTR QSER1. U prvih 800 bp od 3 ’UTR pronađeno je 16 petlji peteljke.[19] 3 ’UTR je gotovo u potpunosti konzerviran kod sisara, s manjom konzervacijom u drugih organizama.[20]
Protein
[uredi | uredi izvor]Opća svojstva
[uredi | uredi izvor]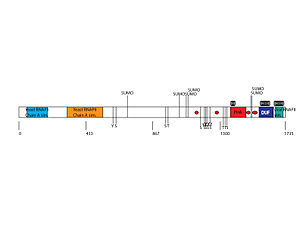
Protein QSER1 dug je 1735 aminokiselina.[21] Sastav peptida je značajno bogat serinom i glutaminom: 14,7% ostataka serina i 8,9% glutamina.[22]
Domeni i motivi
[uredi | uredi izvor]
Protein QSER1 sadrži dva visoko konzervirana domena koja se nalaze ne samo u QSER1 već i u drugim proteinima. To uključuje domen PHA02939 sa aminokiselinama između 1380-1440 i domen DUF4211 od aminokiselina 1522-1642.[23][24] Jedarnu lokalizaciju predvidio je pSORT. Ovo svojstvo je konzervirano od ljudskog QSER1 do celakantnog QSER1. PSORT je također predvidio više konzerviranih signala jedarne lokalizacije unutar proteina QSER1.[25]
Aminokiselinska sekvenca
[uredi | uredi izvor]Dužina polipeptidnog lanca je 1.735 aminokiselina, a molekulska težina 189.972 Da.[26]
| 10 | 20 | 30 | 40 | 50 | ||||
|---|---|---|---|---|---|---|---|---|
| MNFLSTAESR | TAQAAASGTT | LLPQFRAPSW | QTGMHSSAAT | ELFATGPLPS | ||||
| TGTLPPSLSA | YQHPTTFSNR | NFATTSPLVL | QDSTFNTTSN | GILSHHDPLL | ||||
| QIKTSQGTVP | TALAFERLGS | SVLSNSIPPQ | SSTYRSAQES | APHLLQPQFS | ||||
| LLPSALGGSQ | QTPQAYSSTL | FTSSTASIER | ALLRECSVIK | HHQRPSGTQS | ||||
| IQAQLTGSQH | SLHSYLSNSS | VVNFQETTRQ | SSLSCSPIGD | STQVSNGGLQ | ||||
| QKTSQVSVEL | AQSYSSAIPS | SGYPPSTTKI | KSCSTEQPLT | STKTPKPQSI | ||||
| IPPVQTLSYS | KPLHNQSSVI | SGQAQIYSTA | QLPSLLSVSQ | SQNYGLVQPH | ||||
| NVPSIVHSQV | YRSSKVEKLP | PLYKTLTFSG | SSQTVTPENQ | TLNYSSNQQE | ||||
| VLSSVTNENY | PAQTRDLSSV | SQSQSYSSGH | SQGLSPVSQT | QVSYSSQSQV | ||||
| LSVVSLSESY | ASGESLTLTA | PSLSYSSASR | AQNLPDSSPT | QNYISMHSSQ | ||||
| NVQTQESSSP | QSQKFLPAVQ | SSSFASSTHC | QTLQNNITSP | DPKSYAERKL | ||||
| DSDVYPSSKQ | EDGFPMQELQ | VLQPQASLES | STQRLSDGEI | NAQESTYKVS | ||||
| KADDRYSQSV | IRSNSRLEDQ | VIGVALQASK | KEESVVGSVT | QLNQQIGQVN | ||||
| NAATLDLKNS | TNLIQTPQIR | LNTKDLKQQH | PLILKVHESK | VQEQHDQIIN | ||||
| ASSQIQIPNH | ALGHGHQASL | PNTQVLLDSA | CDLQILQQSI | LQAGLGQVKA | ||||
| SLQAQRVQSP | QQIVHPFLQM | EGHVIQSNGD | HSQQQLHPQN | SEVMKMDLSE | ||||
| SSKPLQQHLT | TKGHFSETNQ | HDSKNQFVSL | GSMCFPEAVL | LSDERNILSN | ||||
| VDDILAATAA | ACGVTPTDFS | KSTSNETMQA | VEDGDSKSHF | QQSLDVRHVT | ||||
| SDFNSMTATV | GKPQNINDTS | LNGNQVTVNL | SPVPALQSKM | TLDQQHIETP | ||||
| GQNIPTKVTS | AVVGPSHEVQ | EQSSGPFKKQ | SATNLESEED | SEAPVDSTLN | ||||
| NNRNQEFVSS | SRSISGENAT | SESEFTLGGD | DSGVSMNPAR | SALALLAMAQ | ||||
| SGDAVSVKIE | EENQDLMHFN | LQKKRAKGKG | QVKEEDNSNQ | KQLKRPAQGK | ||||
| RQNPRGTDIY | LPYTPPSSES | CHDGYQHQEK | MRQKIKEVEE | KQPEVKTGFI | ||||
| ASFLDFLKSG | PKQQFSTLAV | RMPNRTRRPG | TQMVRTFCPP | PLPKPSSTTP | ||||
| TPLVSETGGN | SPSDKVDNEL | KNLEHLSSFS | SDEDDPGYSQ | DAYKSVSTPL | ||||
| TTLDATSDKK | KKTEALQVAT | TSPTANTTGT | ATTSSTTVGA | VKQEPLHSTS | ||||
| YAVNILENIS | SSESSKPIEL | DGLPSDQFAK | GQDTVAIEGF | TDEEDTESGG | ||||
| EGQYRERDEF | VVKIEDIETF | KEALKTGKEP | PAIWKVQKAL | LQKFVPEIRD | ||||
| GQREFAATNS | YLGYFGDAKS | KYKRIYVKFI | ENANKKEYVR | VCSKKPRNKP | ||||
| SQTIRTVQAK | PSSSSKTSDP | LASKTTTTKA | PSVKPKVKQP | KVKAEPPPKK | ||||
| RKKWKEEFSS | SQSDSSPEIH | TSSSDDEEFE | PPAPFVTRFL | NTRAMKETFK | ||||
| SYMELLVSIA | LDPDTMQALE | KSNDELLLPH | MKKIDGMLND | NRKRLLLNLH | ||||
| LDQSFKNALE | SFPELTIITR | DSKAKSGGTA | ISKIKMNGKA | YNKKTLRTSK | ||||
| TTTKSAQEFA | VDPEKIQLYS | LYHSLHHYKY | HVYLICKDEI | SSVQKKNEDL | ||||
| GQEEIVQLCM | KNVKWVEDLF | EKFGELLNHV | QQKCS |
Struktura
[uredi | uredi izvor]Predviđanja strukture proteina QSER1 ukazuju da protein sadrži mnogo alfa-heliksa.[27][28][29] NCBI cBLAST predvidio je strukturnu sličnost između proteina QSER1 i Schizosaccharomyces pombe (fisijski kvasac) RNK-polimeraza II A. Dvije regije sličnosti javljaju se između aminokiselina 56-194 i 322-546.[28] Ova prva regija (56-194) je regulatorna regija i u RNA polimerazi II čovjeka i kvasca koja sadrži više ponavljanja sekvence YSPTSPSYS. Fosforilacija ostataka serina u ovoj regiji regulira progresiju kroz korake transkripcije gena.[30] Za ovu regiju osigurana je 3D struktura. Strukturno slično područje nalazi se na vanjskoj strani molekule proteina i čini dio rascjepa za vezanje DNK.
Dalja strukturna sličnost sa proteinom virusne RNK koja veže polimerazu je predviđena prema Phyre2. Ova struktura se nalazi na samom kraju proteina, između aminokiselina 1671 i 1735. Struktura ima dugu regiju alfa-heliksa, što je također predvidio SDSC Biology Workbench PELE. Slika strukturno sličnog područja i poravnavanja sekvence prikazana je s desne strane. Regije prije identificiranog strukturno sličnog domena pokazuju dva druga alfa-heliksa, predviđena s velikom pouzdanošću.[29]
Konzervacija
[uredi | uredi izvor]Protein QSER1 je visoko konzerviran u vrstama hordata. Donja tabela prikazuje informacije o proteinskim ortolozima.

| Rod i vrsta | Uobičajeno ime | Pristupni NCBI proteinski broj[31] | Identitet sekvence sa ljudskim proteinom [31] |
|---|---|---|---|
| Homo sapiens | Čovjek | NP_001070254.1 | |
| Pan troglodytes | Čimpanza | XP_508354.3 | 99% |
| Macaca mulatta | Rezus-makak | NP_001244647.1 | 98% |
| Callithris jacchus | Marmozet | XP_002755192.1 | 96% |
| Ailuropoda melanoleuca | Gigantska panda | XP_002917539.1 | 90% |
| Loxodonta africana | Slon | XP_003412344.1 | 88% |
| Mus musculus | Miš | NP_001116799.1 | 81% |
| Monodelphis domestica | Oposum | XP_001368629.1 | 71% |
| Ornithorhynchus anatinus | Kljunar | XP_001506659.2 | 75% |
| Taeniopygia guttata | Zebrasta zeba | XP_002195876.1 | 69% |
| Gallus gallus | Kokoš | NP_001186343.1 | 69% |
| Anolis carolinensis | Karolinska anola (gušter) | XP_003214747.1 | 62% |
| Takifugu rubripes | Japanska pufer-riba | XP_003977915.1 | 48% |
| Latimeria chalumnae | Latimerija | N/A | 62% |

Postoji 12 potvrđenih mjesta fosforilacije proteina QSER1. Osam su fosfoserini, jedan fosfotirozin i tri fosfotreonina. Pokazalo se da su tri od ovih mjesta fosforilirana ATM-om i ATR-om, kao odgovor na oštećenje DNK.[32] Ostala 123 moguća mjesta fosforilacije predviđena su pomoću alata ExPASy NetPhos.[33]
SUMOilacija
[uredi | uredi izvor]Interakcija QSER1 proteina sa SUMO zabilježena je u više proteinskih širokih studija.[34][35] Predviđena mjesta SUMOilacije pronađena su u proteinu QSER1. Visoko konzervirana mjesta SUMOilacije javljaju se sa sekvencom MKMD na aminokiselini 794, VKIE na 1057, VKTG na 1145, LKSG na 1157, VKQP na 1487 i VKAE na 1492.[36]

Interakcije
[uredi | uredi izvor]ATM/ATR
[uredi | uredi izvor]Fosforilacija QSER1 u tri serinska ostatka, S1228, S1231 i S1239, pomoću ATM-a i ATR-a kao odgovor na oštećenje DNA pronađena je u proteom-širokoj studiji.[32]
SUMO
[uredi | uredi izvor]Interakcija QSER1 sa protzeinom SUMO potvrđena je u više studija.Uloga SUMOlacija u funkciji QSER1 nije jasna. Međutim, može postojati veza između QSER1 i SUMO kao odgovor na endoplazmatskoretikulumski (ER) stres (često uzrokovan nakupljanjem pogrešno savijenih proteina). U studiji o ER stresu, QSER1 je označen kao gen za odgovor na stres na ER sa promijenjenom ekspresijom.[37] Nadalje, u studiji o SUMOilaciji kao odgovor na akumulirane pogrešno savijene proteine i ER stres utvrđeno je da je QSER1 SUMO interaktant u ovoj situaciji. Svaka veza između ove dvije aktivnosti nije proučena i nepotvrđena.
Direktna interakcija QSER1 s RNK-polimerazom II pronađena je u studiji koju su proveli Moller et al. Pokazalo se da dolazi do interakcije sa podjedinicom DNK usmjerene RNK-polimeraze II, RPB1, RNK- polimeraze II, tokom mitoza i interfaza. Kolokalizacija/interakcija QSER1 pokazana je u regulatornoj regiji RPB1 sa 52 ponavljanja heptapeptida (YSPTSPSYS).[30]
NANOG i TET1
[uredi | uredi izvor]Pokazalo se da je interakcija između homeoboksnog proteina NANOG i tet metilcitozin-dioksigenaze 1 (TET1) važna za uspostavljanje pluripotencije tokom stvaranja indukovanih pluripotentnih matičnih ćelija. Protein QSER1 stupa u interakciju s NANOG-om i TET1-om.[38]
Ubikvitin
[uredi | uredi izvor]Utvrđeno je da QSER1 stupa u interakciju s ubikvitinom, u dvije studije supstrata na cijelom proteomu.[39][40] Posebni detalji o ovoj interakciji nisu proučavani.
Patologija
[uredi | uredi izvor]Promijenjena ekspresija QSER1 zabilježena je kod patološke kardiomiopatije, Burkittovog limfoma, raka prostate i nekih gore navedenih karcinoma dojke.[9][10][14][16] NCBI AceView navodi više mutacija povezanih s drugim patološkim promjenama, uključujući osam parova baza i 13 delecija baznih parova u QSER1 povezanih s leiomiosarkomom maternice, te 57 parova baza razlika u neuroblastomu. Navedene su i više varijanti prerade sa skraćenim krajevima 5 'i/ili 3' koji se često primjećuju u kancerogenim stanjima.[41] Nadalje, prema bazi podataka NCBI OMIM, više patoloških stanja povezano je s promjenama u regiji 11p13 i stoga može uticati na QSER1.[42] To uključuje eksudativnu vitreoretinopatiju 3,[43] porodičnu kandidijazu 3,[44] centralopatsku epilepsiju,[45] autosomno recesivnu gluhoću 51.[46] QSER1 je također zabilježen kao gen osjetljivosti na Parkinsonovu bolest.[37]
Reference
[uredi | uredi izvor]- ^ a b c GRCh38: Ensembl release 89: ENSG00000060749 - Ensembl, maj 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000074994 - Ensembl, maj 2017
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ a b c d "NCBI QSER1 Gene".
- ^ Dixon, Gary; Pan, Heng; Yang, Dapeng; Rosen, Bess P.; Jashari, Therande; Verma, Nipun; Pulecio, Julian; Caspi, Inbal; Lee, Kihyun; Stransky, Stephanie; Glezer, Abigail; Liu, Chang; Rivas, Marco; Kumar, Ritu; Lan, Yahui; Torregroza, Ingrid; He, Chuan; Sidoli, Simone; Evans, Todd; Elemento, Olivier; Huangfu, Danwei (2021). "QSER1 protects DNA methylation valleys from de novo methylation". Science. 372 (6538): eabd0875. doi:10.1126/science.abd0875. ISSN 0036-8075.
- ^ a b "Genecards QSER1".
- ^ "NCBI PRR12 Gene".
- ^ a b "Genomatix Tools: El Dorado". Arhivirano s originala, 2. 12. 2021. Pristupljeno 25. 8. 2021.
- ^ a b c "NCBI GeoProfiles db; QSER1 GDS596".
- ^ "NCBI EST Profile db; QSER1".
- ^ Clarkson MR, Murphy M, Gupta S, Lambe T, Mackenzie HS, Godson C, Martin F, Brady HR (Mar 2002). "High glucose-altered gene expression in mesangial cells. Actin-regulatory protein gene expression is triggered by oxidative stress and cytoskeletal disassembly". The Journal of Biological Chemistry. 277 (12): 9707–12. doi:10.1074/jbc.M109172200. PMID 11784718.
- ^ "NCBI GeoProfiles db; QSER1 GDS1891".
- ^ a b Galindo CL, Skinner MA, Errami M, Olson LD, Watson DA, Li J, McCormick JF, McIver LJ, Kumar NM, Pham TQ, Garner HR (9 Dec 2009). "Transcriptional profile of isoproterenol-induced cardiomyopathy and comparison to exercise-induced cardiac hypertrophy and human cardiac failure". BMC Physiology. 9 (23): 23. doi:10.1186/1472-6793-9-23. PMC 2799380. PMID 20003209.
- ^ "NCBI GeoProfiles db; QSER1 GDS3596".
- ^ a b "NCBI GeoProfiles db; QSER1 GDS1746".
- ^ "NCBI GeoProfiles db; QSER1 GDS3721".
- ^ "NCBI GeoProfiles db; QSER1 GDS2810".
- ^ "mfold".
- ^ "SDSC Biology Workbench ClustalW".
- ^ "NCBI QSER1 Protein".
- ^ "SDSC Biology Work Bench; SAPS".
- ^ "NCBI Conserved domains db; DUF4211".
- ^ "NCBI Conserved domains db; PHA02939".
- ^ "pSORT II Prediction".
- ^ "UniProt, Q2KHR3". Pristupljeno 25. 8. 2021.
- ^ "SDSC Biology Workbench; PELE".
- ^ a b "NCBI cBLAST; QSER1".
- ^ a b "Phyre2". Arhivirano s originala, 25. 8. 2021. Pristupljeno 25. 8. 2021.
- ^ a b Möller A, Xie SQ, Hosp F, Lang B, Phatnani HP, James S, Ramirez F, Collin GB, Naggert JK, Babu MM, Greenleaf AL, Selbach M, Pombo A (Jun 2012). "Proteomic analysis of mitotic RNA polymerase II reveals novel interactors and association with proteins dysfunctional in disease". Molecular & Cellular Proteomics. 11 (6): M111.011767. doi:10.1074/mcp.M111.011767. PMC 3433901. PMID 22199231.
- ^ a b "NCBI National Center for Biotechnology Information".
- ^ a b Matsuoka S, Ballif BA, Smogorzewska A, McDonald ER, Hurov KE, Luo J, Bakalarski CE, Zhao Z, et al. (maj 2007). "ATM and ATR substrate analysis reveals extensive protein networks responsive to DNA damage". Science. 316 (5828): 1160–6. Bibcode:2007Sci...316.1160M. doi:10.1126/science.1140321. PMID 17525332. S2CID 16648052.
- ^ "ExPASy NetPhos".
- ^ Tatham MH, Matic I, Mann M, Hay RT (21 Jun 2011). "Comparative proteomic analysis identifies a role for SUMO in protein quality control". Science Signaling. 4 (178): rs4. doi:10.1126/scisignal.2001484. PMID 21693764. S2CID 649212.
- ^ Bruderer R, Tatham MH, Plechanovova A, Matic I, Garg AK, Hay RT (Feb 2011). "Purification and identification of endogenous polySUMO conjugates". EMBO Reports. 12 (2): 142–8. doi:10.1038/embor.2010.206. PMC 3049431. PMID 21252943.
- ^ "ExPASy SUMOplot".
- ^ a b Dombroski BA, Nayak RR, Ewens KG, Ankener W, Cheung VG, Spielman RS (maj 2010). "Gene expression and genetic variation in response to endoplasmic reticulum stress in human cells" (PDF). American Journal of Human Genetics. 86 (5): 719–29. doi:10.1016/j.ajhg.2010.03.017. PMC 2869002. PMID 20398888. Arhivirano s originala (PDF), 4. 10. 2013. Pristupljeno 2. 5. 2013.
- ^ Costa Y, Ding J, Theunissen TW, Faiola F, Hore TA, Shliaha PV, Fidalgo M, Saunders A, Lawrence M, Dietmann S, Das S, Levasseur DN, Li Z, Xu M, Reik W, Silva JC, Wang J (Mar 2013). "NANOG-dependent function of TET1 and TET2 in establishment of pluripotency". Nature. 495 (7441): 370–4. Bibcode:2013Natur.495..370C. doi:10.1038/nature11925. PMC 3606645. PMID 23395962.
- ^ Kim W, Bennett EJ, Huttlin EL, Guo A, Li J, Possemato A, Sowa ME, Rad R, Rush J, Comb MJ, Harper JW, Gygi SP (Oct 2011). "Systematic and quantitative assessment of the ubiquitin-modified proteome". Molecular Cell. 44 (2): 325–40. doi:10.1016/j.molcel.2011.08.025. PMC 3200427. PMID 21906983.
- ^ Danielsen JM, Sylvestersen KB, Bekker-Jensen S, Szklarczyk D, Poulsen JW, Horn H, Jensen LJ, Mailand N, Nielsen ML (Mar 2011). "Mass spectrometric analysis of lysine ubiquitylation reveals promiscuity at site level". Molecular & Cellular Proteomics. 10 (3): M110.003590. doi:10.1074/mcp.M110.003590. PMC 3047152. PMID 21139048.
- ^ "NCBI AceView db; QSER1".
- ^ "NCBI OMIM db; 11p13".
- ^ "NCBI OMIM db; Exudative Vitreoretinopathy 3".
- ^ "NCBI OMIM db; Candidiasis, Familial 3".
- ^ "NCBI OMIM db; Centralopathic Epilepsy".
- ^ "NCBI OMIM db; Autosomal Recessive Deafness 51".




