UniProt
|
|
L'article o secció necessita millores deformat. |
 | |
 | |
| URL | https:// uniprot.org/ |
|---|---|
| Tipus | Base de dades de seqüències de proteïnes |
| Tema | biologia,ésser humà,ratolí comú,growth hormone releasing hormone(en) |
| Llengua | anglès |
| Llicència | Domini públic |
| Part de | web semàntic,diagrama de dades obertes enllaçades,SIB Swiss Institute of Bioinformatics(en) |
| Creador | Consorci UniProt: EIB, SIB i PIR |
| Mantenidor | Alex Bateman,Sandra Orchard(en) |
| Llançament | 2002 |
| Estat | Regne Unit,SuïssaiEstats Units d'Amèrica |
| Estat | Actiu |
UniProt (Universal Protein Resource) és unabase de dadesde seqüències deproteïnesi la seva corresponent informació funcional. És de lliure accés i conté moltes entrades derivades de projectes deseqüenciacióde genomes. Conté al voltant de 60 milions de seqüències de proteïnes, derivada de la literatura científica, sobre la funció biològica de les proteïnes, la qual s’actualitza a mesura que es genera més coneixement.[1][2]
Uniprot neix delconsorci UniProtque està format perEBI(European Bioinformatic Institute), SIB (Swiss Institute of Bioinformatics), organitzacions bioinformàtiques europees i PIR (Protein Information Resource) organització americana de dades de proteïnes. UniProt ofereix accés a quatre bases de dades de proteïnes:The UniProt Knowledgebase(UniProtKB),The UniProt Reference Clusters(UniRef),The UniProt Metagenomicsand Environmental Sequences database(UniMES) i The UniProt Archive (UniParc).[1][2]
Consorci UniProt
[modifica]Elconsorci UniProtcomprèn l'Institut Europeu de Bioinformàtica(EBI), l'Institut Suís de Bioinformàtica(SIB) i elRecurs d'Informació de Proteïnes(PIR).
- L'EBIestà ubicat alWellcome Trust Genome Campusa Hinxton, Regne Unit i comprèn un gran recurs de bases de dades i serveis de bioinformàtica.
- ElSIBestà ubicat a Ginebra, Suïssa i manté els servidorsExPASy(Expert Protein Analysis System) que són un recurs central per a eines i bases de dades de proteòmica.
- ElPIRestà organitzat per la Fundació Nacional de Recerca Biomèdica (NBRF) al centre mèdic universitari de Georgetown a Washington, DC, Estats Units. És l’hereu de la base de dades de seqüències de proteïnes més antiga: l’Atlas de seqüència i estructura de proteïnes, deMargaret Dayhoff,publicada per primer cop el 1965.[3]
L’any 2002, EBI, SIB i PIR es van unir formant el consorci UniProt.[4]
Les arrels de les bases de dades UniProt
[modifica]Cadascun dels membres del consorci està molt implicat en el manteniment i l’anotació de bases de dades de proteïnes. Fins fa poc, EBI i SIB produïen de manera conjunta les bases de dadesSwiss-ProtiTrEMBL,mentre que PIR produïa la base de dades de seqüències de proteïnes (PIR-PSD). Aquestes bases de dades coexistien amb diferents prioritats de cobertura i anotació deseqüències de proteïnes.[5][6][7]
Swiss-Prot va ser creat inicialment l’any 1986 perAmos Bairochdurant el seu doctorat i desenvolupat per l'Institut Suís de Bioinformàtica,posteriorment va ser desenvolupat perRolf Apweiler a l'Institut Europeu de Bioinformàtica.[8][9]L’objectiu que pretenia complir Swiss-Prot era el de proporcionar seqüències proteiques fiables associades a un alt nivell d’anotació, com per exemple la descripció de la funció d’una proteïna, la seva estructura dedomini,modificacions post-traduccionals,variants, etc., un nivell mínim deredundànciai un nivell d’integració amb altres bases de dades. Les dades de seqüències s’estaven creant a un ritme superior a la capacitat de Swiss-Prot, per mantenir-se al dia, aleshores es va crear TrEMBL (Translated EMBL Nucleotide Sequence Data Library), per tal de proporcionar anotacions automàtiques a les proteïnes que no apareixien a Swiss-Prot. Al mateix temps, PIR mantenia el PIR-PSD i bases de dades relacionades, inclosaiProClass,una base de dades de seqüències de proteïnes i famílies seleccionades.
Els membres del consorci van agrupar els seus recursos i coneixements superposats i van llançar UniProt el desembre de 2003.[10]

Organització de bases de dades UniProt
[modifica]Sota el dominiUniProttrobem tres bases de dades bàsiques:UniProtKB(amb els sub-partsSwiss-ProtiTrEMBL),UniParciUniRef.
| UniProtKB/Swiss-Prot | UniProtKB/TrEMBL | UniParc | UniRef | |
|---|---|---|---|---|
| Revisió | Manual | Automàtica | Automàtica | Automàtica |
| Informació[11] | 550.000 seqüències | 60 milions de seqüències | Seqüènices que UniProtKB no considera prou importants: descatalogades, pseudogens, etc (desenvolupat a continuació). | Seqüències agrupades en clústers, amaga la redundància |
UniProtKB
[modifica]
UniProt Knowledgebase(UniProtKB) és una base de dades de proteïnes revisada per experts, que consta de dues seccions. La primera ésSwiss-Proti conté entrades revisades i anotades manualment per revisors. En segon lloc, hi haTrEMBL,que conté entrades no revisades per revisors però automàticament anotades. Així doncs, la diferència principal entre els dos webs és si el mètode de revisió de les seqüències és manual o automàtic.[12]
Origen de les dades d'UniprotKB
[modifica]Les seqüències de proteïnes d'UniprotKB provenen de latranscripcióde les seqüències denucleòtidscodificants que proporciona EMBL-Bank/GeneBank/DDBJ. Al seu torn, aquestes seqüències són obtingudes a través de treball experimental o per programes de predicció de gens. A totes les seqüències traduïdes se’ls hi assigna un identificador que és elmateixen el web original d'EMBL-Bank/GeneBank/DDBJ i a UniProtKB.[13]
La necessitat de crear TrEMBL prové de la rapidesa en què es generen noves seqüències queimpossibilita la revisió exhaustivade cadascuna d’elles. Com a solució, es va crear TrEMBL que emmagatzema seqüències revisades per mètodes computacionals d’alta qualitat. Aquests mètodes fins i tot permeten dur a terme classificacions i anotacions automàtiques. Presenten l’avantatgeque poden fer-se públiques les noves seqüències amb més rapidesa i eldesavantatgeque no estan revisades manualment i per tant la qualitat de les dades és inferior. Convé mantenir unaseparacióentre els dos tipus de dades perquè no es dilueixin les dades d’alta qualitat.[13]
UniProtKB/Swiss-Prot
[modifica]UniProtKB/Swiss-Protés una base de dades de seqüències de proteïnes no redundants anotadesmanualment.Combina informació extreta de literatura científica i anàlisi computacional revisada per revisors. L'objectiu d'UniProtKB/Swiss-Prot és proporcionar tota la informació rellevant coneguda sobre una proteïna en particular. L'anotació esrevisa regularmentper mantenir-se al dia amb els descobriments científics actuals. L'anotació manual d'una entrada implica una anàlisi detallada de la seqüència de proteïnes i de la literatura científica.[14]
Les seqüències del mateixgeni de la mateixaespèciees combinen en la mateixa entrada de base de dades. S’identifiquen les diferències entre seqüències i es documenta la seva causa que poden ser per exemple:empalmament alternatiu,variació natural, llocs d’iniciació incorrectes, límits d'exonsincorrectes, desplaçaments de marcs de lectura, conflictes no identificats. Les prediccions computacionals s’avaluen manualment i es seleccionen els resultats rellevantsper incloure'ls a l'entrada. Aquestes prediccions inclouen modificacions post-traduccionals,doministransmembrana i topologia,pèptids senyal,identificació de dominis i classificació de famílies de proteïnes.[14]
Les publicacions rellevants s’identifiquen cercant en bases de dades comPubMed.Es llegeix el text complet de cada article i s’extreu informació i s’afegeix a l'entrada. L'anotació sorgida de la literatura científica inclou, entre d'altres:
| Trets que s'inclouen en l'anotació |
|---|
| Noms de proteïnes i gens |
| Funció |
| Informació específica d'enzims:activitat catalítica,cofactors,iresidus catalítics |
| Localització subcel·lular |
| Interacció entre proteïnes |
| Patrons d’expressió |
| Localització i rols de dominis i llocs importants |
| Ions,substratsi llocs d’unió de cofactor |
| Formes variants de les proteïnes produïdes per variació genètica natural,modificació de RNA,splicing alternatiu, processosproteolíticsi modificacions post-transcripcionals |
Cada seqüència és revisada abans de ser inclosa en UniProtKB/Swiss-Prot i s’actualitza cada entrada quan hi ha informació nova.[14]
UniProtKB/TrEMBL
[modifica]UniProtKB/TrEMBLconté les seqüències transcrites a partir de seqüències dEMBL-Bank/GeneBank/DDBJque són processades automàticament i dirigides a UniProtKB/TrEMBL. A més també inclou seqüències de PDB i de predicció de gens comEnsembl,RefSeqiCCDS.[13]
Anotació automàtica
[modifica]El sistema computacional que permet l’anotació automàtica deseqüències no revisades manualment,permet augmentar la qualitat de les mateixes. El programa que utilitzen per a la classificació i anotació de dominis automàtica ésInterPro,que permet fer una anàlisi funcional de les seqüències que inclou: classificar-les en famílies, predir dominis inucleòtidsimportants. InterPro es basa en models predictius que provenen de diverses bases de dades que conformen el consorci InterPro.[15]Per altra banda, l'anotació automàtica es du a terme per dos sistemes:UniRulei ARBA (Association-Rule-Based Annotator). UniRule permet anotar:[16]
| UniRule |
|---|
| Nom de la proteïna |
| Funció |
| Activitat catalítica |
| Localització subcel·lular |
| Posicions on es duen a terme modificacions post-transcripcionals |
| Llocs catalítics |
ARBA utilitzaaprenentatge automàticper crear les anotacionsmés precisesbasant-se en llargada de la seqüènecia, InterPro group membership i taxonomia. Això permet anotar propietats de la proteïna excloent el nom de la proteïna i caràcters que requereixen de predicció.[17]
Seqüències excloses d'UniprotKB
[modifica]UnProtKB conté la majoria de seqüències proteiques obertes al públic peròexcloules següents seqüències:[18]
| Seqüències excloses |
|---|
| Immunoglobulinesi receptors decèlules T |
| Seqüències sintètiques |
| Seqüències que s’apliquen en patents |
| Seqüències inferiors a 8aminoàcids |
| Pseudogens |
| Seqüències de proteomes redundants |
| Seqüències que RefSeq i NCBI genomes considera de baixa qualitat |
| Proteïnes de fusióo truncades |
| Proteïnes no reals |
Algunes d’aquestes seqüències a vegades poden no ser reconegudes pels programes automàtics d’anotació i ser accidentalment incolses a UniProtKB. Per aquest motiu cal que, per exemple, el tipus 8 i 9 siguinexcloses manualmentper revisors. Totes les excloses acaben a la secció UniParc.[18]
UniParc
[modifica]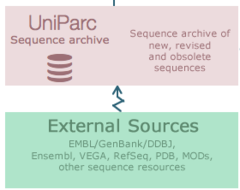
UniProt Archive(UniParc) és una bases de dades integral i no redundant, conté totes les seqüències de proteïnes de les principals bases de dades disponibles públicament. Les proteïnes poden existir en diverses bases de dades de fonts diferents i en diverses còpies a la mateixa base de dades. Per tal d’evitar la redundància, UniParcnomés emmagatzema una vegada cada seqüènciaúnica.[19]
Es combinen seqüències idèntiques independentment de que provinguin de la mateixa espècie o no. A cada seqüència se li proporciona un identificador únic i estable (UPI), quepermet identificar la mateixa proteïnaa partir de diferents bases de dades d'origen. UniParc només conté seqüències de proteïnes, sense cap anotació. Les referències creuades de bases de dades a les entrades UniParc permeten obtenir més informació sobre la proteïna de les bases de dades d'origen. Quan canvien les seqüències de les bases de dades d'origen, aquests canvis són rastrejats per UniParc i s'arxiva a l'historial de tots els canvis.[20]
Actualment, UniParc conté seqüències de proteïnes de les següents bases de dades disponibles:
| Bases de dades d'on s'extreu la informació: |
|---|
| Bases de dades de nucleòtids:INSDC,EMBL-Bank/DDBJ/GenBank |
| Ensembl |
| European Patent Office(EPO) |
| FlyBase: the primary repository of genetic and molecular data for the insect family Drosophilidae(FlyBase) |
| H-Invitational Database(H-Inv) |
| International Protein Index(IPI) |
| Japan Patent Office(JPO) |
| Protein Information Resource(PIR-PSD) |
| Protein Data Bank(PDB) |
| Protein Research Foundation(PRF) |
| RefSeq |
| Saccharomyces Genome Database(SGD) |
| The Arabidopsis Information Resource(TAIR) |
| TROME |
| US Patent Office(USPTO) |
| UniProtKB/Swiss-Prot, UniProtKB/Swiss-Prot protein isoforms, UniProtKB/TrEMBL |
| Vertebrate and Genome Annotation Database(VEGA) |
| WormBase |
UniRef
[modifica]
UniRef fa referència alsclústersde referència d'UniProt que consisteixen en tres bases de dades de conjunts agrupats de seqüències de proteïnes d’UniProtKB (incolent les isoformes) i registres seleccionats d’UniParc. Això amaga les seqüències redundants i obté una cobertura de l'espai de la seqüència a tres dimensions.[21]
- UniRef100:combina seqüències idèntiques i fragments de seqüència amb 11 o més residus de qualsevolorganismeen una sola entrada d'UniRef. Es mostren: la seqüència d’una proteïna representativa, elsnúmeros d’accésde totes les entrades combinades i els enllaços als corresponents registres UniProtKB i UniParc.
Les seqüències UniRef100 s’agrupen mitjançant l'algorismeCD-HIT per construir UniRef90 i UniRef50.
- UniRef90:és construida a partir de l’agrupació de seqüències UniRef100 utilitzant l'algorismeMMseqs2de manera que cada clúster està compost per seqüències que tenen un 90% d’identitat de seqüència i un 80% de superposició amb la seqüència més llarga del clúster.[22][23]
- Uniref50:és construida a partir de l’agrupació de seqüències de UniRef90 amb un mínim d’identitat del 50% i un 80% de superposició amb la seqüència més llarga del clúster.[23]
Abans del 2013, no hi ha havia cap llindar de superposició, pel que els clústers tenien una longitud més heterogènia. UniRef90 i UniRef 50 van produir una reducció de mida de les bases de dades d'aproximadament un 58% i un 79% respectivament. L’agrupació de seqüències redueix significativament la mida de la base de dades i permet fer cerques de seqüències més ràpides.[23]
Cada clúster es compon de seqüències que tenen entre un 90% o un 50% d’identitat de seqüència, respectivament, fins a la seqüència més llarga. Però la seqüència més llarga no és sempre la més informativa. Sovint hi ha més informació rellevant biològicament en altres membres del clúster. Les proteïnes d'un clúster es classifiquen amb la següent prioritat per tal de facilitar la selecció d'un representat rellevant biològicament pel clúster:[23]
- Qualitat de l'entrada:es prefereixen les entrades revisades manualment (de la secció d'UniProtKB/Swiss-Prot).
- Puntuació d'anotació:es prefereixen les entrades que tinguin puntuacions d'anotació UniProtKB més altes. Això també significa que les entrades d'UniProtKB sempre tindran prioritat sobre les entrades que es troben a UniParc, però no a UniProtKB (la puntuació d'anotació no està definida a UniParc, que no conté cap anotació).
- Organisme:es prefereixen entrades de proteomes de referència i organismes model.
- Llargada de la seqüència:es prefereix la seqüència més llarga.[23]
Eines
[modifica]Uniprot conté tres eines principals:BLAST(Basic Alignment Search Tools),AligniRetrive/ID Mapping.[24]
BLAST
[modifica]Permet comprovar la similitud entre dues seqüències de proteïnes.[24]
Com executar Blast
[modifica]- Seleccionar la proteïna d'interès aUniProtKB,UniRefoUniParc.
- En els resultats de la proteïna trobarem una taula i s'ha de seleccionar en una casella a mà esquerra.
- Un cop seleccionada es fa clic al botó BLAST que es troba sobre la taula de resultats a l'esquerra. Es pot seleccionar el mode predeterminat o l'avançat.[24]

Align
[modifica]Aquesta eina permet dur a terme alineaments múltiples mitjançant l’algoritmeClustal Omegaper trobar àrees de semblança entre seqüències.[24]
Com executar Align
[modifica]Hi hadiverses formes d’utilitzaraquesta eina. Una d’elles és la següent:
- En aquest cas s’ha d’entrar directament a l'enlaç d’Aligndins el web d'UniProt.
- En segon lloc hem d’entrar les seqüències d’interès que volem comparar en format FASTA o cercant la proteïna pel seu identificador d'UniProt.
- ClicarRun align.[24]
Retrive/ID Mapping
[modifica]Aquesta eina permet penjar una llista d'identificadors d’UniProt i cercar-los encentenars de bases externes.Gràcies a aquesta eina podem obtenir informació com l'estructura 3D, interaccions entre proteïnes, famílies i grups, química i modificacions post-transcripcionals entre d’altres.[24]
Com executar Retrieve/ID Mapping
[modifica]Una de les maneres d'utilitzar aquesta eina és la següent:
- Entrar directament a l'enllaç deRetrieve/ID Mappingdes del web d'UniProt.
- Incloure els identificadors a la casella de:Provide your identifiers.
- Seleccionar la base de dades que volem als camps:Fromito.
- Obtenir els resultats.[24]
Finançament
[modifica]UniProt està finançat per subvencions de l'Institut Nacional de Recerca del Genoma Humà,elsInstituts Nacionals de Salut(NIH), laComissió Europea,el Govern Federal Suís a través de l’Oficina Federal d’Educació i Ciència,NCI-caBIGi el Departament de Defensa dels Estats Units.[25][26]
Vegeu també
[modifica]Referències
[modifica]- ↑1,01,1The UniProt Consortium«UniProt: the universal protein knowledgebase» (en anglès).Nucleic Acids Research,45, D1, 04-01-2017, pàg. D158–D169.DOI:10.1093/nar/gkw1099.ISSN:0305-1048.PMC:PMC5210571.PMID:27899622.
- ↑2,02,1Pundir,Sangya;Magrane,Michele;Martin,Maria J.;O'Donovan,Claire;The UniProt Consortium«Searching and Navigating UniProt Databases» (en anglès).Current Protocols in Bioinformatics,50, 1, 2015-06.DOI:10.1002/0471250953.bi0127s50.ISSN:1934-3396.PMC:PMC4522465.PMID:26088053.
- ↑Dayhoff,Margaret O.Atlas of protein sequence and structure..Silver Spring, Md: National Biomedicat Resarach Foundation, 1965.
- ↑«2002 NHGRI Funds Global Protein Database - National Human Genome Research Institute (NHGRI)» (en anglès). [Consulta: 19 desembre 2020].
- ↑O'Donovan,C. «High-quality protein knowledge resource: SWISS-PROT and TrEMBL».Briefings in Bioinformatics,3, 3, 01-01-2002, pàg. 275–284.DOI:10.1093/bib/3.3.275.ISSN:1467-5463.
- ↑Wu,Cathy H.;Yeh,Lai-Su L.;Huang,Hongzhan;Arminski,Leslie;Castro-Alvear,Jorge «The Protein Information Resource».Nucleic Acids Research,31, 1, 01-01-2003, pàg. 345–347.ISSN:0305-1048.PMID:12520019.
- ↑Boeckmann,Brigitte;Bairoch,Amos;Apweiler,Rolf;Blatter,Marie-Claude;Estreicher,Anne «The SWISS-PROT protein knowledgebase and its supplement TrEMBL in 2003».Nucleic Acids Research,31, 1, 01-01-2003, pàg. 365–370.ISSN:0305-1048.PMID:12520024.
- ↑Bairoch,A;Apweiler,R «The SWISS-PROT protein sequence data bank and its new supplement TREMBL.».Nucleic Acids Research,24, 1, 01-01-1996, pàg. 21–25.ISSN:0305-1048.PMID:8594581.
- ↑Bairoch,A. «Serendipity in bioinformatics, the tribulations of a Swiss bioinformatician through exciting times!».Bioinformatics,16, 1, 01-01-2000, pàg. 48–64.DOI:10.1093/bioinformatics/16.1.48.ISSN:1367-4803.
- ↑Apweiler,Rolf;Bairoch,Amos;Wu,Cathy H «Protein sequence databases» (en anglès).Current Opinion in Chemical Biology,8, 1, 01-02-2004, pàg. 76–80.DOI:10.1016/j.cbpa.2003.12.004.ISSN:1367-5931.
- ↑11,011,1The UniProt Consortium«UniProt: the universal protein knowledgebase» (en anglès).Nucleic Acids Research,45, D1, 04-01-2017, pàg. D158–D169.DOI:10.1093/nar/gkw1099.ISSN:0305-1048.PMC:PMC5210571.PMID:27899622.
- ↑«Why is UniProtKB composed of 2 sections, UniProtKB/Swiss-Prot and UniProtKB/TrEMBL?». [Consulta: 19 desembre 2020].
- ↑13,013,113,2«Where do the UniProtKB protein sequences come from?».
- ↑14,014,114,2«How do we manually annotate a UniProtKB entry?». [Consulta: 19 desembre 2020].
- ↑«InterPro». [Consulta: 19 desembre 2020].
- ↑«UniRule». [Consulta: 19 desembre 2020].
- ↑«ARBA». [Consulta: 19 desembre 2020].
- ↑18,018,1«Does UniProtKB contain all protein sequences?». [Consulta: 19 desembre 2020].
- ↑Leinonen,Rasko;Diez,Federico Garcia;Binns,David;Fleischmann,Wolfgang;Lopez,Rodrigo «UniProt archive» (en anglès).Bioinformatics,20, 17, 22-11-2004, pàg. 3236–3237.DOI:10.1093/bioinformatics/bth191.ISSN:1367-4803.
- ↑«UniParc». [Consulta: 20 desembre].
- ↑Suzek,B. E.;Huang,H.;McGarvey,P.;Mazumder,R.;Wu,C. H. «UniRef: comprehensive and non-redundant UniProt reference clusters» (en anglès).Bioinformatics,23, 10, 15-05-2007, pàg. 1282–1288.DOI:10.1093/bioinformatics/btm098.ISSN:1367-4803.
- ↑Steinegger,Martin;Söding,Johannes «Clustering huge protein sequence sets in linear time» (en anglès).Nature Communications,9, 1, 29-06-2018, pàg. 2542.DOI:10.1038/s41467-018-04964-5.ISSN:2041-1723.
- ↑23,023,123,223,323,4«UniRef». [Consulta: 20 desembre].
- ↑24,024,124,224,324,424,524,6Pundir,Sangya;Martin,Maria J.;O'Donovan,Claire;The UniProt Consortium«UniProt Tools» (en anglès).Current Protocols in Bioinformatics,53, 1, 2016-03.DOI:10.1002/0471250953.bi0129s53.ISSN:1934-3396.PMC:PMC4941944.PMID:27010333.
- ↑«The Universal Protein Resource (UniProt) in 2010».Nucleic Acids Research,38, suppl_1, 20-10-2009, pàg. D142–D148.DOI:10.1093/nar/gkp846.ISSN:0305-1048.PMC:PMC2808944.PMID:19843607.
- ↑The UniProt Consortium«UniProt: a worldwide hub of protein knowledge» (en anglès).Nucleic Acids Research,47, D1, 08-01-2019, pàg. D506–D515.DOI:10.1093/nar/gky1049.ISSN:0305-1048.PMC:PMC6323992.PMID:30395287.
