Nudiviridae
| Nudiviridae | ||||||||
|---|---|---|---|---|---|---|---|---|

Nudiviridae | ||||||||
| Systematik | ||||||||
| ||||||||
| Taxonomische Merkmale | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Nudiviridae | ||||||||
| Links | ||||||||
|
DieNudiviridaesind eine Familie von Viren (Nudiviren). Als natürliche Wirte dienenPancrustacea(Insektenund marineKrebstiere). Mit Stand Juni 2021 gibt es in dieser Familie vier vomInternational Committee on Taxonomy of Viruses(ICTV) bestätigte Gattungen (AlphanudivirusbisDeltanudivirus). Die Infektion mit diesen Viren bedeutet für ihre Wirte chronische Erkrankung beiAdultenund Tod beiLarven.[2][1]
Die Familie ist Mitglied der 2021 neu geschaffenen OrdnungLefaviralesin der ebenfalls neuen KlasseNaldaviricetes. Diese Taxonomie löst provisorische Bezeichnungen ab wie beispielsweise „Baculo-like viruses “.[3]
Die Vorsilbe ‚nudi‘ ist abgeleitet aus dem lateinischen Wort für ‚nackt‘ und deutet an, dass dieNudiviridaenicht wie die mit ihnen verwandtenBaculoviridaeihreVirionen(Viruspartikel) in Okklusionskörperchen gebündelt einschließen (englischnon-occluded).[2]
Aufbau
[Bearbeiten|Quelltext bearbeiten]

DieVirionen(Virusteilchen) derNudiviridaebesitzen eine stäbchenförmige Geometrie und sind behüllt. Im Gegensatz zu den ansonsten ähnlichenBaculoviridae(mit ebenfalls stäbchenförmigen Virionen) sind diese aber nicht in Okklusionskörperchen verpackt, sondern ‚nackt‘.[2][4]
DasGenomderNudiviridaebesteht aus ringförmiger (zirkulärer) Doppelstrang-DNA(dsDNA).[2]
Vermehrungszyklus
[Bearbeiten|Quelltext bearbeiten]Die Replikation der Viren geschieht imZellkern(nukleär). Die Methode derTranskriptionist DNA-gestützt. Das Virus verlässt die Wirtszelle via Zusammenbruch der Kernhülle oder Export durch die Kernporen. Die Übertragungswege sind elterlich (von Müttern auf ihre Nachkommen) und sexuell.[2]
- IntranukleäreAssemblierungund Virionen-Transport am Beispiel vonOryctes rhinoceros nudivirus(OrNV)
-
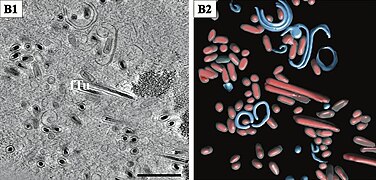
Lange gefüllte Tubuli (fTu) und von Membranen umhüllte Virionencluster. Balken 500nm
-
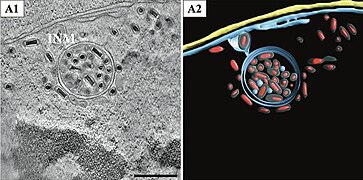
In einemVesikelmit Doppelmembran eingeschlossene Virionen. Balken 500 nm.
-
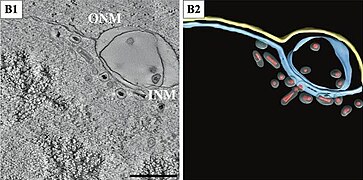
Virionen innerhalb eines erweitertenLumensder Kernhülle (NE). Balken 500 nm.
-
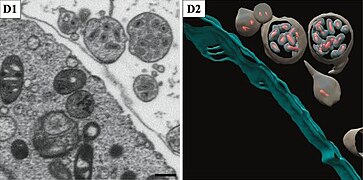
Virionen, die in Multimembranvesikel (MMVs) im Zytoplasma eingekapselt sind. Balken 500 nm.
-
Vesikel, die vollständig umhüllte Virionen enthalten. Balken 500 nm.

Wirte
[Bearbeiten|Quelltext bearbeiten]Die Wirte der GattungAlphanudivirussindTaufliegen,Käfer(Coleoptera) undGrillen(Grylloidea), die der GattungBetanudivirussindSchmetterlinge(Lepidoptera).[2]Die Wirte der GattungGammanudivirussindZehnfußkrebse,die vonDeltanudivirusKohlschnaken(s. u.).
Histone
[Bearbeiten|Quelltext bearbeiten]Unter denNudiviridaegibt es offenbar Vertreter, dieHistoneoder Histon-Homologekodieren.2022 wurde die Rolle von Histonen imReplikationszykluseiner Reihe von Viren umfassend untersucht.[5]
Systematik
[Bearbeiten|Quelltext bearbeiten]Innere Systematik
[Bearbeiten|Quelltext bearbeiten]Mit Stand Mai 2024 sind vom ICTV folgende Spezies bestätigt:[6][7]
- Familie:Nudiviridae
- Gattung:Alphanudivirus[8]
- Spezies:Alphanudivirus alterdromelanogasteris(früherDrosophila melanogaster nudivirus B) mit Tomelloso virus (TV),
– Wirte:TaufliegenwieDrosophila melanogaster - Spezies:Alphanudivirus droinnubilaemit Drosophila innubila nudivirus (DiNV),
– Wirte:TaufliegenwieDrosophila innubila[9][10] - Spezies:Alphanudivirus dromelanogasteris(früherDrosophila melanogaster nudivirus A) mit Kallithea virus (KV),
– Wirte: Taufliegen wieD. melanogaster[11][12] - Spezies:Alphanudivirus grybimaculatimit Gryllus bimaculatus nudivirus (GbNV)
– Wirte:Grillen - Spezies:Alphanudivirus oryrhinocerotis(ehem. Typusart) mit Oryctes rhinoceros nudivirus (OrNV)
– Wirte:Riesenkäfer(GattungOryctes) - Spezies:Alphanudivirus quartudromelanogasteris(früherDrosophila melanogaster nudivirus D) mit Mauternbach virus (MV),
– Wirte:TaufliegenwieD. melanogaster - Spezies:Alphanudivirus tertidromelanogasteris(früherDrosophila melanogaster nudivirus C) mit Esparto virus (EV),
– Wirte:TaufliegenwieD. melanogaster
- Spezies:Alphanudivirus alterdromelanogasteris(früherDrosophila melanogaster nudivirus B) mit Tomelloso virus (TV),
- Gattung:Alphanudivirus[8]
- Gattung:Betanudivirus[13]
- Spezies:Betanudivirus hezeae(ehem. Typusart) mit Heliothis zea nudivirus 1 (HzNV1) und Helicoverpa zea nudivirus 2 (HzNV2),[14]
– Wirte: Schmetterlinge (Baumwollkapselbohrer)
- Spezies:Betanudivirus hezeae(ehem. Typusart) mit Heliothis zea nudivirus 1 (HzNV1) und Helicoverpa zea nudivirus 2 (HzNV2),[14]
- Gattung:Betanudivirus[13]
- Gattung:Gammanudivirus
- Spezies:Gammanudivirus cameanadismit Carcinus maenas nudivirus (CmNV)
– Wirte:Gemeine Strandkrabbe - Spezies:Gammanudivirus cracrangonismit Crangon crangon nudivirus (CcNV)
– Wirte:Nordseegarnele - Spezies:Gammanudivirus hogammarimit Homarus gammarus nudivirus (HgNV)
– Wirte:Hummer - Spezies:Gammanudivirus pemonodonismit Penaeus monodon nudivirus (PmNV)
– Wirte:Giant Tiger Prawn(Penaeus monodon)[14]
- Spezies:Gammanudivirus cameanadismit Carcinus maenas nudivirus (CmNV)
- Gattung:Gammanudivirus
- Gattung:Deltanudivirus
- Spezies:Deltanudivirus tipoleraceaemit Tipula oleracea nudivirus (ToNV)
– Wirte:Kohlschnaken[11]
- Spezies:Deltanudivirus tipoleraceaemit Tipula oleracea nudivirus (ToNV)
- Gattung:Deltanudivirus
Offenbar gibt es auch Insekten mit einemGenom,das integrierte (‚endogene‘) Nudiviren enthält: So etwa die brauneSpornzikadeNilaparvata lugens(Nlu,englischbrown planthopper,BPH) mit demNilaparvata lugens endogenous nudivirus(NlENV).[15][16]Nach Bézieret al.ist NlENV in oder bei der GattungAlphanudiviruszu verorten.[11]
Äußere Systematik
[Bearbeiten|Quelltext bearbeiten]Die Nudiviridae bilden offenbar mit denNimaviridae,Hytrosaviridae,Baculoviridae(allesamt aus der 2021 neu geschaffenen KlasseNaldaviricetes) und der GattungBracovirus(der vermutet polyphyletischen)Polydnaviridae[17]eine noch unbenannte Verwandtschaftsgruppe. Ein Konsenskladogramm der Autoren Kooninet al.(2015 und 2019),[18][19] Bézieret al.(2014),[11]Yanget al.(2014),[14]sowie Kawatoet al.(2018)[20][14]könnte etwa wie folgt aussehen:
| Naldaviricetes |
| ||||||||||||||||||||||||||||||||||||||||||
Im 1. Halbjahr 2021 hat dea ICTV diese Gruppe als KlasseNaldaviricetes– (noch) ohneBracovirus– offiziell anerkannt.[1] Nach Bézieret al.(2014) geht die GattungBracovirusdirekt aus denNudiviridaehervor und ist eine Schwesterkladeder (inzwischen vom ICTV benannten) GattungDeltanudivirus(mit ToNV).[11]
Einzelnachweise
[Bearbeiten|Quelltext bearbeiten]- ↑abcdICTV:ICTV Master Species List 2020.v1,New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- ↑abcdefViral Zone: Nudiviridae.ExPASy,abgerufen am 30. Juli 2019.
- ↑SIB:Double Strand DNA Viruses,auf: ViralZone.
- ↑Viral Zone: Baculoviridae.ExPASy,abgerufen am 31. Juli 2019.
- ↑ Paul B. Talbert, Karim-Jean Armache, Steven Henikoff:Viral histones: pickpocket’s prize or primordial progenitor?In: BMC Epigenetics & Chromatin, Band 15, Nr. 21, 28. Mai 2022;doi:10.1186/s13072-022-00454-7,PMID 35624484,PMC 9145170(freier Volltext).
- ↑ICTV:Taxonomy Browser.
- ↑ICTV:Virus Metadata Resource (VMR).
- ↑SIB:Alphanudivirus,auf: ViralZone
- ↑Tom Hill, Robert L. Unckless:The dynamic evolution of Drosophila innubila Nudivirus.In:Infect Genet Evol.Band 57, Januar 2018; S. 151–157;doi:10.1016/j.meegid.2017.11.013,Epub 16. November 2017 (englisch).
- ↑Tom Hill, Boryana S. Koseva, Robert L. Unckless:The Genome ofDrosophila innubilaReveals Lineage-Specific Patterns of Selection in Immune Genes.In:Molecular Biology and Evolution,Band 36, Nr. 7, Juli 2019, S. 1405–1417;doi:10.1093/molbev/msz059,Epub 13. März 2019 (englisch).
- ↑abcdeAnnie Bézier, Julien Thézé, Frederick Gavory, Julien Gaillard, Julie Poulain, Jean-Michel Drezen, Elisabeth A. Herniou; G. McFadden (Hrsg.):The Genome of the Nucleopolyhedrosis-Causing Virus fromTipula oleraceaSheds New Light on theNudiviridaeFamily,in: Journal of Virology 2014,doi:10.1128/JVI.02884-14,PMID 25540386
- ↑K. A. Dyer, J. Jaenike:Evolutionarily stable infection by a male-killing endosymbiont in Drosophila innubila: molecular evidence from the host and parasite genomes.In:Genetics.168. Jahrgang,Nr.3,November 2004,S.1443–1455,doi:10.1534/genetics.104.027854,PMID 15579697,PMC 1448788(freier Volltext) – (englisch).
- ↑SIB:Betanudivirus,auf: ViralZone
- ↑abcdYi-Ting Yang, Der-Yen Lee, Yongjie Wang, Jer-Ming Hu, WH. Li, JH. Leu, GD. Chang, HM. Ke, ST. Kang, SS. Lin, HH. Kou, CF. Lo:The genome and occlusion bodies of marine Penaeus monodon nudivirus (PmNV, also known as MBV and PemoNPV) suggest that it should be assigned to a new nudivirus genus that is distinct from the terrestrial nudiviruses,in: BMC genomics, 15(1):628, Juli 2014,doi:10.1186/1471-2164-15-628,PMID 25063321
- ↑Eddy Dijkstra, Jose M. Rubio, Rory J. Post:Resolving relationships over a wide taxonomic range in Delphacidae (Homoptera) using the COI gene,in: Phylogenomics, 9. Februar 2003,doi:10.1046/j.1365-3113.2003.00203.x
- ↑Ruo-Lin Cheng, Yu Xi, Yi-Han Lou, Zhuo Wang, Ji-Yu Xu, Hai-Jun Xu, Chuan-Xi Zhang; A. Simon (Hrsg.):Brown Planthopper Nudivirus DNA Integrated in Its Host Genome,in: Journal of Virology 2014,doi:10.1128/JVI.03166-13
- ↑Dupuy C, Huguet E, Drezen JM:Unfolding the evolutionary story of polydnaviruses.In:Virus Res.117. Jahrgang,Nr.1,2006,S.81–89,doi:10.1016/j.virusres.2006.01.001,PMID 16460826.
- ↑Eugene V. Koonin, Natalya Yutin:Evolution of the Large Nucleocytoplasmatic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism,in: Advances in Virus Research, Band 103, AP 21. Januar 2019,doi:10.1016/bs.aivir.2018.09.002,S. 167–202
- ↑Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic:Origins and evolution of viruses of eukaryotes: The ultimate modularity,in:VirologyMai 2015; 479–480. 2–25.PMC 5898234(freier Volltext),PMID 25771806.
- ↑Satoshi Kawato, Aiko Shitara, Yuanyuan Wang, Reiko Nozaki, Hidehiro Kondo, Ikuo Hirono; Joanna L. Shisler (Hrsg.):Crustacean Genome Exploration Reveals the Evolutionary Origin of White Spot Syndrome Virus,in: Journal of Virology 2018,doi:10.1128/JVI.01144-18,PMID 30404800