Ribonukleinsäure

Ribonukleinsäure(Ribo|nukle-in|säure, kurzRNS;englischRNAfürribonucleic acid;lateinisch-französisch-griechischesKunstwort) ist eineNukleinsäure,die sich alsPolynukleotidaus einer Kette von vielenNukleotidenzusammensetzt. DasBiomolekülist bei bestimmten Virentypen (RNA-Viren,Retroviren) sowie den hypothetischen urzeitlichenRibozytenTräger derErbinformation,also die materielle Basis derGene.Das Wort setzt sich zusammen ausRiboseundNukleinsäure.
Eine wesentliche Funktion der RNA in derbiologischen Zelleist die Umsetzung von genetischer Information inProteine(sieheProteinbiosynthese,TranskriptionundTranslation), in Form dermRNAfungiert sie hierbei als Informationsüberträger. Daneben erfüllen spezielle RNA-Typen weitere Aufgaben; bei RNA-Virenmacht sie sogar dasGenomselbst aus. Weiterhin bestehen auch Teile der für die Umsetzung dieser Information verantwortlichen Zellbestandteile aus RNA: Bei der Reifung der mRNA sindsnRNAundsnoRNAbeteiligt, die katalytischen Bestandteile derRibosomenbildet dierRNA,und dietRNAtransportiert die Bausteine für die Proteine. Ferner sind spezielle RNAs an derGenregulationbeteiligt.
RNA kann auch Aufgaben von Enzymen übernehmen (Ribozym) oder ähnlich Antikörpern wirken (Aptamer).
Aufbau und Unterschied zur DNA
[Bearbeiten|Quelltext bearbeiten]
Vom Aufbau her ist die RNA derDNAähnlich. RNA-Moleküle sind – im Gegensatz zur doppelsträngigen DNA – in der Regel einzelsträngig, können allerdings in kurzen Strecken mit komplementären Basensequenzen (A–U, G–C) charakteristische Rückfaltungen ausbilden, die intramolekular den Eindruck einer Doppelstrang-Helix erwecken. Beide sindPolynukleotide,bei denen dieNukleobasenan Zuckern überPhosphorsäurediestermiteinander verknüpft sind. Die Einzelsträngigkeit erhöht die Zahl der Möglichkeiten für dreidimensionale Strukturen der RNA und erlaubt ihr chemische Reaktionen, die der DNA nicht möglich sind. Jedes Nukleotid besteht bei der RNA aus einerRibose(d. h. einerPentose:einemZuckermit fünf C-Atomen), einemPhosphatrestund einerorganischen Base.Die Ribose der RNA ist mit derjenigen der DNA identisch, bis auf eineHydroxygruppestatt einesWasserstoff-Atoms an der2'-Positionim Pentose-Ring (daher auchDesoxyribonukleinsäure,DNA). Dieser Unterschied macht die RNA weniger stabil im Vergleich zur DNA, da er eineHydrolysedurch Basen ermöglicht: Die OH-Gruppe an der 2'-Position des Zuckers wird durch ein negativ geladenesHydroxidioneiner Base ihresProtonsberaubt und der dann zurückgebliebene Sauerstoff geht eineRingbindungmit dem Phosphor ein, wodurch die Bindung zum nächsten Nukleotid gelöst wird. Die RNA wird so in ihre Nukleotide zerlegt.
In der RNA kommen die folgenden organischenBasenvor:Adenin,Guanin,CytosinundUracil.Die ersten drei Basen kommen auch in der DNA vor. Uracil dagegen ersetztThyminals komplementäre Base zu Adenin. Vermutlich nutzt RNA Uracil, da dieses energetisch weniger aufwändig herzustellen ist (keine Methyl-Substituierung).
AlsSekundärstrukturensind bei der RNA vor allemHairpin-, Stemloop-und Loop-Strukturen bekannt, eine Helix-Konformation ist aber ebenfalls möglich, wobei Hairpin- und Stemloop-Strukturen sowohl Einzelstrang- als auch Doppelstrangbereiche aufweisen. Die Loop-Strukturen bezeichnen einzelsträngige Schlaufenstrukturen innerhalb eines Moleküls.
RNA kann wie DNA ebenfalls als doppelsträngiges Molekül vorliegen. Sie weist dabei die typischen Merkmale einer Watson-Crick-Helix auf: antiparallele Anordnung der RNA-Stränge und rechtsgewundene Helix. Sie nimmt dabei die Form einer A- oder A´-Helix an (sieheDNA). Die A-RNA wird auch als RNA-11 bezeichnet, homolog zur A´-RNA, die als RNA-12 bezeichnet wird. Hierbei gibt die Zahl nach dem Spiegelstrich die Anzahl der Basenpaare je Helixwindung wieder. A´-RNA kommt häufig bei hohen Salzkonzentrationen vor (über 20 %).
A-RNA: 11 Basenpaare pro Helixwindung, Ganghöhe 2,7 nm bis 2,8 nm, Neigungswinkel zur Helixachse ca. 14°
A´-RNA: 12 Basenpaare pro Helixwindung, Ganghöhe 3 nm, Neigungswinkel zur Helixachse 16° bis 19°
Das in Lebewesen vorkommendeEnantiomerder RNA ist dieD-RNA. Sie ist ausD-Ribonukleotiden aufgebaut. DieChiralitätszentrenliegen in derD-Ribose. Durch Verwendung vonL-Ribose, bzw.L-Ribonukleotiden lässt sichL-RNA synthetisieren. Diese ist vergleichsweise stabiler gegenüber dem enzymatischen Abbau durchRNasen.[1]
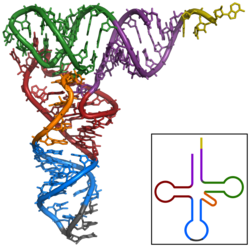
Tertiärstruktur
[Bearbeiten|Quelltext bearbeiten]Nukleinsäuren können ebenfalls komplexe räumliche Strukturen einnehmen:tRNAsmüssen für ihre Funktion in der korrekten Tertiärstruktur vorliegen.
-
Tertiär- und Sekundärstruktur (im Bild unten rechts) einer tRNA
-
Tertiärstruktur einesPseudoknotens
Synthese von RNA
[Bearbeiten|Quelltext bearbeiten]Das EnzymRNA-Polymerasekatalysiert an der DNA durch den Prozess derTranskriptionausNukleosidtriphosphat(NTP) die RNA. Dafür setzt sich die RNA-Polymerase an einePromotorgenannte Nukleotid-Sequenz der DNA (Transkriptionsinitiation). Dann trennt sie die DNA-Doppelhelix durch Lösen der Wasserstoffbrücken in einem kurzen Bereich in zwei DNA-Einzelstränge auf. Amcodogenen Strangder DNA lagern sich durchBasenpaarungkomplementäre Ribonukleotide an. Sie werden unter Eliminierung einesPyrophosphatdurch eine esterartige Bindung zwischenPhosphorsäureundRibosemiteinander verknüpft. Die Ableserichtung der DNA verläuft vom 3'-Ende zum 5'-Ende, die Synthese der komplementären RNA dementsprechend 5'→3'. Die Öffnung der DNA-Doppelhelix erfolgt nur in einem kurzen Bereich, so dass der bereits synthetisierte Teil der RNA aus dieser Öffnung heraushängt und zwar mit dem 5'-Ende der RNA voran. Die Synthese der RNA wird an einemTerminatorgenannten DNA-Abschnitt beendet. Danach wird das RNA-Transkript entlassen und die RNA-Polymerase löst sich von der DNA.
RNA kann perPhosphoramidit-Synthesekünstlich erzeugt werden.
Biologische Bedeutung
[Bearbeiten|Quelltext bearbeiten]RNA-Moleküle können unterschiedliche Funktionen ausüben. Die RNA kann genetische Information übertragen. Andere RNA-Moleküle tragen zur Übersetzung dieser Information in Proteine bei sowie bei derRegulation der Gene.Darüber hinaus kann RNA auch katalytische Funktionen ähnlich einemEnzyminnehaben. RNA wird daher – je nach ihrer Funktion – auch verschieden benannt. Vorangestellte Kleinbuchstaben kennzeichnen die unterschiedlichen RNA-Typen:[2]
- DiemRNA,Boten-RNA(engl.messenger RNA) kopiert die in einem Gen auf der DNA liegende Information und trägt sie zumRibosom,wo mit Hilfe dieser Information dieProteinbiosynthesestattfinden kann. Jeweils drei imLeserasterdes Polynukleotidstrang nebeneinander liegende Nukleotide bilden einCodon,mit dessen Hilfe sich eine spezifischeAminosäure,die in einProteineingebaut werden soll, eindeutig bestimmen lässt. Dieser Zusammenhang wurde 1961 vonHeinrich MatthaeiundMarshall Warren Nirenberggefunden. Die Entschlüsselung desgenetischen Codesmarkiert einen Neubeginn in fast allen Bio-Wissenschaften.
- Nukleosid-modifizierte mRNAist eine synthetische, chemisch modifizierteBoten-Ribonukleinsäure(mRNA), in der einzelneNukleosidedurch andere natürliche modifizierte Nukleoside oder durch synthetische Nukleosid-Analoga ersetzt sind. Sie wird experimentell oder therapeutisch eingesetzt.
Die folgenden RNA-Klassen werden allgemein alsnichtcodierende Ribonukleinsäurenbezeichnet.
- DieasRNA,antisense-RNA,dient der Regulation derGenexpression.
- DiecircRNA,zirkuläre RNAist durch Bindung an miRNA an der Regulation beteiligt.[3]
- DiehnRNA,heterogene Kern-RNA(engl. heterogeneous nuclear RNA), kommt im Zellkern vonEukaryotenvor und ist eine Vorstufe der reifen mRNA, häufig wird sie daher auch als prä-mRNA (oder engl. pre-mRNA für precursor mRNA) bezeichnet.
- DiemiRNAs,microRNAssind eng verwandt mit den siRNAs und dient der Regulation zellulärer Prozesse wie z. B.Proliferationund Zelltod.
- DieRiboswitchesdienen derGenregulation.Sie können entweder aktivierend oder reprimierend wirken.
- DieRibozymesindkatalytischaktive RNA-Moleküle. Sie katalysieren wieEnzymechemische Reaktionen.
- DierRNA,ribosomale RNA,trägt, ähnlich wie die tRNA, keine genetische Information, sondern ist am Aufbau desRibosomsbeteiligt und ist bei der Knüpfung derPeptidbindungauch katalytisch aktiv.
- DiesaRNA,selbstampflifizierende RNA,wird beiRNA-Impfstoffenverwendet, um die Wirkdauer zu verlängern.
- DiesiRNA,small interfering RNA,entsteht bei einem Signalweg der Zelle, der alsRNAi(RNA Interference) zusammengefasst wird. Dabei wird dsRNA (doppelsträngige RNA; englisch double-stranded RNA) durch das EnzymDicerin viele kleinere Fragmente von ca. 22 Nukleotiden Länge zerteilt (diesiRNAs) und in den EnzymkomplexRISC(RNA-induced silencing complex) eingebaut. Mithilfe der inkorporierten RNA-Fragmente bindet RISC komplementär an DNA, z. B. Genbereiche, oder mRNA und kann diese damit „abschalten “. siRNA's werden aktuell (2006) intensiv auf ihre Beteiligung an verschiedenen Zellvorgängen und Krankheiten erforscht.
- DieshRNAwird zur RNAi verwendet.
- DiesnoRNA,small nucleolar-RNA,finden sich imNukleolus,und die eng verwandtenscaRNAsin denCajal Bodies.
- DiesnRNA,small nuclear-RNA,im Zellkern vonEukaryoten,ist verantwortlich für dasSpleißender hnRNA amSpleißosom.
- DielncRNA,long non-coding RNA,sind länger als 200 Nukleotide und unterscheiden sich dadurch von kleinen regulatorischen RNAs, wie den miRNAs und den siRNAs.[4]
- DiepiRNA,Piwi-interacting RNA,sind 26–31 Nukleotide lang und unterscheiden sich dadurch von den etwas kleineren miRNAs und siRNAs. Sie bilden Komplexe mit PIWI-Proteinen die amepigenetischenund posttranskriptionellenSilencingin Keimzellen beteiligt sind.[5]
- DietRNA,Transfer-RNA,codiert keine genetische Information, sondern dient als Hilfsmolekül bei derProteinbiosynthese,indem sie eine einzelne Aminosäure aus demCytoplasmaaufnimmt und zum Ribosom transportiert. Die tRNA wird durch ein bestimmtesRNA-Gencodiert.
- DietracrRNA,die beimCRISPR/Cas9System eine wichtige Rolle spielt.
In der Mehrzahl der Lebewesen spielt die RNA als Informationsträger eine der DNA untergeordnete Rolle: Die DNA ist hier das permanente Speichermedium für die genetische Information, die RNA dient als Zwischenspeicher. NurRNA-Viren(die Mehrzahl aller Viren) nutzen RNA anstelle der DNA als permanentes Speichermedium. ZurTaxonomie von Virenunterscheidet man folgende RNA-Typen:
*dsRNA:Doppelstrang-RNA;
*ss(+)RNA:Einzelstrang-RNA, die als mRNA verwendet wird;
*ss(−)RNA:Einzelstrang-RNA, die als Matrize zur mRNA-Produktion dient.
Darüber hinaus nutzen einige Viren RNA als Replikationsintermediat (z. B.Hepadnaviren).
Abbau von RNA
[Bearbeiten|Quelltext bearbeiten]Da ständig neue RNA gebildet wird und da zu unterschiedlichen Zeitpunkten verschiedene Transkripte benötigt werden (differentielle Genexpression), darf die RNA in der Zelle nicht zu stabil sein, sondern muss auch einem Abbau unterliegen. Dies geschieht mit Hilfe vonRNasen,Enzymen, die die Verbindungen des Zucker-Gerüstes der RNA trennen und somit die Monomere (bzw. Oligomere) bilden, welche wieder zur Bildung neuer RNA verwendet werden können. Wann eine RNA abgebaut werden soll, wird dabei vor allem (aber nicht ausschließlich) durch die Länge desPoly-A-Schwanzesbestimmt, der mit zunehmender Verweildauer der RNA im Cytoplasma sukzessive verkürzt wird. Sinkt die Länge dieses Schwanzes unter einen kritischen Wert wird die RNA schnell degradiert. Zusätzlich können die RNAs stabilisierende oder destabilisierende Elemente enthalten, die eine weitere Regulation ermöglichen.
Zumindest bei der mRNA vonEukaryotenfindet der RNA-Abbau nicht irgendwo imCytoplasmastatt, sondern in den so genannten „P-Bodies “(processing bodies), die sehr reich an RNasen und anderen, am RNA-turnover (-Abbau)-beteiligten Enzymen sind. Zusammen mitStress Granulesdienen diese Körper weiterhin der kurzzeitigen Lagerung von mRNA und demonstrieren so wiederum die enge Verknüpfung des RNA-Metabolismus (hierTranslationund RNA-Abbau).
Die RNA-Welt-Hypothese
[Bearbeiten|Quelltext bearbeiten]Die RNA-Welt-Hypothese besagt, dass RNA-Moleküle bei derchemischen Evolutionvermutlich Vorläufer der Organismen waren. Die Hypothese lässt sich ableiten aus der Fähigkeit der RNA zur Speicherung, Übertragung und Vervielfältigung genetischer Informationen sowie aus ihrer Fähigkeit, alsRibozymeReaktionen zu katalysieren. In einer Evolutionsumgebung würden diejenigen RNA-Moleküle gehäuft vorkommen, die sich selbst bevorzugt vermehren.
Nobelpreise
[Bearbeiten|Quelltext bearbeiten]Für die Forschung an RNA sind bereits mehrereNobelpreiseverliehen worden:
- 1959 erhieltenS. OchoaundA. Kornbergden Nobelpreis für Medizin für ihre Studien zur Synthese der RNA durch RNA-Polymerasen.
- Für die Entdeckung der katalytischen Aktivität von RNA-Molekülen (vgl.Ribozym) wurdenS. AltmanundT. Cech1989 mit dem Nobelpreis für Chemie geehrt.
- 1993 erhieltenR. RobertsundP. Sharpden Nobelpreis für Medizin für ihre Studien zur Prozessierung der RNA inEukaryoten(siehe auchSpleißen (Biologie)).
- Im Jahr 2006 erhieltenAndrew FireundCraig Melloden Nobelpreis für Medizin für die Entdeckung derRNA-Interferenz;im selben Jahr wurdeRoger Kornberg(Sohn des Nobelpreisträgers A. Kornberg) für seine Studien zur RNA-Polymerase mit dem Nobelpreis für Chemie geehrt.
- 2009 wurdeAda Yonathzusammen mitVenkatraman RamakrishnanundThomas A. SteitzderNobelpreis für Chemie„für die Studien zur Struktur und Funktion des Ribosoms “zugesprochen.
RNA-Reinigung und Nachweis
[Bearbeiten|Quelltext bearbeiten]RNA kann durch eineRNA-Reinigung,z. B. perRNA-Extraktion,von anderenBiomolekülengetrennt werden. Bestimmung der Menge und Reinheit der isolierten RNA erfolgt durchphotometrischeMessung bei einerWellenlängevon 260 und 280 nm. Weitere Hinweise auf die Qualität der RNA erhält man durchAgarose-Gelelektrophoresegefolgt mit einer Anfärbung durch Farbstoffe wieSYBR Green II,Methylenblau,Stains-Alloder durch eineSilberfärbung.Der qualitative Nachweis von RNA (ob eine bestimmte RNA vorliegt) erfolgt meistens durch eineRT-PCR,teilweise mit einer anschließendenDNA-Sequenzierung,oder durch einenNorthern Blot.Der quantitative Nachweis (wie viel von einer bestimmten RNA vorliegt) erfolgt meistens durch eineqRT-PCR,bei gereinigten Proben mit nur einer RNA-Sequenz kann dieKonzentrationauch durchPhotometriebestimmt werden. DurchMolecular Combingkann die RNA gestreckt und ausgerichtet werden. MittelsIn situ-Hybridisierunglassen sich spezifische RNAs in Zellen und Geweben ohne vorhergehende Isolierung nachweisen.
Verwendung
[Bearbeiten|Quelltext bearbeiten]RNA wird für unterschiedliche Zwecke verwendet. BeiRibozymenbesitzt die RNA eineenzymatische Aktivität,währendAptamereeine längerfristige Bindung an eine Zielstruktur eingeht. Kurze doppelsträngige RNA in Form vonsiRNAundshRNAwird zur temporären Unterdrückung derGenexpressionperRNA-Interferenzverwendet.RNA-Impfstoffegehören zu dengenetischen Impfstoffen,bei denen dasAntigeninnerhalb der Zellen des Geimpften hergestellt wird. EinigeCRISPR-Cas-Systeme können, für temporärere Edits als bei DNA, zurVeränderungvon RNA– etwa zur Behandlung von Krankheiten – verwendet werden.[6]Eine freie Plattform für das Design von RNA-Zielsequenzen wurde 2020 veröffentlicht.[7][8]Das erste RNA-basierteArzneimittelwarPatisiran,das 2018 von derEMAin Europa und von derFDAin den USA zugelassen wurde.[9]
Siehe auch
[Bearbeiten|Quelltext bearbeiten]Literatur
[Bearbeiten|Quelltext bearbeiten]- J. Marx:P-Bodies Mark the Spot for Controlling Protein Production.In:Science.Bd. 310, Nr. 5749, S. 764–765. 2005,PMID 16272094doi:10.1126/science.310.5749.764.
- Seyffert:Lehrbuch der Genetik.2. Auflage, S. 42. Spektrum Akademischer Verlag, 2003.
- Albert Gossauer:Struktur und Reaktivität der Biomoleküle – Eine Einführung in die organische Chemie.S. 525. Wiley-VCH, 2006.
- Kapranov P, St Laurent G, Raz T,et al.:The majority of total nuclear-encoded non-ribosomal RNA in a human cell is 'dark matter' un-annotated RNA.In:BMC Biol.8. Jahrgang,Nr.1,Dezember 2010,S.149,doi:10.1186/1741-7007-8-149,PMID 21176148.
Einzelnachweise
[Bearbeiten|Quelltext bearbeiten]- ↑Vater A, Klussmann S:Turning mirror-image oligonucleotides into drugs: the evolution of Spiegelmer therapeutics.In:Drug Discovery Today.20. Jahrgang,Nr.1,Januar 2015,S.147–155,doi:10.1016/j.drudis.2014.09.004,PMID 25236655.
- ↑Brosius, J. & Tiedge, H. (2004):RNomenclature.In:RNA Biol.1(2):81–83.PMID 17179746PDF
- ↑S. Memczak, M. Jens u. a.:Circular RNAs are a large class of animal RNAs with regulatory potency.In:Nature.Band 495, Nummer 7441, März 2013, S. 333–338,doi:10.1038/nature11928.PMID 23446348.
- ↑Jeffrey M. Perkel:Visiting “Noncodarnia”.In:BioTechniques.54. Jahrgang,Nr.6,2013,ISSN1940-9818,doi:10.2144/000114037.
- ↑Anita G. Seto, Robert E. Kingston, Nelson C. Lau:The Coming of Age for Piwi Proteins.In:Molecular Cell.26. Jahrgang,Nr.5,2007,ISSN1097-2765,S.603–609,doi:10.1016/j.molcel.2007.05.021.
- ↑Sara Reardon:Step Aside, CRISPR: RNA Editing Is Taking Off.In:Scientific American.Abgerufen am 25. September 2020(englisch).
- ↑New kind of CRISPR technology to target RNA, including RNA viruses like coronavirusIn:phys.org.Abgerufen am 3. April 2020 (amerikanisches Englisch).
- ↑Hans-Hermann Wessels, Alejandro Méndez-Mancilla, Xinyi Guo, Mateusz Legut, Zharko Daniloski, Neville E. Sanjana:Massively parallel Cas13 screens reveal principles for guide RNA design.In:Nature Biotechnology.16. März 2020,S.1–6,doi:10.1038/s41587-020-0456-9,PMC 7294996(freier Volltext).
- ↑P. Löffler:Review: Vaccine Myth-Buster - Cleaning Up With Prejudices and Dangerous Misinformation.In:Frontiers in immunology.Band 12, 2021, S. 663280,doi:10.3389/fimmu.2021.663280,PMID 34177902,PMC 8222972(freier Volltext).
Weblinks
[Bearbeiten|Quelltext bearbeiten]- Literatur von und über Ribonukleinsäureim Katalog derDeutschen Nationalbibliothek
- Thomas Tuschl – auf dem Weg zum Nobelpreis(vom 7. Dezember 2007 imInternet Archive)Thomas Tuschl beistern.deüber RNA-Interferenz, 19. Juli 2004
- Die Zeit:Schatz im Erbgut,Die ZeitNr. 48/2004 vom 18. November 2004