Haplogrupo M (ADNmt)
En genética humana el haplogrupo M es un haplogrupo mitocondrial cuyos descendientes están esparcidos por todos los continentes, siendo predominante en Eurasia Oriental. Tiene una antigüedad aproximada de 60 000 a 65 000 años y probablemente se originó en la India. Está definido por los marcadores genéticos 489, 10400, 14783 y 15043.
Dada su gran distribución global se le llama también macrohaplogrupo M*, y al igual que el haplogrupo N* es derivado del haplogrupo L3. Cuenta entre sus descendientes a los haplogrupos C, Z, D, E, G, Q y numerosos subgrupos de M.
Origen
[editar]El origen del haplogrupo M, al igual que N, está relacionado con la expansión del Homo sapiens fuera de África. Es probable que primero pobladoras L3 dejaran el África Nor-oriental (Etiopía) atravesando el Cercano Oriente y que al llegar a la India hace 60 000 años dieran origen al haplogrupo M. El origen indostánico se ve reforzado por la gran cantidad de haplogrupos antiguos derivados de M presentes al Sur de Asia;[1] además es en India y Bangladés donde se encuentra la mayor concentración del macrohaplogrupo M* con frecuencias que van del 60 al 80 % de la población.[2]
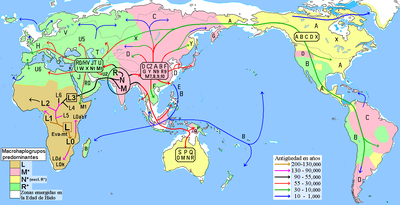
La población precursora de este grupo, habitó durante la Edad de Hielo llamada Würm/Wisconsin, última que conoció la tierra terminando el Pleistoceno, llamada también la “glaciación antropológica” que comenzó hace 100 000 años y terminó hace 10 000. M se desarrolla en regiones meridionales, que aún durante este período presentaban climas tan benignos y tropicales como los de África de donde provenían. Esta región iba en un continuo desde el valle del Indo hasta Sondalandia (Sureste asiático), y resultaron muy favorables para el crecimiento demográfico, de tal manera que probablemente no exista en el Paleolítico un caso similar de rápida diversificación genética; tal como lo demuestran los numerosos descendientes de M representados por al menos 44 clados descubiertos que ya están bien definidos.[3]
Dispersión
[editar]Desde la India se produce una expansión principalmente costera hacia el Sudeste de Asia de los descendientes de M conjuntamente con otros descendientes de N, y desde allí continúa una subsecuente expansión hacia el Asia Oriental (China) por un lado y otra hacia Sahul (Australia) por el otro; así pues estas poblaciones representan a los primeros Homo sapiens que colonizaron estas regiones y al mismo tiempo constituyen la base del origen de las actuales poblaciones nativas del Extremo Oriente y Oceanía.
En sentido opuesto, una migración trayendo el haplogrupo M1 hacia el Cercano Oriente regresaría al África, lo que se relaciona con la expansión de las lenguas afroasiáticas,[4] si bien algunos autores consideraban a M1 de origen no indio por la posible aparición de mutaciones paralelas,[5] esto quedaría desvirtuado ya que en realidad M1 es un subgrupo que descendería del clado M1'20'51, el cual tiene además una relación con las migraciones que se dirigieron hacia el Extremo Oriente. La evidencia sugiere que M1 coloniza África durante el neolítico.[6]
Muchos de los primeros pobladores americanos, los cuales provienen de Siberia, descienden del macrohaplogrupo M (subgrupos C y D) y bien pudieron ser los primeros en colonizar América; pues al extremo Sur de Sudamérica su frecuencia se acerca al 100 % en las poblaciones nativas como los mapuches, patagones y fueguinos. Incluso se encontró una variedad del haplogrupo M en restos de pobladores amerindios en Columbia Británica de hace 5000 años,[7] así como los restos más antiguos analizados genéticamente, encontrados en la isla del Príncipe de Gales (costa del Pacífico de Alaska) de hace 10 300 años. [8]
Subgrupos de M* y su distribución
[editar]El haplogrupo M (489 10400 14783 15043) y sus haplogrupos descendientes se relacionan del siguiente modo:[9]
- Haplogrupo M
- M1'20'51 (14110)
- M1 (195 6446 6680 12403 12950C 16129 16189 16249 16311): Encontrado en el Cuerno de África, especialmente en Etiopía con 15 % y Somalia 11 %, Importante también en África del Norte y Medio Oriente. Frecuencias menores en África Oriental, Sur de Europa y Cáucaso.[10]
- M1a (o M1abd): Bien extendido en la región del Mediterráneo, llegando a África Oriental y el Cáucaso, y con la mayor frecuencia en Etiopía.[4]
- M1b (o M1c): El Mediterráneo, llegando en África hasta Senegal.[10]
- M20: En China.[11]
- M51: En Camboya,[12] pueblo cham, Sumatra.
- M1 (195 6446 6680 12403 12950C 16129 16189 16249 16311): Encontrado en el Cuerno de África, especialmente en Etiopía con 15 % y Somalia 11 %, Importante también en África del Norte y Medio Oriente. Frecuencias menores en África Oriental, Sur de Europa y Cáucaso.[10]
- M2 (447G 1780 8502 11083 15670 16274 16319): Característico del subcontinente indio, con altas concentraciones al SE de India y en Bangladés. La mayor diversidad está entre tribus drávidas.[13]
- M3: Altas concentraciones al NO de India y al oeste, en especial en Karnataka (39 %).[14] También en Pakistán.
- M4"67 (12007)
- M4 (16311)[18]
- M18'38 (246)
- M18: En el subcontinente indio. En Bengala Occidental frecuencias del 27 %[14]
- M38: En la India.
- (16189)
- M30: Ampliamente distribuido en la India, tanto en caucasoides como australoides.[10] También se ha encontrado en Pakistán, en el Medio Oriente (Palestina, Yemen, Arabia), África del Norte (Egipto, Túnez) y Sur de China.
- M37: En Gujarat, India[2]
- M43: En Nepal y en etnias al Nordeste de la India como los monba y shertukpen.[16]
- M63: Tribus gond (madia gond) del centro de la India.[16]
- M64 o M50: Tribu nihali del Centro-oeste de la India.[16]
- M66 o M4c: En Nepal.
- M5: Especialmente en Orissa, India[2]
- M5a'd
- M5a: India, Nepal y Rusia.[21] Es típico del pueblo gitano (20-28 %).[22]
- M5d: Nepal.
- M5b'c
- M5b: Tribus del Este de la India.[16]
- M5c: Nepal.
- M5a'd
- M6: En Arunachal Pradesh (India), Cachemira[14] y Bangladés.
- M7 (6455 9824): Este y Sudeste de Asia, especialmente en Tailandia con 22,5 %,[23] Japón, Corea e Insulindia.
- M8 (4715 7196A 8584 15487T 16298)
- M8a: Asia Oriental, SE de Asia y Siberia, especialmente en Koriakia (ulchis y koriakis).
- CZ
- Haplogrupo C
- C1: Extendido en amerindios.
- C4: En Eurasia Oriental, típico de Siberia y menor en Asia Oriental y del Sudeste.
- Haplogrupo Z: En poblaciones de Eurasia como hazaras, fineses y samis. También en Rusia, Corea, China y Asia Central.
- Haplogrupo C
- M9 (4491 16362)
- M9a'b (153 3394)
- Haplogrupo E: Especialmente en austronesios.[26]
- E1
- E2
- M10: En Asia Oriental: China, Japón y especialmente en el Tíbet. También en el Sudeste de Asia (Vietnam, Tailandia), Asia Central y Siberia.
- M11: En China, Japón y extremo Este de la India.
- M12'G
- M12: En Tíbet, poco en Japón y Sudeste de Asia.
- Haplogrupo G: Aisladamente en Asia, particularmente en Japón y Tailandia. Las más altas frecuencias en Siberia en itelmenis y koriakis.
- G1
- G2
- G3
- M13'46'61
- M14 (234, 4216, 6962)
- (DQ904234): Arabia Saudita.[29]
- (EF495222): Australia.
- M15 (o M13): En nativos de Australia se encontró 22 %.[30]
- M17 o M45: Bien extendido en el Sudeste de Asia (Indonesia e Indochina).[31]
- M17a: Indonesia.
- M17c: Indonesia, Camboya.
- M21: En el Sudeste de Asia, especialmente entre los negritos semang de Malasia como los mendriq con 87 % y los batek 48 %[32]
- M22: En los pueblos malayo,[33] cham, vietnamita y en China.
- M23'75
- M23: Poco en Madagascar.[34]
- M75: En China, especialmente en los han.[11]
- M24'41
- M24: En Palawan (Filipinas).[20]
- M41: En drávidas de Andhra Pradesh[2] y en otras zonas al este de la India.
- M25: En Himachal. También en Kerala, Maharashtra y otros estados de la India.[15]
- M26: En Sumatra (Indonesia).[35]
- M27: En Melanesia: Islas de Papúa Nueva Guinea y Salomón.[36]
- M27a: Bougainville.
- M27b: Nueva Bretaña.
- M27c: Nueva Irlanda y Bougainville.
- M28: Característico de Melanesia, encontrándose en Vanuatu, Papúa Nueva Guinea, Nueva Caledonia, Islas Salomón y Fiyi, con la frecuencia más alta en la isla de Nueva Bretaña con 41 %.[36] Poco en Polinesia (Samoa y Tonga).
- M28a: Vanuatu, Nueva Bretaña y Malaita.[37]
- M28b: Nueva Bretaña.
- M29'Q
- M29: Nueva Bretaña (Papúa Nueva Guinea).[37]
- Haplogrupo Q: Típico de Oceanía, destacando Papúa Occidental con 65 %.[38]
- Q1'2
- Q1: En melanesios, polinesios[37] e islas menores de la Sonda (Indonesia).
- Q2: En aborígenes australianos y en Papúa Nueva Guinea.
- Q3: En papúes.
- Q1'2
- M31
- M32'56
- M32
- M32a: En los gran andamaneses, onge y jarawa de las Islas Andamán.[39]
- M32b (o M46): Madagascar.[34]
- M56: Korku, tribu munda del Centro de la India.[16]
- M32
- M33: En India.
- M34'57
- M34: En Karnataka[2] y otros estados de la India.
- M57: Tribus de la India del Oeste.[16]
- M35
- M35a: India.
- M35b: India, Nepal y Eslovaquia.[45]
- M36: Sur de la India.[16]
- M39'70
- M39: Disperso en la India, especialmente al Sur y Este.[16]
- M70: En China.
- M40'62
- M42'74
- M42: En Australia e India.[46] Encontrado en Adonara (Indonesia).[47]
- M42a: En aborígenes australianos.
- M42b: En tribus austroasiáticas y drávidas del Centro de la India.
- M74
- M42: En Australia e India.[46] Encontrado en Adonara (Indonesia).[47]
- M44 o M55: Costa Oeste de la India.[16]
- M47: Poco en Sumatra.[31]
- M48:[40] Oeste de la India.
- M49 (o M59): Extremo Oeste de la India;[16] antigua Terqa (territorio de la actual Siria.[19]
- M50: Poco al oeste de la India.[40]
- M52'58
- M52 (o M48[49]): Raramente en Arabia Saudita[29] e India.[5]
- M58: India.[16]
- M60: En Arunachal Pradesh (India).[16]
- M71: Al sur de China, en Tailandia, Vietnam, Indonesia, Filipinas y Malasia.[11]
- M76: Poco al sur de China y en el Sureste asiático.[11]
- M80'D
- M80: En nativos batak (negritos de las Filipinas).[20]
- Haplogrupo D: Al este de Eurasia, en América, Asia Central y ocasionalmente llega a Europa.
- D4: Extendido en Asia Oriental, Sudeste de Asia, Siberia, Asia Central y América originaria.
- D5: En el Extremo Oriente.
- M1'20'51 (14110)
Véase también
[editar]| Eva mitocondrial (L) | |||||||||||||||||||||||||||||||||||||||
| L0 | L1–6 | ||||||||||||||||||||||||||||||||||||||
| L1 | L2 | L3 | L4 | L5 | L6 | ||||||||||||||||||||||||||||||||||
| M | N | ||||||||||||||||||||||||||||||||||||||
| CZ | D | E | G | Q | A | I | O | R | S | W | X | Y | |||||||||||||||||||||||||||
| C | Z | B | F | R0 | R2'JT | P | U | ||||||||||||||||||||||||||||||||
| HV | JT | K | |||||||||||||||||||||||||||||||||||||
| H | V | J | T | ||||||||||||||||||||||||||||||||||||
Enlaces externos
[editar]- Árbol filogenético de M de van Oven M & Kayser M. 2009
- Mitochondrial DNA Site de Ian Logan
- Dispersión del Haplogrupo M, de National Geographic
Referencias
[editar]- ↑ Rajkumar et al. (2005), Phylogeny and antiquity of M macrohaplogroup inferred from complete mt DNA sequence of Indian specific lineages, BMC Evolutionary Biology 2005, 5:26 doi:10.1186/1471-2148-5-26
- ↑ a b c d e Thangaraj et al. (2006), In situ origin of deep rooting lineages of mitochondrial Macrohaplogroup 'M' in India, BMC Genomics 2006, 7:151
- ↑ PhyloTree.org - mtDNA subtree M Archivado el 12 de mayo de 2010 en Wayback Machine. Árbol filogenético de M de van Oven M & Kayser M. 2009
- ↑ a b Olivieri et al. (2006), The mtDNA legacy of the Levantine early Upper Palaeolithic in Africa, Science. 2006 Dec 15;314(5806):1767-70
- ↑ a b Sun et al. (2006), The Dazzling Array of Basal Branches in the mtDNA Macrohaplogroup M from India as Inferred from Complete Genomes, Molecular Biology and Evolution 2006 23(3):683-690; doi:10.1093/molbev/msj078
- ↑ Gonzalez et al. (2007), Mitochondrial lineage M1 traces an early human backflow to Africa, BMC Genomics 2007, 8:223 doi:10.1186/1471-2164-8-223
- ↑ R. Malhi et al. 2006 Mitochondrial haplogroup M discovered in prehistoric North Americans Journal of Archaeological Science xx (2006) 1-7
- ↑ Kemp, Brian et al 2007, Genetic Analysis of Early Holocene Skeletal Remains From Alaska and its Implications for the Settlement of the Americas
- ↑ a b van Oven M, Kayser M. 2009. Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation. Hum Mutat 30(2):E386-E394. http://www.phylotree.org, mtDNA tree Build 2 (14 Oct 2008)
- ↑ a b c Ana M González et al 2007. Mitochondrial lineage M1 traces an early human backflow to Africa BMC Genomics. 2007; 8: 223.
- ↑ a b c d e Kong, Qing-Peng et al 2010, Large-Scale mtDNA Screening Reveals a Surprising Matrilineal Complexity in East Asia and Its Implications to the Peopling of the Region
- ↑ Hartmann et al. 2009, Validation of microarray-based resequencing of 93 worldwide mitochondrial genomes
- ↑ Kumar S. et al 2008, The earliest settlers' antiquity and evolutionary history of Indian populations: evidence from M2 mtDNA lineage. BMC Evolutionary Biology 2008, 8:230doi:10.1186/1471-2148-8-230
- ↑ a b c d Metspalu et al. 2004-05. "Most of the extant mtDNA boundaries in South and Southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically modern humans"
- ↑ a b Maji S et al. 2009, Phylogeographic distribution of mitochondrial DNA macrohaplogroup M in India Indian Academy of Sciences. J. Genet. 88, 127–139
- ↑ a b c d e f g h i j k l m n ñ o p q Chandrasekar et al 2009, Updating Phylogeny of Mitochondrial DNA Macrohaplogroup M in India: Dispersal of Modern Human in South Asian Corridor. PLoS ONE 4(10): e7447. doi:10.1371/journal.pone.0007447
- ↑ a b c Fornarino, Simona et al 2009, Mitochondrial and Y-chromosome diversity of the Tharus (Nepal): a reservoir of genetic variation.
- ↑ Haplogroup M4 , Ian Logan 2010
- ↑ a b c Witas, Henryk W.; Jacek Tomczyk; Krystyna Jędrychowska-Dańska; Gyaneshwer Chaubey & Tomasz Płoszaj (2013) "mtDNA from the Early Bronze Age to the Roman Period Suggests a Genetic Link between the Indian Subcontinent and Mesopotamian Cradle of Civilization"; PLOS ONE, September 11, 2013. DOI: 10.1371/journal.pone.0073682.
- ↑ a b c Scholes, Clarissa et al 2011, Genetic diversity and evidence for population admixture in Batak Negritos from Palawan
- ↑ Logan I. 2009 Haplogroup M5
- ↑ Malyarchuk, B.A. et al 2005, Mitochondrial DNA Diversity in the Polish Roma
- ↑ a b Han-Jun Jin et al. 2008, The Peopling of Korea Revealed by Analyses of Mitochondrial DNA and Y-Chromosomal Markers
- ↑ a b Masashi Tanaka et al. 2004, Mitochondrial Genome Variation in Eastern Asia and the Peopling of Japan. Genome Res. 2004. 14: 1832-1850
- ↑ Derenko M. et al 2007, Phylogeographic Analysis of Mitochondrial DNA in Northern Asian Populations. AjHG 81, Issue 5, Nov 2007, 1025-1041
- ↑ Soares et al. (2008), Climate Change and Postglacial Human Dispersals in Southeast Asia, Molecular Biology and Evolution, June 2008; 25: 1213
- ↑ a b Zhao, Mian et al 2009, Mitochondrial genome evidence reveals successful Late Paleolithic settlement on the Tibetan Plateau.
- ↑ Dancause K. et al 2009, Origins of the Moken Sea Gypsies inferred from mitochondrial hypervariable region and whole genome sequences.
- ↑ a b Abu-Amero, Khaled K et al 2008, Mitochondrial DNA structure in the Arabian Peninsula and additional files BMC Evol Biol. 2008; 8: 45
- ↑ Hudjashov, G. et al 2007 Revealing the prehistoric settlement of Australia by Y chromosome and mtDNA analysis.
- ↑ a b Hill, C. et al 2007, A Mitochondrial Stratigraphy for Island Southeast Asia.
- ↑ Hill, Catherine et al. 2006, Phylogeography and Ethnogenesis of Aboriginal Southeast Asians, Mol. Biol. Evol. 23(12):2480–2491. 2006 doi:10.1093/molbev/msl124.
- ↑ Pierson, M. et al 2006, Deciphering Past Human Population Movements in Oceania: Provably Optimal Trees of 127 mtDNA Genomes.
- ↑ a b Ricaut, François et al. 2009, A new deep branch of eurasian mtDNA macrohaplogroup M reveals additional complexity regarding the settlement of Madagascar BMC Genomics 2009, 10:605 doi:10.1186/1471-2164-10-605
- ↑ Gunnarsdóttir, Ellen Dröfn et al 2001b, Larger mitochondrial DNA than Y-chromosome differences between matrilocal and patrilocal groups from Sumatra
- ↑ a b Merriwether, D. Andrew et al 2005, Ancient mitochondrial M haplogroups identified in the Southwest Pacific. PNAS September 13, 2005 102 37, 13034-13039
- ↑ a b c Friedlaender, Jonathan et al 2007, Melanesian mtDNA Complexity.
- ↑ Tommaseo-Ponzetta et al 2002. Mitochondrial DNA variability of West New Guinea populations. Am J Phys Anthropol 117:49-67.
- ↑ a b Endicott, Ph. et al 2006, Multiplexed SNP Typing of Ancient DNA Clarifies the Origin of Andaman mtDNA Haplogroups amongst South Asian Tribal Populations.
- ↑ a b c Reddy et al 2007, Austro-Asiatic Tribes of Northeast India Provide Hitherto Missing Genetic Link between South and Southeast Asia.
- ↑ Homo sapiens isolate NP19 mitochondrion, complete genome GenBank: KC990660.1.
- ↑ a b Logan, Ian. «Haplogroup M33». Consultado el 15 de febrero de 2023.
- ↑ a b Jiao-Yang Tian; Hua-Wei Wang; Yu-Chun Li; Wen Zhang; Yong-Gang Yao; Jits van Straten; Martin B. Richards; Qing-Peng Kong (11 de febrero de 2015). «A Genetic Contribution from the Far East into Ashkenazi Jews via the Ancient Silk Road». Scientific Reports 5: 8377. PMID 25669617. doi:10.1038/srep08377.
- ↑ Brook, Kevin (2022). The Maternal Genetic Lineages of Ashkenazic Jews (en inglés) (primera edición). Boston: Academic Studies Press. p. 80. ISBN 978-1-64469-984-3.
- ↑ Malyarchuk, B. et al 2008c, Mitochondrial DNA Variability in Slovaks, with Application to the Roma Origin,
- ↑ Kumar, Satish et al 2009, Reconstructing Indian-Australian phylogenetic link
- ↑ Mona, Stefano et al 2009, Genetic admixture history of eastern Indonesia as revealed by Y-chromosome and mitochondrial DNA analysis Mol Biol Evol (2009) doi: 10.1093/molbev/msp097
- ↑ Gunnarsdóttir, Ellen et al 2010, High-throughput sequencing of complete human mtDNA genomes from the Philippines
- ↑ Logan 2008, Haplogroup M48
