ARMAN
| Domaine | Archaea |
|---|---|
| Embranchement | Euryarchaeota |
| Classe | Incertae sedis |
| Ordre | Incertae sedis |
| Famille | Incertae sedis |
| Genre | Incertae sedis |
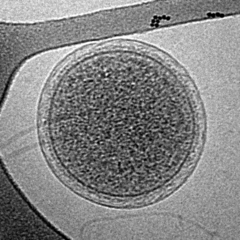
LesmicroorganismesARMAN,de l'anglaisArchaeal Richmond Mine Acidophilic Nanoorganisms,sont desarchéesnon cultivées de très petite taille — inférieure à0,5μm— observées pour la première fois dans desbiofilmsprélevés dans lesdrainages acidesissus de lamine d'Iron Mountain(en),unemineparticulièrementacide—pHinférieur à 1,5 — située près deReddingenCalifornie du Nord.L'acronyme ARMAN signifie « nanoorganismes archéens acidophiles de Richmond Mine », dans la mesure où la mine d'Iron Mountain est également appeléeRichmond Mine at Iron Mountain;il s'agit cependant d'une mine distincte de celle appelée égalementRichmond Minesituée plus au sud, près deSacramento.Ces nanoorganismes ont été caractérisés pour la première fois en 2006 par Bakeret al.[5]au laboratoire Jill Banfield de l'université de Californie à Berkeleyà l'aide de techniques deséquençage shotgunqui ont permis de distinguer trois lignées appeléesARMAN-1,ARMAN-2etARMAN-3demeurées inobservées jusqu'alors car elles présentent des séquences différentes desamorcescouramment utilisées enPCRsur lesgènesd'ARN ribosomique 16S.Des travaux ultérieurs[6]ont complété ces résultats en 2009 avec les lignéesARMAN-4etARMAN-5.
Les ARMAN sont des archées de l'embranchementdesEuryarchaeotaqui présente également des caractéristiquesgénétiquesetbiochimiquesles rapprochant desCrenarchaeotavoire, dans une moindre mesure, de certainesbactéries[6].On les trouve dans lesbiofilmschimioautotrophesqui se développent dans les eaux de drainage riches enmétauxdissous— concentration millimolaire, voire molaire, defer,dezinc,decuivreou encore d'arsenic— de certainesmines.Ces biofilms contiennent essentiellement desbactériesdugenreLeptospirillumainsi que desarchéesde l'ordredesThermoplasmatales,notamment du genreFerroplasma.Les cellules d'ARMAN qui s'y trouvent représentent de5 à 25 %du total et ont chacune un volume très réduit de 0,009 à 0,04μm3pour une moyenne de 0,03 ± 0,01μm3,les plus petites étant proches de la limite théorique des formes de vie cellulaires; ces cellules contiennent chacune environ 92ribosomes— cent fois moins d'une cellule d'E. colien culture — disposés sur la surface intérieure de lamembrane plasmique,et presque toutes contiennent unorganitetubulaire d'environ200μmde long sur60μmde large à la fonction inconnue[1].
Malgré leur petite taille, ces cellules sont souvent la cible d'au moins deux types devirus,et un petit nombre d'entre elles interagissent avec des archées de l'ordre desThermoplasmatales,qui émettent des protubérancescytoplasmiquesà travers la membrane des ARMAN[6].Il est possible que cela permette à ces nanoorganismes d'acquérir auprès de leur partenaire plus grand desmétabolitesque ces petites cellules aumétabolismedéfectif ne sont pas en mesure de produire. Les ARMAN pos sắc dent en effet quelquesgènescodant desenzymesde laglycolyse,mais pos sắc dent en revanche tous (ou presque tous, selon les lignées) les gènes nécessaires aux réactions ducycle de Krebset sont capables dedégraderlesacides grasparβ-oxydation;lasuccinate déshydrogénasea été détectée dans toutes les lignées d'ARMAN, ce qui démontre que ces nanoorganismes pos sắc dent unerespiration cellulaireaérobie.Ce mode de vie aérobie est confirmé par la présence d'unesuperoxyde dismutase(EC)et de deux gènes semblables à ceux de laperoxyrédoxine(EC),qui interviennent en réponse austress oxydant.
Bakeret al.ont proposé d'appelerles lignéesARMAN-2,ARMAN-4etARMAN-5respectivementCandidatus "Micrarchaeum acidiphilum",Candidatus "Parvarchaeum acidiphilum"etCandidatus "Parvarchaeum acidophilus"[6].
Outre la mine d'Iron Mountain, des organismes apparentés aux ARMAN ont également été identifiés dans un drainage acide près duRío TintoenEspagne[7],autour d'unetourbièreacide enFinlande[8],et même dans unesource chaudeprofonde légèrementbasiqueauJapon[9].
Notes et références
[modifier|modifier le code]- (en)Luis R Comolli, Brett J. Baker, Kenneth H. Downing, Cristina E. Siegerist et Jillian F. Banfield,«Three-dimensional analysis of the structure and ecology of a novel, ultra-small archaeon»,The ISME Journal,vol.3,,p.159-167(lire en ligne)DOI10.1038/ismej.2008.99
- (en)RéférenceNCBI:Candidatus Micrarchaeum acidiphilum(taxons inclus)
- (en)RéférenceNCBI:Candidatus Parvarchaeum acidiphilum(taxons inclus)
- (en)RéférenceNCBI:Candidatus Parvarchaeum acidophilus(taxons inclus)
- (en)Brett J. Baker, Gene W. Tyson, Richard I. Webb, Judith Flanagan, Philip Hugenholtz, Eric E. Allen et Jillian F. Banfield,«Lineages of Acidophilic Archaea Revealed by Community Genomic Analysis»,Science,vol.314,no5807,,p.1933-1935(lire en ligne)DOI10.1126/science.1132690
- (en)Brett J. Baker, Luis R. Comolli, Gregory J. Dick, Loren J. Hauser, Doug Hyatt, Brian D. Dill, Miriam L. Land, Nathan C. VerBerkmoes, Robert L. Hettich et Jillian F. Banfield,«Enigmatic, ultrasmall, uncultivated Archaea»,Proceedings of the National Academy of Sciences of the United States of America,vol.107,no19,,p.8806-8811(lire en ligne)DOI10.1073/pnas.0914470107
- (en)Linda A Amaral-Zettler, Erik R Zettler, Susanna M Theroux, Carmen Palacios, Angeles Aguilera et Ricardo Amils,«Microbial community structure across the tree of life in the extreme Río Tinto»,The ISME Journal,vol.5,,p.42-50(lire en ligne)DOI10.1038/ismej.2010.101
- (en)Heli Juottonen, Eeva-Stiina Tuittila, Sari Juutinen, Hannu Fritze et Kim Yrjälä,«Seasonality of rDNA- and rRNA-derived archaeal communities and methanogenic potential in a boreal mire»,The ISME Journal,vol.2,,p.1157-1168(lire en ligne)DOI10.1038/ismej.2008.66
- (en)Shinnosuke Murakami, Kosuke Fujishima, Masaru Tomita et Akio Kanai,«Metatranscriptomic analysis of microbes in an ocean-front deep subsurface hot spring reveals novel small RNAs and type-specific tRNA degradation»,Applied and Environmental Microbiology,,AEM.06811-11(lire en ligne)DOI10.1128/AEM.06811-11