Árbore filoxenética
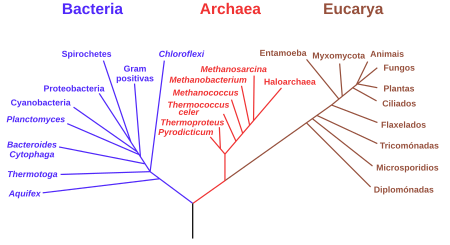
Unhaárbore filoxenética(xeralmente sinónimo deárbore evolutiva) é undiagramaramificado ou "árbore"que mostra as relaciónsevolutivasinferidas entre variasespeciesbiolóxicas ou outros grupos, é dicir a súafiloxenia,baseándose nas semellanzas e diferenzas nas súas características físicas ou xenéticas. Ostaxonsque quedan unidos na árbore considérase que descenden dunantepasado común.
Nunha árbore filoxenética enraizada, cada nodo con descendentes representa oantepasado común máis recenteinferido dos descendentes, e nalgunhas árbores a lonxitude das liñas pode interpretarse como unha estimación do tempo. Cada nodo denomínase unidade taxonómica. Os nodos internos denomínanse xeralmente unidades taxonómicas hipotéticas, xa que non poden ser observadas directamente. As árbores son útiles en campos da bioloxía como abioinformática,asistemática,e afiloxenética comparativa.
Historia
[editar|editar a fonte]
A idea dunha "árbore da vida"procede das antigas nocións dun progreso na evolución similar a subir por unha escada desde as formas" inferiores "ás" superiores "davida.As representacións iniciais de árbores filoxenéticas "ramificadas" incluían un "gráfico paleontolóxico" que mostraba as relacións xeolóxicas entre as plantas e os animais como as do libroElementary Geology(Xeoloxía Elemental) de Edward Hitchcock (primeira edición de 1840).
Charles Darwin(1859) tamén creou unha das primeiras ilustracións e popularizou a noción de"árbore" evolutivano seu influente libroThe Origin of Species(A orixe das Especies). Un século máis tarde, osbiólogos evolutivosaínda utilizan diagramas en árbore para ilustrar aevoluciónporque ditos diagramas transmiten con efectividade o concepto de que aespeciaciónocorre por medio dunha separaciónadaptativae semialeatoria das liñaxes de seres vivos. Co tempo, a clasificación das especies foi facéncose menos estática e máis dinámica.
Tipos
[editar|editar a fonte]Árbore enraizada
[editar|editar a fonte]
Unha árbore filoxenética enraizada ou con raíz é unha árboredirixidacun único nodo correspondente ao antepasado común máis recente (xeralmenteimputado) de todas as entidades situadas nas follas da árbore. O método máis común para elaborar árbores enraizadas é o uso dungrupo externo(outgroup) o suficientemente próximo e non controvertido para permitir facer a inferencia a partir da secuencia de datos de trazos, pero o suficientemente afastado para ser claramente un grupo externo.
Árbore sen raíz
[editar|editar a fonte]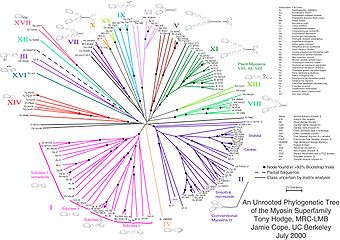
As árbores sen raíz ou non enraizadas ilustran o grao de relación entre osnodos follasen facer asuncións sobre cal foi o seu antepasado. Para construílos ou inferilos non requiren unha raíz ancestral.[2]As árbores sen raíz sempre poden xerarse a partir das árbores enraizadas simplemente omitindo a raíz. Ao contrario, inferir a raíz a partir dunha árbore sen raíz require que haxa algunha maneira para poder identificar o antepasado. Isto faise normalmente incluíndo un grupo externo nos datos utilizados para que a raíz estea necesariamente entre o grupo externo e o resto dos taxons da árbore, ou introducindo asuncións adicionais sobre as velocidades relativas de evolución en cada póla, como pode ser aplicar unha hipótese dereloxo molecular.A figura 2 mostra unha árbore filoxenética sen raíz para asmiosinas,unhasuperfamilia de proteínas.[3]
Árbore bifurcada
[editar|editar a fonte]Tanto as árbores filoxenéticas enraizadas coma as non enraizadas poden ser bifurcadas ou multifurcadas, e tamén etiquetadas ou non etiquetadas. Unha árbore bifurcada enraizada ten exactamente dous descendentes que se orixinan en cada nodo interior (é dicir, forma unhaárbore binaria), e unha árbore bifurcada non enraizada ten a forma dunha árbore binaria sen raíz, unha árborelibrecon exactamente tres veciños en cadanodo interior.A diferenza dos anteriores, unha árbore multifurcada enraizada pode ter máis de dous descendentes nalgúns nodos, e unha árbore multifurcada non enraizada pode ter máis de tres veciños nalgúns nodos. Unha árbore etiquetada ten valores específicos asignados ás súas follas, mentres que unha árbore non etiquetada, ás veces chamada forma arbórea, define soamente a topoloxía. O número de posibles árbores para un número dado de nodos folla depende do tipo específico de árbore, pero sempre hai máis árbores multifurcadas que bifurcadas, e máis árbores etiquetadas que non etiquetadas, e máis árbores enraizadas que non enraizadas. A última distinción é de grande relevencia biolóxica; prodúcese porque hai moitos lugares nunha árbore non enraizada onde se pode poñer a raíz. Para unha árbore bifurcada etiquetada habería:
árbores enraizadas totais e
árbores non enraizados totais, onderepresenta o número de nodos folla. Entre as árbores bifurcadas etiquetadas, o número de árbores non enraizadas confollas é igual ao número de árbores enraizadas confollas.[4]
Tipos de árbores especiais
[editar|editar a fonte]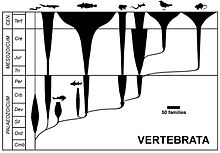
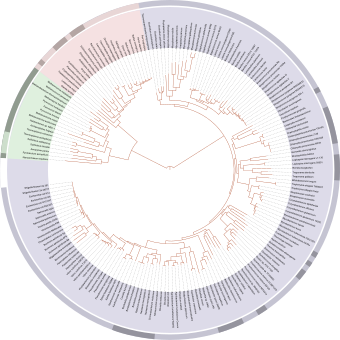
- Dendrogramaé un termo amplo para designar as representacións diagramáticas dunha árbore filoxenética.
- Uncladogramaé unha árbore filoxenética formada utilizando métodoscladísticos.Este tipo de ábore só representa un patrón de ramificación, é dicir, o tamaño das súas pólas non representa o tempo ou a cantidade relativa de cambio de caracteres.
- Un filograma é unha árbore filoxenética que ten tamaños das pólas proporcionais á cantidade de cambio nos caracteres.
- Un cronograma é unha árbore filoxenética que representa explicitamente o tempo evolutivo polo tamaño das súas pólas.
- Un diagrama de fuso (a miúdo chamado Romerograma polo paleontólogo norteamericanoAlfred Romer) é a representación da evolución e abundancia de varios taxons a través do tempo, indicada polo grosor das liñas con forma de fuso ao longo do tempo.
Construción
[editar|editar a fonte]As árbores filoxenéticas entre un número non trivial de secuencias input constrúense usando métodos defiloxenética computacional.Métodos de matriz de distancia comoneighbor-joiningouUPGMA,que calculan adistancia xenéticaa partir dealiñamentos de secuenciamúltiple, son os máis simples para aplicar, pero non implican un modelo evolutivo. Moitos métodos de aliñamento de secuencias comoClustalWtamén crean árbores usando algoritmos máis simples (como os baseados na distancia) para a construción de árbores. Amáxima parsimonia[7]é outro método simple de facer estimación de árbores filoxenéticas, pero leva implícito un modelo de evolución (é dicir parsimonia). Outros métodos máis avanzados usan ocriterio de optimalidade[8]demáxima verosimilitude,a miúdo cunharede bayesiana,e aplican un modelo explícito de evolución para a estimación dunha árbore filoxenética.[4]A identificación da árbore óptima usando moitas destas técnicas é o que na teoría de complexidade computacional se chamaNP-hard(NP-complexo),[4]así que a investigaciónheurísticae os métodos deoptimizaciónse usan en combinación con outras funcións para identificar unha árbore razoablemente boa que se corresponda cos datos.
Os métodos de construción de árbores poden ser avaliados baseándose en varios criterios:[9]
- eficiencia (canto se tarda en computar a resposta ou canta memoria se necesita)
- potencia (se fai bo uso dos datos ou desperdíciase a información)
- consistencia (se converxerá na mesma resposta repetidamente se se lle dan diferentes datos para o mesmo problema modelo)
- robustez (se se apaña ben coas violacións das asuncións do modelo subxacente)
- falsifiabilidade (se alerta cando non paga a pena usalo, é dicir cando as asuncións son violadas)
As técnicas de construción de árbores atraeron tamén a atención dos matemáticos. As árbores poden tamén construírse usando ateoría T.[10]
Limitacións
[editar|editar a fonte]
Aínda que as árbores filoxenéticas elaboradas baseándose en xenes secuenciados ou datos xenómicos de diferentes especies poden reflectir a evolución desas especies, teñen tamén importantes limitacións. Pode que non representen con exactitude a historia evolutiva dos taxons incluídos no estudo. Os datos nos que están baseados inclúen "ruído"; a análise pode confundirse polarecombinación xenética,[11]atransferencia horizontal de xenes,[12]ahibridaciónentre especies que non eran os veciños máis próximos na árbore antes de que dita hibridación tivese lugar,evolución converxente,esecuencias conservadas.
Tamén hai problemas cando a análise se basea nun só tipo de carácter, como un só xene ou proteína ou só en análises morfolóxicas, porque outras árbores construídas a partir doutros datos de fontes non relacionadas a miúdo difiren da primeira. Isto é especialmente certo con material xenético que está suxeito a transferencias laterais de xenes e recombinación, no que diferentes bloques dehaplotipospoden ter diferentes historias. En xeral, a árbore obtida nunha análise filoxenética é unha estimación da filoxenia duncarácterou trazo (é dicir unha árbore dun xene) e non a filoxenia duntaxon(unha árbore de especies), aínda que, idealmente, ambos os dous deberían ser moi parecidos. Por esta razón, para minimizar estes problemas, os estudos filoxenéticos máis completos xeralmente usan unha combinación de xenes que proceden de diferentes fontes xenómicas (por exemplo, demitocondriasouplastidiosfronte a xenomas nucleares), ou xenes que se esperaría que evolucionasen baixo diferentes réximes selectivos, para que esahomoplasia(falsahomoloxía) sexa improbable que se orixine porselección natural.
Cando se inclúen nas árbores especies extintas, estas son nodos terminais (nodos folla), xa que é improbable que sexan os antepasados directos dalgunha especie existente. Cando se utilizan datos de ADN nas árbores, hai que ter en conta que pouco "ADN antigo"útil (de especies extintas) queda preservado durante máis de 100.000 anos.
A gama de materiais de ADN útiles aumentou cos avances nas tecnoloxías de extracción e secuenciación. O desenvolvemento de tecnoloxías capaces de inferir secuencias de pequenos fragmentos, ou de patróns espaciais de produtos da degradación do ADN, poderían ampliar máis a gama de ADN considerado útil.
Nalgúns organismos, osendosimbiontesteñen unha historia xenética independente do seuhóspede.
Utilízanse asredes filoxenéticascando as árbores bifurcadas non son as axeitadas, debido a estas complicacións que suxiren unha historia evolutiva máis reticulada dos organismos estudados.
Notas
[editar|editar a fonte]- ↑Hodge T, Cope M (1 October 2000)."A myosin family tree".J Cell Sci113(19): 3353–4.PMID10984423.
- ↑http://www.ncbi.nlm.nih.gov/Class/NAWBIS/Modules/Phylogenetics/phylo9.html
- ↑Maher BA (2002)."Uprooting the Tree of Life".The Scientist16:18. Arquivado dendeo orixinalo 02 de outubro de 2003.Consultado o 09 de xullo de 2014.Arquivado02 de outubro de 2003 enWayback Machine.
- ↑4,04,14,2Felsenstein J. (2004).Inferring PhylogeniesSinauer Associates: Sunderland, MA.
- ↑Letunic, I; Bork, P (2007). "Interactive Tree Of Life (iTOL): an online tool for phylogenetic tree display and annotation.".Bioinformatics23(1): 127–8.PMID17050570.doi:10.1093/bioinformatics/btl529.
- ↑Ciccarelli, FD; Doerks, T; Von Mering, C; Creevey, CJ; Snel, B; Bork, P (2006). "Toward automatic reconstruction of a highly resolved tree of life".Science311(5765): 1283–7.Bibcode:2006Sci...311.1283C.PMID16513982.doi:10.1126/science.1123061.
- ↑A árbore filoxenética preferida é a que supón o menor cambio evolutivo para explicar os datos observados.
- ↑Optimality criterion.Mide a adecuación dos datos a unha hipótese data.
- ↑Penny, D.; Hendy, M. D.;Steel, M. A.(1992)."Progress with methods for constructing evolutionary trees".Trends in Ecology and Evolution7:73–79.doi:10.1016/0169-5347(92)90244-6.
- ↑A. Dress, K. T. Huber, and V. Moulton. 2001. Metric Spaces in Pure and Applied Mathematics.Documenta MathematicaLSU 2001:121-139
- ↑Arenas M, Posada D (2010). "The effect of recombination on the reconstruction of ancestral sequences".Genetics184(4): 1133–1139.doi:10.1534/genetics.109.113423.
- ↑Woese C (2002)."On the evolution of cells".Proc Natl Acad Sci USA99(13): 8742–7.Bibcode:2002PNAS...99.8742W.PMC124369.PMID12077305.doi:10.1073/pnas.132266999.
Véxase tamén
[editar|editar a fonte]Outros artigos
[editar|editar a fonte]- Campos de estudo
- Cladística
- Filoxenética comparada
- Filoxenética computacional
- Taxonomía evolutiva
- Bioloxía evolutiva
- Aliñamento de árbore xeneralizado
- Filoxenética
- Software
Bibliografía
[editar|editar a fonte]- Schuh, R. T. and A. V. Z. Brower. 2009.Biological Systematics: principles and applications (2nd edn.)ISBN 978-0-8014-4799-0
- MEGA,un software libre para debuxar árbore filoxenética.
Ligazóns externas
[editar|editar a fonte]| Commonsten máis contidos multimedia sobre: Phylogenetic tree |
- Imaxes
- Phylogenetic Trees Based on 16s rDNA
- Poster-sized tree of life illustrationArquivado10 de febreiro de 2015 enWayback Machine.
- Human Y-Chromosome 2002 Phylogenetic Tree
- En 2003, a revistaSciencededicou un número especial á árbore da vida, incluíndo unhaversión online dunha árbore da vida.
- iTOL: Interactive Tree Of Life
- Phylogenetic Tree of Artificial Organisms Evolved on Computers
- Miyamoto and Goodman's Phylogram of Eutherian Mammals
- Xeral
- Resumo de diferentes métodos de visualización de árbores dispoñible endoi 10.1016/j.tree.2011.12.002
- Discover LifeÁrbore interactiva baseada no Tree of Life Project
- PhyloCode
- A Multiple Alignment of 139 Myosin Sequences and a Phylogenetic Tree
- Tree of Life Web Project
- Phylogenetic inferring on the T-REX serverArquivado18 de setembro de 2020 enWayback Machine.
- NCBI's Taxonomy Database[1]
- ETE: A Python Environment for Tree ExplorationLibraría de programación para analizar, manipular e visualizar árbore filoxenéticas.Ref.
- A daily-updated tree of (sequenced) lifeArquivado09 de xuño de 2014 enWayback Machine.cite doi|10.1038/srep02015




