Ácido desoxirribonucleico


Oácido desoxirribonucleico(ADN,ouDNA,nas súas siglas eninglés) é unácido nucleicoque contén as instruciónsxenéticasusadas no desenvolvemento e funcionamento de todos osorganismosvivos coñecidos e algúnsvirus,e é responsable da súatransmisión hereditaria.O papel principal da molécula de ADN é o almacenamento a longo prazo deinformación.O ADN é comparado a miúdo cun plano ou unha receita, ou uncódigo,xa que contén as instrucións necesarias para construír outros compoñentes dascélulas,como asproteínase as moléculas deARN.Os segmentos de ADN que levan esta información xenética son chamadosxenes,pero as outrassecuencias de ADNteñen propósitos estruturais ou toman parte na regulación do uso desta información xenética.
Quimicamenteo ADN consiste en dous longospolímerosformados por unidades sinxelas chamadasnucleótidos,con columnas feitas de azucres e grupos fosfato unidos por vínculoséster(esqueleto pentosa-fosfato). Estes dous polímeros van en direccións opostas, polo que se di que sonantiparalelose trénzanse entre si formando unhadobre hélice.Unidas a cada azucre hai un dos catro tipos de moléculas chamadosbases,que son:adenina(A),guanina(G),citosina(C) etimina(T). As dúas febras do ADN soncomplementariasen bases, xa que a adenina dunha cadea sempre está enfronte dunha timina da outra cadea, e viceversa, e a citosina sempre está en fronte dunha guanina. Son as secuencias formadas por estas catro bases as que codifican a información. Esta información é lida usando ocódigo xenético,o cal especifica osaminoácidosque se deben utilizar durante atradución das proteínas.Aexpresión dos xenesrealízase copiando o anaco do ADN correspondente a ese xene noutro ácido nucleico, oARNm,nun proceso chamadotranscrición,e traducindo este ARNm a proteínas nosribosomas.
En canto á súa forma, as moléculas de ADN donúcleodas células eucarióticas son de forma linear, pero as dosprocariotas,mitocondriasecloroplastosson tipicamente circulares.
Propiedades físicas e químicas
[editar|editar a fonte]
O ADN é un longopolímeroformado por unidades repetitivas, osnucleótidos.[1][2]Unha dobre cadea de ADN mide de 22 a 26ángstroms(2,2 a 2,6nanómetros) de diámetro, e unha unidade (un nucleótido) mide 3,3 Å (0,33 nm) de longo.[3]Aínda que cada unidade individual que se repite é moi pequena, os polímeros de ADN poden sermoléculasenormes que conteñen millóns denucleótidos.Por exemplo, o cromosoma humano máis longo, que é ocromosoma 1,ten aproximadamente 220 millóns depares de bases.[4]
Nos organismos vivos, o ADN non adoita existir como unha molécula individual, senón como unha parella de moléculas longas estreitamente asociadas. As dúas cadeas de ADN enrólanse unha sobre a outra formando unha especie de escaleira de caracol, denominadadobre hélice.O modelo de estrutura en dobre hélice foi proposto en1953porJames D. WatsoneFrancis Crick(o seu artigoMolecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acidpublicouse o 25 de abril de 1953 enNature).[5]O éxito deste modelo debíase á súa consistencia coas propiedades físicas e químicas do ADN coñecidas. O estudo mostraba ademais que acomplementariedade de basespodía ser relevante na súareplicación,e tamén a importancia dasecuencia de basescomo portadora de información xenética.[6][7][8]Cada unidade que se repite, o nucleótido, contén un segmento da estrutura de soporte (azucre+fosfato), que mantén a cadea unida, e unhabase nitroxenada,que interacciona coa outra cadea de ADN na hélice. En xeral, unha base ligada a un azucre denomínasenucleósidoe unha base ligada a un azucre e a un grupo fosfato recibe o nome denucleótido.A dobre hélice do ADN é estabilizada principalmente por dúas forzas:enlaces de hidróxenoentre as bases dos nucleótidos e as interaccións entre as basesaromáticasque se sitúan unhas enriba das outras.[9]
Cando se unen moitos nucleótidos, como ocorre no ADN, o polímero que se forma denomínasepolinucleótido.[10]
Compoñentes
[editar|editar a fonte]Estrutura de soporte (esqueleto ou columna pentosa-fosfato):A estrutura de soporte dunha febra de ADN está formada por unidades alternas de gruposfosfatoeazucre.[11]O azucre no ADN é apentosadesoxirribosa.[12]

- A súafórmula químicaé H3PO4.Cadadesoxirribonucleótido(dN) contén un grupo fosfato (monofosfato: dNMP). Dos desoxirribonucleótidos derivan osdesoxirribonucleósidos(sic)[13]con dous ou tres fosfatos (dNDP e dNTP). Non obstante, como monómeros constituíntes dosácidos nucleicossó aparecen en forma de monofosfato (dNMP), aínda que chegan á zona onde se está producindo areplicación do ADNen forma de dNTP, momento en que, durante a súa incorporación ao ADN, perden dous fosfatos en forma dunpirofosfato.[14]Os fosfatos teñen un dos seus átomos de O ionizado apHcelular, o que fai que o ADN estea cuberto de cargas negativas e sexa moihidrófilo.Estas cargas negativas únense ás cargas positivas das proteínashistonas,o que facilita o empaquetamento do ADN coas histonas para formar acromatina.[15]
- A desoxirribosa ou, máis exactamente, 2-desoxirribosa, é unmonosacáridode 5átomosdecarbono(unhapentosaderivada daribosa), que forma parte da estrutura dos nucleótidos do ADN. A súa fórmula é C5H10O4.Unha das principais diferenzas entre o ADN e oARNé oazucreque levan, que no ADN é a 2-desoxirribosa(sen OH no carbono 2) e noARNé aribosa(igual, pero cun OH no carbono 2).[8]
- As moléculas de azucre únense entre si por medio dos seus gruposfosfato,que formanenlaces fosfodiésterentre os átomos de carbono terceiro (3′, «tres prima») e quinto (5′, «cinco prima») de dous aneis adxacentes de azucre.[16]Nunhadobre hélice,a dirección dosnucleótidosnunha cadea (3' → 5') é oposta ádirecciónna outra cadea (5' → 3'). Esta organización das cadeas de ADN denomínaseantiparalela,e significa que son cadeas paralelas, pero con direccións opostas. Do mesmo xeito, os extremos asimétricos das cadeas de ADN denomínanseextremo 5'eextremo 3',respectivamente.[17]
- As catro bases nitroxenadas maioritarias que se encontran no ADN son aadenina(A), acitosina(C), aguanina(G) e atimina(T).[18]Cada unha destas catro bases está unida ao armazón de azucre-fosfato a través do azucre para formar o nucleótido completo (base-azucre-fosfato). As bases soncompostos heterocíclicosearomáticoscon dous ou máisátomosdenitróxeno,e, dentro das bases maioritarias, clasifícanse en dous grupos: as bases púricas oupurinas(adenina e guanina), coa estrutura da purina e formadas por dous aneis, e as bases pirimidínicas oupirimidinas(citosina e timina), coa estrutura da pirimidina e cun só anel.[8]Nos ácidos nucleicos existe unha quinta base pirimidínica, denominadauracilo(U), que normalmente ocupa o lugar da timina noARNe difire desta en que carece dungrupo metilono seu anel. O uracilo non se encontra habitualmente no ADN, só aparece raramente como un produto residual da degradación da citosina por procesos dedesaminación,ou tamén en certos virusbacteriófagoscomo os bacteriófagos PBS1 e PBS2 deBacillus subtilis,o fago piR1-37 deYersiniae o S6 deStaphylococcusnos que a timina foi substituída por uracilo.[19][20]

-
- Represéntase pola letraT.É unha pirimidina cun grupo oxo (=O) nas posicións 2 e 4, e un grupo metil (-CH3) na posición 5.[21]Forma onucleótidodesoxitimidina monofosfato(dTMP). No ADN, a timina sempre se aparea coa adenina da cadea complementaria por medio de dúaspontes de hidróxeno,T=A.[22]

-
- Represéntase coa letraC.É unha pirimidina cun grupo amino (-NH2) en posición 4 e un grupo oxo (=O) en posición 2.[21]Forma onucleótidodesoxicitidina monofosfato(dCMP). A citosina sempre se aparea no ADN coa guanina da cadea complementaria por medio dun triplo enlace,C≡G.[22]

-
- Represéntase coa letraA.É unha purina cun grupo amino (-NH2) na posición 6.[21]Forma o nucleótidodesoxiadenosina monofosfato(dAMP). No ADN sempre se aparea coa timina da cadea complementaria por medio de dúas pontes de hidróxeno,A=T.[22]

-
- Represéntase coa letraG.É unha purina cun grupo oxo na posición 6 e un grupo amino na posición 2.[21]Forma o nucleótidodesoxiguanosina monofosfato(dGMP). A guanina sempre se aparea no ADN coa citosina da cadea complementaria por medio de tres enlaces de hidróxeno,G≡C.[22]
Tamén existen outras bases nitroxenadas, as chamadasbases nitroxenadas minoritarias,derivadas de forma natural ou sintética dalgunha outra base maioritaria. Por exemplo ahipoxantina,relativamente abundante noARNt,ou acafeína,ambas as dúas derivadas da adenina. Outras, como oaciclovir,derivadas da guanina, son análogos sintéticos usados en terapia antiviral.[23]Algunhas, como o5-fluorouracilo,derivada do uracilo, son antitumorais.[24]Unha base rara que se orixina por modificación da timina e que pode considerarse un uracilo modificado é abase J(beta-D-glicopiranosiloximetiluracilo), que se encontra nalgúns microorganismos como osflaxeladosDiplonemaeEuglena,e noscinetoplástidos.[25][26]
As bases nitroxenadas teñen unha serie de características que lles confiren unhas propiedades determinadas. Unha característica importante é o seu carácteraromático,consecuencia da presenza no anel dedobres enlacesen posición conxugada. Isto dálles a capacidade de absorber luz na zonaultravioletadoespectroen torno aos 260nm,o cal pode aproveitarse para determinar ocoeficiente de extinción[27]do ADN e achar a concentración existente dos ácidos nucleicos, e tamén para calcular ocontido G+C[28].Outra das súas características é que presentantautomeríaouisomeríadegrupos funcionais,debido a que un átomo dehidróxenounido a outro átomo pode migrar a unha posición veciña[29][30];nas bases nitroxenadas danse dous tipos de tautomerías: tautomeríalactama-lactima,onde o hidróxeno migra do nitróxeno aoosíxenodo grupo oxo (orixinando a forma lactama) e viceversa (forma lactima)[21],e tautomeríaimina-aminaprimaria, onde o hidróxeno pode estar formando o grupo amina (forma amina primaria) ou migrar ao nitróxeno adxacente (forma imina). A adenina só pode presentar tautomería amina-imina, a timina e o uracilo mostran tautomería lactama-lactima, e a guanina e citosina poden presentar ambas as dúas. Por outro lado, e aínda que se trate de moléculasapolares,as bases nitroxenadas presentan suficiente carácterpolarcomo para establecerpontes de hidróxeno,xa que teñen átomos moielectronegativos(nitróxenoeosíxeno) que presentancarga parcialnegativa, e átomos de hidróxeno con carga parcial positiva, de maneira que se forman dipolos que permiten que se formen estesenlaces febles.[31]
Calcúlase que oxenoma humanohaploideten arredor de 3.000 millóns depares de bases.[32]Para indicar o tamaño das moléculas de ADN indícase o número de pares de bases, e como derivados hai dúas unidades de medida moi utilizadas, a quilobase (kb), que equivale a 1.000 pares de bases, e a megabase (Mb), que equivale a un millón de pares de bases.[33]
Apareamento de bases
[editar|editar a fonte]

Adobre hélicede ADN mantense estable grazas á formación depontes de hidróxenoentre as bases asociadas a cada unha das dúas cadeas. Para a formación dun enlace de hidróxeno unha das bases debe presentar un "doante" de hidróxenos cun átomo de hidróxeno con carga parcial positiva (-NH2ou -NH) e a outra base debe presentar un grupo "aceptor" de hidróxenos cun átomo cargadoelectronegativamente(C=O ou N).[21]As pontes de hidróxeno son unións máis febles cós típicos enlaces químicoscovalentes,como os que conectan os átomos en cada febra de ADN, pero máis fortes cainteraccións hidrófobasindividuais,enlaces de Van der Waalsetc. Como as pontes de hidróxeno non son enlacescovalentes,poden romper e formarse de novo de forma relativamente sinxela. Por esta razón, as dúas febras da dobre hélice poden separarse como unha cremalleira, ben por forza mecánica ou por altatemperatura.[34]A dobre hélice estabilízase ademais poloefecto hidrófoboe o empillamento, aos que non lles inflúe a secuencia de bases do ADN.[35]
Cada tipo de base nunha cadea forma un enlace unicamente cun tipo de base na outra cadea, o que se denominacomplementariedade das bases.[36]Así, as purinas forman enlaces coas pirimidinas, de forma que A se enlaza só con T, e C só con G. A organización de dous nucleótidos apareados ao longo da dobre hélice denomínaseapareamento de bases.Este apareamento explica a observación xa realizada porErwin Chargaff(1905-2002),[37]que mostrou que a cantidade de adenina era similar á cantidade de timina, e que a de citosina era igual á de guanina no ADN. Como resultado desta complementariedade, toda a información contida na secuencia de dobre cadea da hélice de ADN está duplicada en cada cadea, o cal é fundamental durante o proceso dereplicación do ADN.Esta interacción reversible e específica entre pares de bases complementarias é esencial para todas as funcións do ADN nos organismos vivos.[1]
Os dous tipos de pares de bases forman un número diferente de enlaces de hidróxeno: A=T forman dúas pontes de hidróxeno, e C≡G forman tres pontes de hidróxeno (ver imaxes). O par de bases GC é polo tanto máis forte que o par de bases AT. Como consecuencia, tanto a porcentaxe de pares de bases GC como a lonxitude total da dobre hélice de ADN determinan a forza da asociación entre as dúas cadeas de ADN. As dobres hélices longas de ADN con altocontido GCteñen cadeas que interaccionan máis fortemente que as dobres hélices curtas con alto contido en AT.[38]Por esta razón, as zonas da dobre hélice de ADN que necesitan separarse doadamente tenden a ter un alto contido en AT, como por exemplo acaixa TATA(eucariotas) ou a secuencia TATAAT dacaixa de Pribnow(procariotas) dalgúnspromotoresdosxenes.[39]No laboratorio, a forza desta interacción pode medirse buscando a temperatura requirida para romper as pontes de hidróxeno, chamadatemperatura de fusión do ADN(tamén denominada valorTm,do inglésmelting temperature). Cando todos ospares de basesnunha dobre hélice se desligan, as cadeas sepáranse en solución en dúas cadeas completamente independentes. Estas moléculas de ADN de cadea simple non teñen unha única forma común, senón que algunhas conformacións son máis estables ca outras.[40]
Outros tipos de pares de bases
[editar|editar a fonte]


Existen diferentes tipos depares de basesque se poden formar por establecemento de pontes de hidróxeno. Os que se observan normalmente na dobre hélice de ADN son os chamados pares de bases de Watson e Crick, pero tamén existen outros posibles pares de bases, como os denominados de Hoogsteen[41]e cambaleantes, que poden aparecer en circunstancias particulares. Ademais, para cada tipo existe á súa vez o mesmo par inverso, é dicir, o que se dá se se xira a base pirimidínica 180º sobre o seu eixe.[42]
- Par de bases de Watson e Crick(pares de bases canónicos da dobre hélice): os grupos da base púrica que interveñen no enlace de hidróxeno son os que corresponden ás posicións 1 e 6 (N aceptor e -NH2doante se apurinaé unha A) e os grupos dabase pirimidínica,os que se encontran nas posicións 3 e 4 (-NH doante e C=O aceptor se a pirimidina é unha T). No par de bases de Watson e Crick inverso participarían os grupos das posicións 2 e 3 da base pirimidínica.[43]
- Par de bases de Hoogsteen(descubertos porKarst Hoogsteen[44]): neste caso cambian os grupos dabase púrica,que ofrece unha cara diferente (posicións 6 e 7), que forma enlaces cos grupos das pirimidinas das posicións 3 e 4 (como nos de Watson e Crick).[45]Tamén pode haber Hoogsteen inversos. Con este tipo de enlace poden unirse A=U (Hoogsteen e Hoogsteen inverso) e A=C (Hoogsteen inverso).[46]
- Par de bases cambaleante (wobble): este tipo de enlace permite que se unan guanina e citosina cun dobre enlace (G=T). A base púrica (G) forma enlace cos grupos das posicións 1 e 6 (como nos de Watson e Crick) e a pirimidina (T) cos grupos das posicións 2 e 3. Este tipo de enlace non funcionaría con A=C, xa que quedarían enfrontados os dous aceptores e os dous doantes, e só se podería dar no caso inverso. Encontramos pares de bases de tipo cambaleante no ARN, durante o apareamento decodóneanticodón.Con este tipo de enlace poden unirse G=U (cambaleante e cambaleante inverso) e A=C (cambaleante inverso).[47]
En total, na súa formatautoméricamaioritaria, existen 28 posibles pares de bases nitroxenadas: 10 posibles pares de bases purina-pirimidina (2 pares de Watson e Crick e 2 pares de Watson e Crick inverso, 1 par de Hoogsteen e 2 pares de Hoogsteen inverso, 1 par cambaleante e 2 pares cambaleantes inversos), 7 pares homo purina-purina (A=A, G=G), 4 pares A=G e 7 pares pirimidina-pirimidina. Isto sen contar cos pares de bases que poden formarse se tamén temos en conta as outras formas tautoméricas minoritarias das bases nitroxenadas; estes, ademais, poden ser responsables demutacións puntuais por substituciónde tipotransición.[48]
Estrutura
[editar|editar a fonte]O ADN é unhamoléculabicatenaria, é dicir, está formada por dúas cadeas dispostas de formaantiparalelae coas bases nitroxenadas enfrontadas. Na súa estrutura tridimensional, distínguense distintos niveis:[49][50]
- Estrutura primaria:
- É a secuencia denucleótidosencadeados. É nestas cadeas onde se encontra a información xenética, e dado que o esqueleto pentosa-fosfato é o mesmo en todos, a diferenza da información radica na distintasecuencia de basesnitroxenadas. Esta secuencia presenta uncódigo,que determina unha información ou outra, segundo a orde das bases.[22]
- Estrutura secundaria:
- É unhaestrutura en dobre hélice.Permite explicar o almacenamento da información xenética e o mecanismo de duplicación do ADN. Foi postulada por Watson e Crick, baseándose na difracción de raios X que realizaran Franklin e Wilkins, e na equivalencia de bases de Chargaff, segundo a cal a suma de adeninas máis guaninas é igual á suma de timinas máis citosinas.
- É unha cadea dobre,dextroxira(xira á dereita) oulevoxira(xira á esquerda), segundo o tipo de ADN. Ambas as cadeas son complementarias, pois a adenina e a guanina dunha cadea se unen, respectivamente, á timina e á citosina da outra. Ambas as cadeas son antiparalelas, pois o extremo 3´ dunha se enfronta ao extremo 5´ da homóloga.[22]
- Existen tres modelos de ADN. O ADN de tipo B é o máis abundante e é o que ten a estrutura descrita por Watson e Crick.
- Estrutura terciaria:
- É a forma en que se empaqueta o ADN no reducido espazo celular para formar oscromosomas.Varía segundo se trate de organismosprocariotasoueucariotas:
- Enprocariotaso ADN enrólase como unhasúper-hélice,xeralmente en forma circular e asociada a unha pequena cantidade de proteínas. O mesmo ocorre enorgánuloscelulares como asmitocondriase noscloroplastos.
- Eneucariotas,dado que a cantidade de ADN de cadacromosomaé moi grande, o empaquetamento ten que ser máis complexo e compacto; para iso cómpre a presenza de proteínas, como ashistonaseoutras proteínasde natureza non histónica (nosespermatozoidesestas proteínas son asprotaminas), que forman as fibrascromatínicasde 10nmde grosor, e a dobre hélice pode adoptar as formas A, B e Z.[49]
Tipos de estruturas en dobre hélice
[editar|editar a fonte]
O ADN existe en moitas conformacións.[11]Porén, en organismos vivos só se observaron as conformaciónsADN A,ADN BeADN Z.[51]A conformación que adopta o ADN depende da súa secuencia, a cantidade e dirección desuperenrolamentoque presenta, a presenza demodificacións químicasnas bases e as condicións da solución, tales como a concentración deiónsdemetaisepoliaminas.[52]Das tres conformacións, a forma "B" é a máis común nas condicións existentes nas células.[53]As dúas dobres hélices alternativas do ADN difiren na súa xeometría e dimensións.[51]
A forma "A" é unha espiral que xira cara á dereita, máis ampla cá "B", cun suco menor superficial e máis amplo, e un suco maior máis estreito e profundo. A forma "A" aparece en condicións non fisiolóxicas en formas deshidratadas de ADN, mentres que na célula pode orixinarse en casos de apareamentos híbridos ADN-ARN, ademais de en complexos encima-ADN.[54][55]
Os segmentos de ADN nos que as bases foron modificadas pormetilaciónpoden sufrir cambios conformacionais maiores e adoptaren a forma "Z". Neste caso, as cadeas xiran arredor do eixe da hélice nunha espiral que xira cara á esquerda, o oposto á forma "B" máis frecuente.[56]Estas estruturas pouco frecuentes poden ser recoñecidas por proteínas específicas que se unen a ADN Z e posiblemente estean implicadas na regulación datranscrición.[57]
Estruturas en cuádruplex
[editar|editar a fonte]
Nos extremos dos cromosomas lineais existen rexións especializadas de ADN denominadastelómeros.A función principal destas rexións é permitirlle á célula replicar os extremos cromosómicos utilizando o encimatelomerase,xa que os encimas que replican o resto do ADN non poden copiar os extremos 3' dos cromosomas.[59]Estas terminacións cromosómicas especializadas tamén protexen os extremos do ADN, e evitan que os sistemas dereparación do ADNda célula os procesen como ADN danado que debe ser corrixido.[60]Nas células humanas os telómeros son longas zonas de ADN dunha soa cadea que conteñen varios milleiros de repeticións dunha única secuencia TTAGGG.[61]
Estas secuencias ricas en guanina poden estabilizar os extremos cromosómicos por medio da formación de estruturas constituídas por conxuntos amoreados de unidades de catro bases, en lugar dos pares de bases encontrados normalmente noutras estruturas de ADN. Neste caso, catro bases guanina forman unidades con superficie plana que se sitúan unha sobre outra, formando unha estrutura cuádrupla-G estable.[62]Estas estruturas estabilízanse formandopontes de hidróxenoentre os extremos das bases e polaquelacióndun metal iónico no centro de cada unidade de catro bases. Tamén se poden formar outras estruturas, co conxunto central de catro bases procedente quer dunha febra sinxela (monocatenaria) pregada arredor das bases, quer de varias febras paralelas diferentes, de forma que cada unha contribúe cunha base á estrutura central.[63]
Ademais destas estruturas amoreadas, ostelómerostamén forman longas estruturas en lazo, denominadas lazos teloméricos ou lazos T (T-loopsen inglés). Neste caso, as febras simples (monocatenarias) de ADN enrólanse sobre si mesmas formando un amplo círculo estabilizado por proteínas que se unen aos telómeros.[64]No extremo do lazo T, o ADN telomérico de febra sinxela suxéitase a unha rexión de ADN de dobre cadea porque a febra de ADN telomérico altera adobre hélicee emparéllase a unha das dúas febras. Esta estrutura de tripla febra denomínaselazo de desprazamentooulazo D(D-loop).[62]
Sucos maior e menor
[editar|editar a fonte]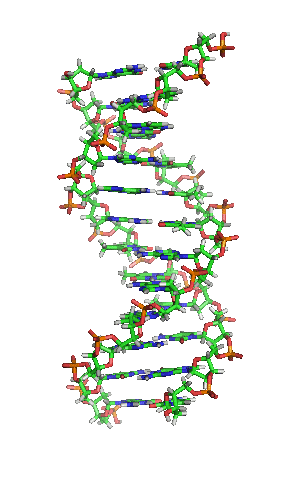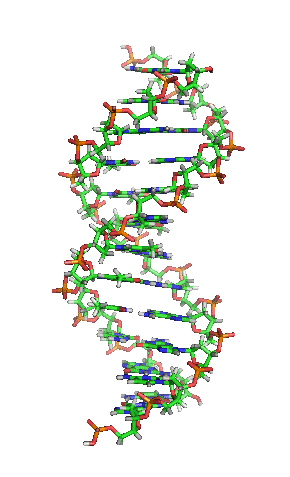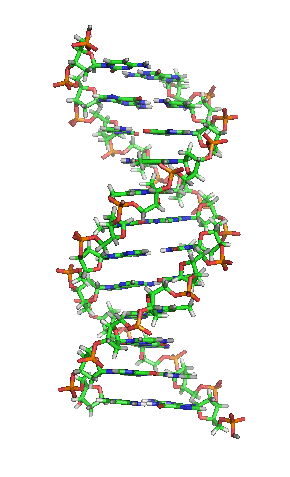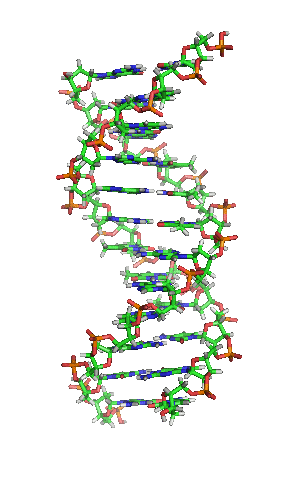

Adobre héliceé unha espiral dextroxira, é dicir, ambas as cadeas denucleótidosxiran á dereita. Isto pode verificarse se nos fixamos, indo de abaixo a arriba, na dirección que seguen os segmentos das cadeas que quedan en primeiro plano. Se as dúas febras xiran á dereita dise que a dobre hélice é dextroxira, e se xiran á esquerda, levoxira. Aínda que a forma levoxira pode aparecer en hélices alternativas debido a cambios conformacionais no ADN, a conformación máis común que adopta a dobre hélice do ADN é dextroxira, xirando cada par de bases respecto ao anterior uns 36º.[66]
Cando as dúas cadeas de ADN se enrolan unha sobre a outra (sexa á dereita ou á esquerda), fórmanse ocos ou sucos entre unha febra e a outra, deixando expostos os laterais dasbases nitroxenadasdo interior (ver a animación). Na conformación máis común que adopta o ADN aparecen, como consecuencia dos ángulos formados entre osazucresde ambas as cadeas de cada par de bases nitroxenadas, dous tipos de sucos arredor da superficie da dobre hélice: un deles, a fenda ou suco maior, que mide 22Å(2,2nm) de largo, e o outro, a fenda ou suco menor, que mide 12 Å (1,2 nm) de largo.[67]Cada volta de hélice (paso de rosca), que é o tramo no que esta fixo un xiro de 360º, ou o que é o mesmo, o tramo que vai desde o principio do suco maior ao final do suco menor, medirá, polo tanto, 34 Å, e en cada unha desas voltas hai uns 10,5pb.[5]

A largura do suco maior implica que os extremos das bases son máis accesibles alí, de forma que a cantidade de grupos químicos expostos tamén é maior, o cal facilita a distinción entre os pares de bases A-T, T-A, C-G, G-C. Como consecuencia, tamén se verá facilitado o recoñecemento de secuencias de ADN por parte de diferentesproteínassen a necesidade de abrir a dobre hélice. Así, proteínas como osfactores de transcriciónque poden unirse a secuencias específicas, frecuentemente contactan cos laterais das bases expostos no suco maior.[68]Ao contrario, os grupos químicos que quedan expostos no suco menor son similares, de forma que o recoñecemento dos pares de bases é máis difícil; por iso se di que o suco maior contén máis información có suco menor.[66]
Sentido e antisentido
[editar|editar a fonte]Unha secuencia de ADN denomínase "sentido"se a súa secuencia é a mesma que a secuencia dunARN mensaxeiroque setraducenunha proteína (pero aARN polimerasenon fixo a transcrición do ARNm complementario a partir da febra sentido senón daantisentido). A secuencia da febra de ADN complementaria denomínase"antisentido".Polo tanto, o ARNm ten unha secuencia complementaria da febra antisentido (da cal se transcribiu) e igual á da febra sentido. En ambas as febras de ADN da dobre hélice poden existir tanto secuenciassentido,que codifican ARNm, comaantisentido,que non o codifican. É dicir, as secuencias que codifican ARNm non están todas presentes nunha soa das febras, senón repartidas entre as dúas febras. Tanto enprocariotascoma eneucariotasprodúcense ARNs con secuenciasantisentido,pero a función deses ARNs non está completamente clara.[69]Propúxose que osARNantisentidoestán implicados naregulación da expresión xénicapor medio de apareamentos ARN-ARN: os ARNs antisentido aparéanse cos ARNm complementarios, bloqueando desta forma a súa tradución.[70]
Nunhas poucas secuencias de ADN en procariotas e eucariotas (este feito é máis frecuente enplásmidosevirus), a distinción entre febrassentidoeantisentidoé máis difusa, debido a que presentan xenes superpostos.[71]Nestes casos, algunhas secuencias de ADN teñen unha función dobre, codificando unha proteína cando se le ao longo dunha febra, e outra proteína cando se le na dirección contraria ao longo da outra febra. Enbacterias,esta superposición pode estar implicada na regulación da transcrición doxene,[72]mentres que en virus os xenes superpostos aumentan a cantidade de información que pode codificarse nos seus diminutos xenomas.[73]
Superenrolamento
[editar|editar a fonte]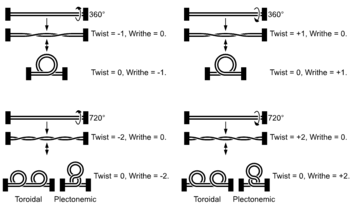
O ADN pode retorcerse como unha corda nun proceso que se denominasuperenrolamento do ADN.Cando o ADN está nun estado "relaxado", unha cadea normalmente xira arredor do eixe da dobre hélice unha vez cada 10,4 pares de bases, pero se o ADN está retorto as febras poden estar unidas máis estreitamente ou máis relaxadamente.[74]Se o ADN está retorto na dirección da hélice, dise que o superenrolamento é positivo, e as bases mantéñense xuntas de forma máis estreita. Se o ADN se retorce na dirección oposta, o superenrolamento chámase negativo, e as bases afástanse. Na natureza, a maior parte do ADN ten un lixeiro superenrolamento negativo que é producido porencimasdenominadostopoisomerases.[75]Estes encimas tamén son necesarios para liberar as forzas de torsión introducidas nas febras de ADN durante procesos como a transcrición e areplicación.[76]
Modificacións químicas
[editar|editar a fonte]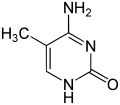
Modificacións de bases
[editar|editar a fonte]- Artigos principais:Metilación do ADNeCromatina.
A expresión dosxenesestá influenciada pola forma na que o ADN está empaquetado en cromosomas, nunha estrutura denominadacromatina.As modificacións de bases poden estar implicadas no empaquetamento do ADN: as rexións que presentan unhaexpresión xénicabaixa ou nula normalmente conteñen niveis altos demetilacióndas basescitosina.Por exemplo, a metilación de citosina produce5-metilcitosina,que é importante para ainactivación do cromosoma X.[77]O nivel medio de metilación varía entre organismos: o vermeCaenorhabditis elegansnon presenta metilación de citosina, mentres que osvertebradospresentan un nivel alto, de modo que ata o 1% do seu ADN contén 5-metilcitosina.[78]Malia a importancia da 5-metilcitosina, esta podedesaminarsepara xerar unha basetimina.As citosinas metiladas son, polo tanto, particularmente sensibles ásmutacións.[79]Outras modificacións de bases inclúen ametilacióndeadeninaenbacterias,e aglicosilaciónque orixina a "base J"encinetoplástidos(que pode considerarse unuraciloglicosilado).[80][81]
Danos no ADN
[editar|editar a fonte]- Artigo principal:Mutación.

O ADN pode quedar danado pola acción de moitos tipos de mutáxenos que cambian a secuencia do ADN, como poden ser:axentes alquilantes,eradiación electromagnéticade alta enerxía, como luzultravioleta(UV) eraios X.O tipo de dano producido no ADN depende do tipo de mutáxeno. Por exemplo, a luz UV pode danar o ADN producindodímeros de timina,que se forman por ligamento cruzado entre basespirimidínicas.[83]Por outro lado, oxidantes tales comoradicais libresou operóxido de hidróxenoproducen múltiples danos, incluíndo modificacións de bases, sobre todo guanina, e roturas da dobre cadea.[84]Nunha célula humana calquera, arredor de 500 bases sofren dano oxidativo cada día.[85][86]Destes danos oxidativos, os máis perigosos son as roturas da dobre cadea, xa que son difíciles de arranxar e poden producirmutacións puntuais,inserciónsedeleciónsdasecuencia de ADN,outranslocacións cromosómicas.[87]
Moitosmutáxenoscolócanse entre dous pares de bases adxacentes, polo que se denominanaxentes intercalantes.A maioría dos axentes intercalantes son moléculasaromáticase planas, como obromuro de etidio,adaunomicina,adoxorrubicinae atalidomida.Para que un axente intercalante poida integrarse entre dous pares de bases, estas deben separarse, distorsionando as febras de ADN e abrindo a dobre hélice. Isto inhibe a transcrición e areplicación do ADN,causando toxicidade e mutacións. Xa que logo, os axentes intercalantes do ADN son frecuentementecarcinóxenos:obenzopireno,asacridinas,aaflatoxinae obromuro de etidioson exemplos ben coñecidos.[88][89][90]Porén, debido á súa capacidade para inhibir a replicación e a transcrición do ADN, estas toxinas tamén se utilizan enquimioterapiapara inhibir o rápido crecemento das célulascancerosas.[91]
Os danos no ADN inician unha resposta que activa diferentes mecanismos de reparación que recoñecen danos específicos no ADN, que son reparados no momento para recuperar a secuencia orixinal do ADN. Igualmente, o dano no ADN provoca a parada dociclo celular,que implica a alteración de numerosos procesos fisiolóxicos, o que á súa vez implica síntese, transporte e degradación de proteínas (véxase taménciclo celular). Alternativamente, se o dano xenómico é demasiado grande para que poida ser amañado, os mecanismos de control inducirán a activación dunha serie de vías celulares que levarán áapoptoseou morte celular.[92]
Funcións biolóxicas
[editar|editar a fonte]As funcións biolóxicas do ADN inclúen o almacenamento de información (xenes e xenoma), a codificación de proteínas (transcrición e tradución) e a súa autoduplicación (replicación do ADN) para asegurar a transmisión da información ás células fillas durante adivisión celular.[36]
Xenes e xenoma
[editar|editar a fonte]- Artigos principais:Núcleo celular,Cromatina,CromosomaeXenoma.
O ADN pódese considerar como un almacén que contén a información (mensaxe) necesaria para construír e soster o organismo no que reside, a cal se transmite de xeración en xeración. O conxunto de información que cumpre esta función nun organismo dado denomínasexenoma,e o ADN que o contén, ADN xenómico.[93]
O ADN xenómico (que se organiza en moléculas decromatina,que á súa vez se ensamblan encromosomas) encóntrase nonúcleo celulardoseucariotas,ademais de pequenas cantidades nasmitocondriasecloroplastos.Enprocariotas,o ADN atópase nunha formación irregular denominadanucleoide.[94]
O ADN codificante
[editar|editar a fonte]
- Artigo principal:Xene.
A información xenética dunxenomaestá contida nosxenes,e o conxunto de toda a información que corresponde a un organismo denomínasexenotipo.Un xene é unha unidade de herdanza e é unha rexión de ADN que inflúe nunha característica particular dun organismo (como acor dos ollos,por exemplo). Os xenes conteñen un "marco aberto de lectura"que pode transcribirse, ademais desecuencias reguladoras,tales comopromotoreseamplificadores(enhancers),[96]que controlan a transcrición do marco aberto de lectura.
Desde este punto de vista, astraballadorasdeste mecanismo son as proteínas. Estas poden serestruturais,como as proteínas dosmúsculos,cartilaxes,pelo etc., oufuncionais,como ahemoglobinaou os innumerablesencimasdo organismo. A función principal da herdanza é a especificación das proteínas, sendo o ADN unha especie deplanooureceitapara producilas. A maior parte das veces a modificación do ADN provocará unha disfunción proteica que dará lugar á aparición dalgunhaenfermidade.Pero en determinados casos, as modificacións poderán provocar cambios beneficiosos que darán lugar a individuos mellor adaptados ao seu contorno.[14]
As máis de trinta mil proteínas diferentes do corpo humano están constituídas por vinteaminoácidosdiferentes, e unha molécula de ADN debe especificar a secuencia en que se unen ditos aminoácidos.
No proceso de elaborar unha proteína, o ADN dun xene lese e transcríbese aARN.Este ARN serve como mensaxeiro entre o ADN e amaquinariaque elaborará as proteínas e por iso recibe o nome deARN mensaxeiroou ARNm. O ARN mensaxeiro serve de molde á maquinaria que elabora as proteínas, para que ensamble os aminoácidos na orde precisa paraarmar a proteína.[14]
Odogma central da bioloxía molecularestablecía que o fluxo de actividade e de información era: ADN → ARN → proteína.[97]Non obstante, xa pouco despois da súa formulación se demostrou que este "dogma" debía ser ampliado, porque se encontraron outros fluxos de información. Hoxe sabemos que nalgúns organismos (virus de ARN) e nosretrotransposónsa información flúe de ARN a ADN; este proceso coñécese comotranscrición inversa,tamén chamada retrotranscrición ou reversotranscrición. Ademais, sábese que existen secuencias de ADN que se transcriben a ARN e son funcionais como tales, sen chegar a traducirse nunca a proteínas: son osARN non codificantes,como por exemplo osARN interferentes.[49][50]
O ADN non codificante
[editar|editar a fonte]- Artigo principal:ADN non codificante.
O ADN do xenoma dun organismo pode dividirse conceptualmente en dous: o que codifica as proteínas (os xenes) e o que non codifica. En moitasespecies,só unha pequena fracción doxenomacodifica proteínas. Por exemplo, só arredor do 1,5% doxenoma humanoconsiste enexónsque codifican proteínas (20.000 a 25.000 xenes), mentres que máis do 90% consiste enADN non codificante.[98]
O ADN non codificante (tamén denominado por veces e con menos precisiónADN lixo) corresponde a secuencias do xenoma que non xeran unha proteína (procedentes de transposicións, duplicacións, translocacións e recombinacións de virus etc.), incluíndo osintróns.Ata hai pouco tempo pensábase que a maior parte do ADN non codificante non tiña utilidade ningunha, mais estudos recentes indican que iso é inexacto. Entre outras funcións, postúlase que parte do chamado "ADN lixo" regula aexpresión diferencial dos xenes.[99]Por exemplo, algunhas secuencias teñen afinidade por proteínas especiais que teñen a capacidade de unirse ao ADN (como oshomeodominios,os complexos receptores dehormonasesteroidesetc.), cun papel importante no control dos mecanismos de transcrición e replicación. Estas secuencias chámanse frecuentemente "secuencias reguladoras",e os investigadores supoñen que só se levan identificado unha pequena fracción das que realmente existen. A presenza de tanto ADN non codificante en xenomas eucarióticos e as diferenzas entamaño do xenomaentre especies representan unha incógnita a resolver que é coñecida como o "enigma do valor C".[100] Recentemente, un grupo de investigadores da Universidade de Yale descubriu unha secuencia de ADN non codificante que sería a responsable de que os seres humanos desenvolveran a capacidade de agarrar e/ou manipular obxectos ou ferramentas.[101]
Por outro lado, algunhas secuencias de ADN desempeñan un papel estrutural nos cromosomas: ostelómerosecentrómerosconteñen poucos ou ningún xene codificante de proteínas, pero son importantes para estabilizaren a estrutura dos cromosomas.[60][102]Algúns xenes non codifican proteínas, pero si se transcriben en ARN:ARN ribosómico,ARN de transferenciaeARN interferente(que é ARN que bloquea a expresión de xenes específicos). A estrutura de intróns e exóns dalgúns xenes (como os deinmunoglobulinaseprotocadherinas) son importantes por permitiren osempalmes alternativosdopre-ARN mensaxeiroque fan posible a síntese de diferentes proteínas a partir dun mesmo xene (sen esta capacidade non existiría o sistema inmune, por exemplo). Algunhas secuencias de ADN non codificante representanpseudoxenesque teñen valor evolutivo, xa que permiten a creación de novos xenes con novas funcións.[50][103][104]Outros ADN non codificantes proceden da duplicación de pequenas rexións do ADN; isto ten moita utilidade, xa que o rastrexo destas secuencias repetitivas permite estudosfiloxenéticos.[105]
Transcrición e tradución
[editar|editar a fonte]- Artigos principais:Transcrición (xenética),Tradución (proteínas)eCódigo xenético.
Nunxene,asecuencia de nucleótidosao longo dunha febra de ADN transcríbese a unARN mensaxeiroe esta secuencia á súa vez tradúcese a unhaproteínaque un organismo é capaz de sintetizar ou "expresar" nun ou varios momentos da súa vida, usando a información de dita secuencia.
A relación entre a secuencia de nucleótidos e a secuencia deaminoácidosda proteína vén determinada polocódigo xenético,que se utiliza durante o proceso de tradución ousíntese de proteínas.[106]A unidade codificadora do código xenético é un grupo de tres nucleótidos (triplete), representado polas tres letras iniciais das bases nitroxenadas (por exemplo, ACT, CAG, TTT). Os tripletes do ADN transcríbense nas súas bases complementarias no ARN mensaxeiro, e neste caso os tripletes denomínansecodóns(para o exemplo anterior, UGA, GUC, AAA).[107]Noribosomacada codón do ARN mensaxeiro interacciona cunha molécula deARN de transferenciaque conteña o triplete complementario, denominadoanticodón.Cada ARNt leva o aminoácido correspondente ao codón de acordo cocódigo xenético,de modo que o ribosoma vai unindo os aminoácidos para formar unha nova proteína de acordo coas "instrucións" da secuencia do ARNm. Existen 64 codóns posibles, de modo que corresponde máis dun para cada aminoácido (por iso se di que o código xenético é un código dexenerado: non é unívoco). Algúns codóns indican a terminación da síntese, o fin da secuencia codificante; estescodóns de terminación,tamén chamados de parada, destopousen sentido,son UAA, UGA e UAG.[49]
Replicación do ADN
[editar|editar a fonte]
- Artigo principal:Replicación do ADN.
A replicación do ADN é o proceso polo cal se obteñen copias ou réplicas idénticas dunha molécula de ADN. A replicación é fundamental para a transferencia da información xenética dunha xeración á seguinte e, polo tanto, é a base da herdanza. O mecanismo consiste esencialmente na separación das dúas febras da dobre hélice, as cales serven de molde para a posterior síntese de cadeas complementarias a cada unha delas. O proceso é diferente en cada unha das febras debido ao mecanismo de funcionamento daADN polimerase.O resultado final son dúas moléculas idénticas á orixinal. Este tipo de replicación denomínasesemiconservativa(ou semiconservadora) debido a que cada unha das dúas moléculas orixinadas na duplicación presenta unha cadea procedente da molécula "nai" e outra acabada de sintetizar.[108][109]
Interaccións ADN-proteína
[editar|editar a fonte]Todas as funcións do ADN dependen das súas interaccións con proteínas. Estas interaccións poden ser inespecíficas, ou a proteína pode unirse de forma específica a unha única secuencia de ADN. Tamén poden unirse ao ADNencimas,entre os cales son especialmente importantes as polimerases, que copian as secuencias de bases do ADN durante a transcrición e a replicación.
Proteínas que se unen ao ADN
[editar|editar a fonte]- Artigos principais:HistonaeNucleosoma.
Interaccións inespecíficas
[editar|editar a fonte]
|
As proteínas estruturais que se unen ao ADN son exemplos ben coñecidos de interaccións inespecíficas ADN-proteínas. Nos cromosomas, o ADN forma complexos con proteínas estruturais. Estas proteínas organizan o ADN nunha estrutura compacta denominadacromatina.Eneucariotasa formación desta estrutura implica a unión do ADN a un complexo formado por pequenas proteínas básicas denominadashistonas,mentres que enprocariotasestán implicadas unha gran variedade de proteínas.[110][111]As histonas forman un complexo de forma cilíndrica denominadonucleosoma,arredor do cal se enrola a dobre hélice do ADN dando case dúas voltas. Estas interaccións inespecíficas quedan determinadas pola existencia de residuos básicos nas histonas, que formanenlaces iónicosco esqueleto de azucre-fosfato do ADN e, polo tanto, son en gran parte independentes da secuencia de bases.[112]Estes aminoácidos básicos experimentan modificacións químicas comometilación,fosforilacióneacetilación,[113]que alteran a forza da interacción entre o ADN e as histonas, facendo ao ADN máis ou menos accesible aosfactores de transcricióne, polo tanto, modificando a taxa de transcrición.[114]
Outras proteínas que se unen ao ADN de maneira inespecífica na cromatina inclúen asproteínas do grupo de alta mobilidade(HMG) que se unen ao ADN pregado ou distorsionado.[115]Estas proteínas son importantes durante o pregamento dos nucleosomas, organizándoos en estruturas máis complexas para constituíren os cromosomas[116]durante o proceso de condensación cromosómica. Propúxose que neste proceso tamén intervirían outras proteínas, formando unha especie de "andamio" sobre o cal se organiza a cromatina; os principais compoñentes desta estrutura serían o encimatopoisomeraseII α(outopoIIalpha) e acondensina13S.[117]Porén, o papel estrutural da topoisomerase II alfa na organización dos cromosomas aínda se discute, xa que algúns investigadores argumentan que este encima se intercambia rapidamente tanto nos brazos cromosómicos coma noscinetocorosdurante amitose.[118]
Interaccións específicas
[editar|editar a fonte]Un grupo ben definido de proteínas que se ligan ao ADN é o conformado polas proteínas que se unen especificamente aoADN monocatenarioou ADN de febra sinxela (ssDNA). En humanos, a proteína A de replicación é a mellor coñecida da súa familia e actúa en procesos nos que a dobre hélice se separa, como areplicación do ADN,arecombinaciónou areparación do ADN.[119]Estas proteínas parecen estabilizar o ADN monocatenario, protexéndoo para evitar que forme estruturas detalo-lazoou de que sexa degradado pornucleases.[120]

Porén, outras proteínas evolucionaron para unirse especificamente a secuencias determinadas de ADN. A especificidade da interacción das proteínas co ADN procede dos múltiples contactos coas bases do ADN, o que lles permite "ler" a secuencia do ADN. A maioría desas interaccións coas bases ocorre nosuco maior,onde as bases son máis accesibles.[122]
As proteínas específicas estudadas con maior detalle son as encargadas de regular a transcrición, denominadasfactores de transcrición.[123]Cada factor de transcrición únese a unha secuencia concreta de ADN e activa ou inhibe a transcrición dos xenes que presentan estas secuencias próximas aos seuspromotores.Os factores de transcrición poden efectuar isto de dúas formas:
- En primeiro lugar, poden unirse áARN polimeraseresponsable da transcrición, ben directamente ou a través doutras proteínas mediadoras. Desta forma, estabilízase a unión entre a ARN polimerase e opromotor,o que permite o inicio da transcrición.[124]
- En segundo lugar, os factores de transcrición poden unirse aencimasque modifican ashistonasdo promotor, o que altera a accesibilidade do molde de ADN á ARN polimerase.[125]
Como os ADN diana poden encontrarse por todo oxenomado organismo, os cambios na actividade dun tipo de factor de transcrición poden afectar a miles dexenes.En consecuencia, estas proteínas son frecuentemente as dianas dos procesos detransdución de sinaisque controlan as respostas a cambios ambientais ou adiferenciacióne desenvolvemento celular.[126]

Encimas que modifican o ADN
[editar|editar a fonte]Nucleases e ligases
[editar|editar a fonte]Asnucleasessonencimasque cortan as febras de ADNcatalizandoahidrólisedosenlaces fosfodiéster.[128]As nucleases que hidrolizannucleótidosa partir dos extremos das febras de ADN denomínanseexonucleases,mentres que as que cortan no interior das febras sonendonucleases.[129]As nucleases máis utilizadas enbioloxía molecularson osencimas de restrición,que son endonucleases que cortan o ADN onde este ten determinadas secuencias de nucleótidos específicas. Por exemplo, o encimaEcoRV,que se mostra na figura da esquerda, recoñece a secuencia de 6 bases 5′-GAT|ATC-3′, e fai un corte en ambas as febras do ADN na liña vertical indicada, xerando dúas moléculas de ADN cos extremos romos. Outros encimas de restrición xeran extremos cohesivos, xa que cortan de forma diferente as dúas febras de ADN.[130]Na natureza, estes encimas preséntanse nasbacterias,e protexen ás mesmas contra as infeccións defagos,ao dixeriren o ADN de ditos fagos cando entra a través da parede bacteriana, actuando como un mecanismo de defensa.[131]Enbiotecnoloxía,estas nucleases específicas de secuencias do ADN utilízanse enenxeñaría xenéticaparaclonarfragmentos de ADN,[132]e na técnica dapegada xenética.[133]
Os encimas denominadosADN ligasespoden unir febras de ADN cortadas ou rotas.[134]As ligases son particularmente importantes nareplicaciónda febra que sofre replicación descontinua no ADN (febra retardada), xa que unen os fragmentos (de Okazaki) curtos de ADN xerados naforquita de replicaciónpara formar unha copia completa do molde de ADN. Tamén se utilizan nareparación do ADNe en procesos derecombinación xenética.[134]
Topoisomerases e helicases
[editar|editar a fonte]Astopoisomerasesson encimas que posúen á vez actividade nuclease e ligase. Estas proteínas varían a cantidade deADN superenrolado.Algúns destes encimas funcionan cortando a hélice de ADN e permitindo que unha sección rote, de maneira que reducen o grao de superenrolamento. Unha vez feito isto, o encima volve unir os fragmentos de ADN.[75]Outros tipos de encimas poden cortar unha hélice de ADN e logo pasar a segunda febra de ADN a través da rotura, antes de unir as hélices.[135]As topoisomerases son necesarias para moitos procesos nos que intervén o ADN, como areplicación do ADNe atranscrición.[76]
Ashelicasesson unhas proteínas que pertencen ao grupo dosmotores moleculares.Utilizan enerxía química almacenada nosnucleósidos trifosfatos,fundamentalmenteATP,para romperenpontes de hidróxenoentre bases e separaren a dobre hélice de ADN en febras simples.[136]Estes encimas son esenciais para a maioría dos procesos nos que os encimas necesitan acceder ás bases do ADN.[137]
Polimerases
[editar|editar a fonte]- Artigos principais:ADN polimeraseeARN polimerase.
Aspolimerasessonencimasque sintetizan cadeas de nucleótidos a partir de nucleósidos trifosfato. A secuencia dos seus produtos é copia de cadeas de polinucleótidos existentes, que se denominanmoldes.Estes encimas funcionan engadindo nucleótidos ao grupohidroxiloen 3' do nucleótido previo da cadea de ADN. En consecuencia, todas as polimerases funcionan en dirección 5′ → 3′.[138]Nossitios activosdestes encimas, o nucleósido trifosfato que se incorpora aparea a súa base coa correspondente no molde: isto permite que a polimerase sintetice de forma precisa a cadea complementaria ao molde.
As polimerases clasifícanse de acordo co tipo de molde que utilizan:
- Nareplicación do ADN,unhaADN polimerase dependente de ADNrealiza unha copia de ADN a partir dunha secuencia de ADN. A precisión é vital neste proceso, polo que moitas destas polimerases teñen unha actividade de comprobación da lectura. Grazas a esta actividade, a polimerase recoñece erros ocasionais no apareamento de bases durante a síntese. Se se detecta un destes erros de apareamento, actívase unha actividade deexonucleaseen dirección 3′ → 5′ e a base incorrecta elimínase.[139]Na maioría dos organismos as ADN polimerases funcionan nun gran complexo denominadoreplisoma,que contén múltiples unidades accesorias, comohelicases.[140]
- AsADN polimerases dependentes de ARNson unha clase especializada depolimerasesque copian a secuencia dunha febra de ARN en ADN. Inclúen atranscriptase inversa,que é un encimaviralimplicado na infección de células porretrovirus,e atelomerase,que é necesaria para a replicación dos telómeros.[59][141]A telomerase é unha polimerase infrecuente, porque contén o seu propio molde de ARN como parte da súa estrutura.[60]
- Atranscriciónlévase a cabo por unhaARN polimerase dependente de ADNque copia a secuencia dunha das febras de ADN en ARN. Para empezar a transcribir un xene, aARN polimeraseúnese a unha secuencia do ADN denominadapromotor,e separa as febras do ADN. Entón copia a secuencia do xene nun transcrito deARN mensaxeiroata que acada unha rexión do ADN denominadaterminador,onde se detén e deslígase do ADN. Como acontece coas ADN polimerases dependentes de ADN en humanos, aARN polimerase II(o encima que transcribe a maioría dos xenes doxenoma humano) funciona como un grancomplexo multiproteicoque contén múltiples subunidades reguladoras e accesorias.[142]
Recombinación xenética
[editar|editar a fonte]
|

|
- Artigo principal:Recombinación xenética.

Unha hélice de ADN normalmente non interacciona con outros segmentos de ADN, e nas células humanas os diferentes cromosomas mesmo ocupan áreas separadas nonúcleo celular,denominadas “territorios cromosómicos”.[144]A separación física dos diferentes cromosomas é importante para que o ADN manteña a súa capacidade de funcionar como un almacén estable de información. Un dos poucos momentos nos que os cromosomas interaccionan é durante osobrecruzamentocromosómico, durante o cal serecombinan.O sobrecruzamento cromosómico ocorre cando dous cromosomas homólogos intercambian segmentos, para o cal as dúas hélices de ADN rompen, intercámbianse e únense de novo.[145]
A recombinación permite que os cromosomas intercambien información xenética e produce novas combinacións de alelos, o que aumenta a eficiencia daselección naturale pode ser importante na evolución rápida de novas proteínas.[146]Durante aprofase Idameiose,unha vez que os cromosomas homólogos están perfectamente apareados formando estruturas chamadas bivalentes, prodúcese osobrecruzamentoou entrecruzamento(crossing-over),no cal ascromátideshomólogas (non irmás), procedentes do pai e da nai, intercambian material xenético. Como consecuencia do sobrecruzamento prodúcese unha recombinación xenética, que acrecenta en gran medida avariabilidade xenéticaentre a descendencia de proxenitores que se reproducen porvía sexual.A recombinación xenética tamén pode estar implicada nareparación do ADN,en particular na resposta celular ás roturas de dobre febra do ADN.[147]
A forma máis frecuente de sobrecruzamento cromosómico é arecombinación homóloga,na que os dous cromosomas implicados comparten secuencias moi similares. A recombinación non homóloga pode ser daniña para as células, xa que pode producirtranslocacións cromosómicase anomalías xenéticas. A reacción de recombinación está catalizada por encimas coñecidos comorecombinases,tales comoRAD51.[148]O primeiro paso no proceso de recombinación é unha rotura de dobre febra no ADN, causada por unhaendonucleaseou por un dano no ADN.[149]Posteriormente, unha serie de pasos catalizados en parte pola recombinase, conducen á unión das dúas hélices formando polo menos unhaunión de Holliday,na que un segmento dunha febra simple é aliñado ou hibridado (annealed) coa febra complementaria na outra hélice. A unión de Holliday é unha estrutura de unión tetraédrica que pode moverse ao longo do par de cromosomas, intercambiando unha febra por outra. A reacción de recombinación detense polo corte da unión e a subseguinte reunión dos segmentos de ADN libres.[150]
Evolución do metabolismo de ADN
[editar|editar a fonte]- Artigos principais:Hipótese do mundo de ARNeOrixe da vida.
O ADN contén a información xenética que permite á maioría dos organismos viventes funcionar, crecer e reproducirse. Porén, non está claro durante canto tempo leva exercendo esta función nos ~3000 millóns de anos dahistoria da vida,xa que se suxeriu que as formas de vida máis temperás poderían ter utilizadoARNcomo material xenético.[151][152]O ARN puido funcionar inicialmente como a molécula central dun metabolismo primixenio, xa que pode transmitir información xenética e simultaneamente actuar comocatalizadorformando parte deribozimas.[153]Este antigomundo de ARNonde os ácidos nucleicos funcionarían como catalizadores e como almacéns de información xenética puido influír naevolucióndocódigo xenéticoactual, baseado en catronucleótidos.Isto deberíase a que o número de bases diferentes nun organismo é un compromiso entre un número pequeno de bases (o que aumentaría a precisión da replicación) e un número grande de bases (que aumentaría a eficiencia catalítica dosribozimas).[154]
Porén, non temos evidencias directas dos sistemas xenéticos primitivos, xa que a recuperación do ADN a partir da maior parte dos fósiles é imposible, porque o ADN só pode sobrevivir no medio ambiente durante menos dun millón de anos, e logo empeza a degradarse lentamente en fragmentos de menor tamaño en solución.[155]Algunhas investigacións pretenden que se obtivo ADN máis antigo, por exemplo un informe sobre o illamento dunha bacteria viable a partir dun cristal salino de 250 millóns de anos de antigüidade,[156]mais estes datos son controvertidos.[157][158]
Non obstante, poden utilizarse ferramentas de evolución molecular para inferir os xenomas de organismos primitivos a partir de organismos contemporáneos.[159][160]En moitos casos, estas inferencias son suficientemente fiables, de maneira que unhabiomoléculacodificada nun xenoma primitivo pode resucitarse no laboratorio para ser estudada hoxe.[161][162]Unha vez que a biomolécula primixenia se resucitou, as súas propiedades poden ofrecer inferencias sobre ambientes e estilos de vida primixenios. Este proceso relaciónase co campo emerxente dapaleoxenética experimental.[163]
Malia todo, o proceso de traballocara atrásdesde o presente ten limitacións inherentes, razón pola cal outros investigadores tratan de aclarar o mecanismo evolutivo traballando desde a orixe da Terra en diante. Tendo suficiente información sobre a química no cosmos, o xeito en que as substancias cósmicas puideron depositarse na Terra, e as transformacións que puideron ter lugar na superficie terrestre primixenia, quizais poderiamos aprender o suficiente sobre as orixes para desenvolver modelos de evolución ulterior da información xenética[164].
Técnicas máis comúns
[editar|editar a fonte]O coñecemento da estrutura do ADN permitiu o desenvolvemento de moitas ferramentas tecnolóxicas que explotan as súas propiedades fisicoquímicas para analizar a súa implicación en problemas concretos: por exemplo, desde análisesfiloxeńeticaspara detectar semellanzas entre diferentestaxons,á caracterización da variabilidade individual dun paciente na súa resposta a un determinadofármaco,pasando por un enfoque global, a nivelxenómico,de calquera característica específica nun grupo de individuos de interese. [165]
Podemos clasificar as metodoloxías de análise do ADN en dous grupos: (1) as que procuran a súa multiplicación, xa sexain vivo,como areacción en cadea da polimerase(PCR), ouin vitro,como aclonación,e (2) as que explotan as propiedades específicas de elementos concretos, ou de xenomas clonados, como é o caso dasecuenciación do ADNe da hibridación consondasespecíficas ( "Southern blot"echips de ADN).
Tecnoloxía do ADN recombinante
[editar|editar a fonte]A tecnoloxía doADN recombinante,pedra angular daenxeñaría xenética,permite multiplicar en grandes cantidades un fragmento de ADN de interese, o cal se di que foi clonado. Para iso, debe introducirse dito fragmento noutro elemento de ADN, xeralmente unplásmido,que posúe na súa secuencia os elementos necesarios para que a maquinaria celular dun hospedador, normalmente a bacteriaEscherichia coli,o replique. Deste modo, unha veztransformadaa cepa bacteriana, o fragmento de ADN clonado reprodúcese cada vez que a bacteria se divide.[166]
Para clonar a secuencia de ADN de interese, empréganseencimascomo ferramentas de corte e empalme do fragmento e do vector (oplásmido). Ditos encimas pertencen a dous grupos: en primeiro lugar, osencimas de restrición,que posúen a capacidade de recoñecer e cortar secuencias específicas; en segundo lugar, aADN ligase,que establece unenlace covalenteentre extremos de ADN compatibles[165](ver secciónNucleases e ligases).
Secuenciación
[editar|editar a fonte]- Artigo principal:Secuenciación do ADN.
A secuenciación do ADN consiste en determinar a orde dosnucleótidosdun polímero de ADN de calquera lonxitude, pero adoita dirixirse á determinación dexenomascompletos, debido a que as técnicas actuais permiten realizar esta secuenciación a gran velocidade, o cal foi de grande importancia para proxectos de secuenciación a grande escala como oProxecto Xenoma Humano.Outros proxectos relacionados, en ocasións froito da colaboración entre científicos a escala mundial, estableceu a secuencia completa do ADN de moitosxenomasde animais, plantas emicroorganismos.[167]
O método de secuenciación deSanger[168]foi o máis empregado durante oséculo XX.Baséase na síntese de ADN en presenza dedesoxirribonucleósidostrifosfato (ddNTP), compostos que, a diferenza dosdesoxinucleósidosnormais (dNTPs), carecen dun grupo hidroxilo no seu extremo 3'. Aínda que os didesoxinucleósidos trifosfatados poden incorporarse á cadea en síntese, a carencia dun extremo 3'-OH imposibilita a xeración dun novoenlace fosfodiésterconucleótidoseguinte; polo tanto, provocan o remate da síntese. Por esta razón, o método de secuenciación tamén se denomina «de terminación de cadea». A reacción realízase usualmente preparando un tubo co ADN molde, a polimerase, uncebadorouprimer,desoxinucleósidos normais e unha pequena cantidade de didesoxinucleósidos marcados confluorescenciana súabase nitroxenada.Deste modo, o ddTTP pode ir marcado en azul, o ddATP en vermello etc. Durante a polimerización, vanse truncando as cadeas crecentes, ao chou, en distintas posicións. Polo tanto, prodúcese unha serie de produtos de distinto tamaño, coincidindo a posición da terminación coa incorporación do ddNTP correspondente. Unha vez terminada a reacción, é posible someter a mestura a unhaelectroforese capilar(que separa todos os fragmentos segundo a súa lonxitude) na cal se le a fluorescencia para cada posición do polímero. No noso exemplo, a lectura azul-vermello-azul-azul traduciríase como TATT.[169][170]
Reacción en cadea da polimerase (PCR)
[editar|editar a fonte]- Artigo principal:Reacción en cadea da polimerase.
A reacción en cadea da polimerase, xeralmente coñecida polas súas siglas en inglés como PCR, é unha técnica debioloxía moleculardescrita en1986porKary Mullis,[171]cuxo obxectivo é obter un gran número de copias dun fragmento de ADN dado, partindo dunha escasa cantidade inicial. Para iso, emprégase unhaADN polimerasetermoestable que, en presenza dunha mestura dos catrodesoxinucleótidos,untampónda forza iónica axeitada e oscatiónsprecisos para a actividade do encima, dous oligonucleótidos (denominados cebadores ouprimers) complementarios dunha parte da secuencia (situados a distancia suficiente e en direcciónantiparalela) e baixo unhas condicións de temperatura adecuadas, controladas por un aparello denominadotermociclador,xera exponencialmente novos fragmentos de ADN semellantes ao orixinal e acoutados polos dous cebadores.[166]
A PCR pode efectuarse como unha técnica de punto final, é dicir, como unha ferramenta de xeración do ADN desexado, ou como un método continuo, no que se avalíe dita polimerización a tempo real. Esta última variante é común naPCR cuantitativa.[165]
Southern blot
[editar|editar a fonte]- Artigo principal:Southern blot.
O método de «transferencia de Southern»[172]ou «Southern blot» permite a detección dunha secuencia de ADN nunha mostra complexa ou non do ácido nucleico. Combina unha separación mediantemasaecarga(efectuada por medio dunhaelectroforese en xel) cunha hibridación cunhasondade ácido nucleico marcada dalgún modo (xa sexa conradioactividadeou cun composto químico) que, tras varias reaccións, dea lugar á aparición dun sinal decoroufluorescencia.Dita hibridación realízase trala transferencia do ADN separado porelectroforesea unha membrana de filtro. Unha técnica semellante, pero na cal non se produce a mencionada separación electroforética denomínasedot blot.[173]
O método recibe o seu nome en honor ao seu inventor, obiólogoinglésEdwin Southern.[174]Por analoxía ao método Southern, desenvolvéronse técnicas parecidas que permiten a detección de secuencias dadas de ARN (métodonorthern,que emprega sondas de ARN ou ADN marcadas)[175]ou de proteínas específicas (técnicawestern,baseada no uso deanticorpos).[176]
Chips de ADN
[editar|editar a fonte]- Artigo principal:Chip de ADN.

Os chips de ADN son coleccións deoligonucleótidosde ADN complementario dispostos en ringleiras fixadas sobre un soporte, frecuentemente de cristal. Utilízanse para o estudo de mutacións de xenes coñecidos ou para monitorizar aexpresión xénicadunha preparación de ARN.[178]
Aplicacións
[editar|editar a fonte]Enxeñaría xenética
[editar|editar a fonte]A investigación sobre o ADN ten unha grande influencia, sobre todo enmedicina,mais tamén en agricultura e gandaría, onde os obxectivos son os mesmos que coas técnicas tradicionais que o home leva utilizando desde hai milenios: domesticación, selección e cruces dirixidos para obter variedades de animais e plantas máis produtivos. A moderna bioloxía e bioquímica fan uso intensivo datecnoloxíadoADN recombinante,introducindo xenes de interese en organismos, co obxectivo de expresar unha proteína recombinante concreta, que pode ser:
- illada para o seu uso posterior: por exemplo, pódense transformarmicroorganismospara convertelos en auténticas fábricas que producen grandes cantidades de substancias útiles, comoinsulinaouvacinas,que posteriormente se illan e se utilizan terapeuticamente.[179][180][181]
- necesaria para substituír a expresión dunxeneendóxeno danado que deu lugar a unha patoloxía, o que permitiría o restablecemento da actividade normal da proteína perdida e levaría á recuperación do estado fisiolóxico normal, non patolóxico. Este é o obxectivo daterapia xénica,un dos campos nos que se está a traballar activamente en medicina, analizando vantaxes e inconvenientes de diferentes sistemas de administración do xene (virais e non virais) e os mecanismos de selección do punto de integración dos elementos xenéticos (distintos para os virus e os transposóns) no xenoma diana.[182]Neste caso, antes de pensar na posibilidade de realizar unha terapia xénica nunha determinada patoloxía, é fundamental comprender o impacto do xene de interese no desenvolvemento de dita patoloxía, para o cal é necesario o desenvolvemento dun modelo animal, eliminando ou modificando dito xene nun animal de laboratorio, por medio da técnica ‘’knockout’’. Só no caso de que os resultados no modelo animal sexan satisfactorios se procedería a analizar a posibilidade de restablecer o xene danado por terapia xénica.[183]
- utilizada para enriquecer un alimento: por exemplo, a composición do leite (unha importante fonte de proteínas para o consumo humano e animal) pode modificarse por medio da transxénese, engadindo xenes exóxenos e desactivando xenes endóxenos para mellorar o seu valor nutricional, reducir infeccións nas glándulas mamarias, proporcionar aos consumidores proteínas antipatóxenas e preparar proteínas recombinantes para o seu uso farmacéutico.[184][185]
- útil para mellorar a resistencia do organismo transformado: por exemplo en plantas pódense introducir xenes que confiren resistencia apatóxenos(virus,insectos,fungos…), ou a axentes estresantes abióticos (salinidade, seca, metais pesados…).[186][187][188]
Medicina forense
[editar|editar a fonte]- Véxase tamén:Pegada xenética.
En investigaciónforensepódese utilizar o ADN presente nosangue,oseme,as células depel,salivaoupelode restos atopados na escena dun crime para identificar ao responsable. Esta técnica denomínasepegada xenética,ou tamén "perfil de ADN". Ao realizar a proba da pegada xenética, compárase a lonxitude de seccións moi variables deADN repetitivo,como osmicrosatélites,entre persoas diferentes. Este método é moi fiable para identificar un criminal.[189]Porén, a identificación pode complicarse se a escena está contaminada con ADN de persoas diferentes.[190]A técnica da pegada xenética foi desenvolvida en 1984 polo xenetista británico SirAlec Jeffreys,[191]e utilizouse por primeira vez en medicina forense para condenar a unha persoa en Inglaterra en 1983 e 1986.[192]Pódese requirir ás persoas acusadas de certos tipos de crimes que fornezan unha mostra de ADN para introducir os resultados nunha base de datos. Isto facilitou o labor dos investigadores na resolución de casos antigos, onde só se obtivo unha mostra de ADN da escena do crime, nalgúns casos permitindo exonerar a un convicto. A pegada xenética tamén pode utilizarse para identificar cadáveres en accidentes en masa,[193]ou para realizar probas de consanguinidade.[194]
Bioinformática
[editar|editar a fonte]- Artigo principal:Bioinformática.
Abioinformáticaimplica a manipulación, procura e extracción de información dos datos da secuencia do ADN. O desenvolvemento das técnicas para almacenar e buscar secuencias de ADN supuxo avances no desenvolvemento dosoftwaredos computadores, para moitas aplicacións, especialmentealgoritmosde busca de frases, aprendizaxe automática e teorías debases de datos.[195]A procura de frases ou algoritmos de coincidencias, que buscan a presenza dunha secuencia de letras dentro dunha secuencia de letras maior, desenvolveuse para buscar secuencias específicas de nucleótidos.[196]Noutras aplicacións comoeditores de textos,poden funcionar mesmo algoritmos simples, pero as secuencias de ADN poden xerar que estes algoritmos presenten un comportamento de case-o-peor-caso, debido ao baixo número de caracteres. O problema relacionado doaliñamento de secuenciaspersegue identificar secuencias homólogas e localizarmutaciónsespecíficas que as diferencien. Estas técnicas, fundamentalmente oaliñamento múltiple de secuencias,utilízanse ao estudar as relaciónsfiloxenéticase a función das proteínas.[197]As coleccións de datos que representan secuencias de ADN do tamaño dunxenoma,tales como as producidas poloProxecto Xenoma Humano,son difíciles de usar sen anotacións, que marcan a localización dos xenes e os elementos reguladores en cadacromosoma.As rexións de ADN que teñen patróns asociados con xenes que codifican proteínas (ou ARN) poden identificarse por algoritmos de localización de xenes, o que permite aos investigadores predicir a presenza de produtos xénicos específicos nun organismo mesmo antes de que fose illado experimentalmente.[198]
Nanotecnoloxía de ADN
[editar|editar a fonte]
A nanotecnoloxía de ADN utiliza as propiedades únicas de recoñecemento molecular do ADN e outros ácidos nucleicos para crear complexos ramificados autoensamblados con propiedades útiles. Neste caso, o ADN utilízase como un material estrutural, máis que como un portador de información biolóxica.[199]Isto levou á creación de retículas periódicas de dúas dimensións usando o método deADN origami,ademais de estruturas en tres dimensións con forma de poliedros.[200] Tamén se demostrou a creación de aparellos nanomecánicos e autoensamblaxes algorítmicas,[201]e estas estruturas de ADN foron utilizadas como moldes para a colocación doutras moléculas como nanopartículas deourocoloidale moléculas da proteínaestreptavidina.[202]
Evolución, historia e antropoloxía
[editar|editar a fonte]- Artigo principal:Filoxenia.
Ao longo do tempo, o ADN almacena mutacións que se herdan e, polo tanto, contén información histórica, de maneira que comparando secuencias de ADN, os xenetistas poden inferir a historia evolutiva dos organismos, a súafiloxenia.[203]A investigación filoxenética é unha ferramenta fundamental enbioloxía evolutiva.Se comparamos as secuencias de ADN dentro dunha especie, osxenetistas de poboaciónspoden coñecer a historia de determinadas poboacións. Isto pódese utilizar nunha ampla variedade de estudos, desdeecoloxíaataantropoloxía.Por outro lado, o ADN tamén se utiliza para estudar relacións familiares recentes.[204]
Almacenamento de información
[editar|editar a fonte]En 2013 publicouse enNatureque oInstituto Europeo de Bioinformáticae a compañíaAgilent Technologiespropuxeran un mecanismo para usar a capacidade do ADN de codificar información como un medio para o almacenamento de datos dixital. O grupo conseguiu codificar 739 kilobytes de datos en código de ADN, sintetizar ese ADN, secuencialo despois e descodificar a información de novo á súa forma orixinal, cunha exactitude do 100%. A información codificada consistía en ficheiros de texto e de son. Anteriormente, publicárase un experimento realizado por investigadores daUniversidade Harvard,onde se codificara o texto dun libro de 54.000 páxinas.[205][206]
Historia da investigación do ADN
[editar|editar a fonte]
O ADN foi illado por primeira vez polo médico suízoFriedrich Miescherque, en 1869, descubriu unha substancia microscópica nopusde vendaxes de feridas. Como se atopaba no núcleo das células chamoulle "nucleína".[207]En 1919,Phoebus Leveneidentificou abase nitroxenada,o azucre e o fosfato da unidade nucleotídica.[208]Levene suxeriu que o ADN consistía nunha cadea de nucleótidos unidos uns a outros polos seus fosfatos. Porén, Levene pensaba que a cadea era moi curta e as bases repetíanse continuamente nunha orde determinada. En 1937William Astburyfixo a primeiradifracción de raios Xdo ADN, que mostraba que tiña unha estrutura regular.[209]
En 1927Nikolai Koltsovpropuxo que as características hereditarias herdábanse por medio dunha "molécula xigante hereditaria" que estaría feita de "dúas cadeas especulares que se replicarían de modo semiconservativo usando cada unha das cadeas como molde".[210]En 1928,Frederick Griffithdescubriu que o carácter "liso" de certas cepas da bacteriapneumococopodía transformarse en "rugoso" mesturando bacterias mortas "lisas" con bacterias vivas "rugosas". Algunha substancia debía pasar dunhas a outras levando a información necesaria, pero non soubo identificar cal era. Naquela época moitos pensaban que a información xenética se almacenaba nas proteínas.[211]Anos despois en 1943Oswald Avery, xunto con MacLeod e McCarty,identificaron ao ADN como molécula causante datransformaciónnoexperimento de Griffith.[212]O papel do ADN como molécula hereditaria foi confirmado nosexperimentos de Hershey e Chasecon virus en 1952.[213]
En 1953,James D. WatsoneFrancis Crickpropuxeron o primeiro modelo de dobre hélice do ADN, que foi publicado en "Nature".[214]O seu modelo de dobre hélice estaba baseado na imaxe de difracción de raios X etiquetada como "Foto 51"[215]tomada porRosalind FranklineRaymond Goslingen maio de 1952, e na información sobre o apareamento de bases obtida anos antes deErwin Chargaff.As regras de Chargaff xogaron un importante papel no establecemento das configuracións de dobre hélice dos ADN B e A.[216]
As probas experimentais que apoiaban o modelo de Watson e Crick foron publicadas nunha serie de cinco artigos nunha mesma edición deNature.[217]Destes cinco artigos, o de Franklin e Gosling foi a primeira publicación das súas propias difraccións de raios X e métodos de análise orixinais que parcialmente sostiñan o modelo de Watson e Crick;[218][219]esta edición tamén contiña un artigo da estrutura do ADN deMaurice Wilkinse dous dos seus colegas, cuxas análises e modelos de raios X do ADN Bin vivotamén apoiaban a presenzain vivoda dobre hélice do ADN proposta por Crick e Watson nas dúas páxinas previas deNature.[220]En 1962, despois da morte de Franklin, Watson, Crick, e Wilkins recibiron conxuntamente oPremio NobeldeFisioloxía e Medicina.[221]Porén, as regras do Nobel daquela época só permitían que fose outorgado a persoas vivas (Franklin xa morrera daquela), e iniciouse un forte debate que aínda continúa sobre quen debería recibir o crédito polo seu descubrimento.[222]
Crick presentou en 1957 odogma central da bioloxía molecular,que predicía as relacións entre o ADN, ARN e proteínas, e formulou a "hipótese do adaptador".[223]O mecanismo dereplicaciónsemiconservativo do ADN foi demostrado nos experimentos de 1958 deMeselson e Stahlcon isótopos do nitróxeno.[224]Posteriores traballos de Crick e colaboradores demostraron que ocódigo xenéticoestaba baseado en tripletes de bases non solapados, chamadoscodóns,o que permitiu aHar Gobind Khorana,Robert W. HolleyeMarshall Warren Nirenbergdescifrar o código xenético.[225]Estes descubrimentos representaron o nacemento dabioloxía molecular.[226]
Notas
[editar|editar a fonte]- Todas as referencias en inglés agás cando se indique o contrario
- ↑1,01,1Alberts, Bruce; Alexander Johnson; Julian Lewis; Martin Raff; Keith Roberts; Peter Walters (2002). Garland Science, ed.Molecular Biology of the Cell; Fourth Edition.New York and London.ISBN0-8153-3218-1.
- ↑Butler, John M. (2001)Forensic DNA Typing"Elsevier". pp. 14–15.ISBN 978-0-12-147951-0.
- ↑Mandelkern M, Elias J, Eden D, Crothers D (1981). "The dimensions of DNA in solution".J Mol Biol152(1): 153–61.PMID7338906.
- ↑Gregory S; et al. (2006). "The DNA sequence and biological annotation of human chromosome 1".Nature441(7091): 315–21.PMID16710414.
- ↑5,05,1Watson JD; Crick FHC. A structure for Deoxyribose Nucleic Acid.Nature171(4356):737-738.. (April, 1953)Texto Completo
- ↑Watson J, Crick F (1953)."Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid"(PDF).Nature171(4356): 737–8.PMID13054692.
- ↑Andrew Bates (2005). "DNA structure". En Oxford University Press.DNA topology.ISBN 0-19-850655-4.
- ↑8,08,18,2Berg J., Tymoczko J. and Stryer L. (2002)Biochemistry.W. H. Freeman and CompanyISBN 0-7167-4955-6
- ↑Yakovchuk P, Protozanova E, Frank-Kamenetskii MD (2006)."Base-stacking and base-pairing contributions into thermal stability of the DNA double helix".Nucleic Acids Res.34(2): 564–74.PMC1360284.PMID16449200.doi:10.1093/nar/gkj454.
- ↑Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their ConstituentsIUPAC-IUB Commission on Biochemical Nomenclature (CBN), consultado o 3 de xaneiro de 2006
- ↑11,011,1Ghosh A, Bansal M (2003). "A glossary of DNA structures from A to Z".Acta Crystallogr D Biol Crystallogr59(Pt 4): 620–6.PMID12657780.
- ↑Clark, Jim (2009)."DNA - structure".chemguide.
- ↑NOTA: A pesar do uso tradicional en textos reputados de bioloxía (Ver:Lehninger, Albert L. (1975). Worth Publishers Inc., ed.Biochemistry: the molecular basis of cell structure and function.New York.doi:10.1002/jobm.19770170116.) (Ver:Stryer, Lubert (1988). W. H. Freeman, ed.Biochemistry(3rd ed.). New York.ISBN9780716719205.) de denominar nucleótidos aos que teñen un, dous ou tres fosfatos, para a IUPAC, só son nucleótidos os que teñen un fosfato, e os outros sonnucleósidosdi etrifosfato.(Ver:International Union of Pure and Applied Chemistry (ed.)."Nucleotides".IUPAC Gold Book.doi:10.1351/goldbook.N04255.Consultado o 14-10-2015.).
- ↑14,014,114,2Bruce Alberts; et al. (1986).Biología Molecular de la Célula.Omega. p. 110-115.ISBN84-282-0752-6.
- ↑ Robinson DJ, Fairall L, Huynh VA, Rhodes D. (2006)."EM measurements define the dimensions of the" 30-mm "chromatin fiber: evidence for a compact, interdigitated structure".PNAS103(17): 6506–11.PMC1436021.PMID16617109.doi:10.1073/pnas.0601212103.
- ↑"Phosphdiester bond".School of BioMedical Sciences Wiki.Arquivado dendeo orixinalo 30 de xuño de 2017.Consultado o 14 de outubro de 2015.
- ↑Bruce Alberts; et al.Molecular Biology of the Cell.(4th ed.)..
- ↑Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and CompanyISBN 0-7167-4955-6
- ↑Kiljunen S, Hakala K, Pinta E, Huttunen S, Pluta P, Gador A, Lönnberg H, Skurnik M (2005). "Yersiniophage phiR1-37 is a tailed bacteriophage having a 270 kb DNA genome with thymidine replaced by deoxyuridine".Microbiology151(12): 4093–4102.PMID16339954.doi:10.1099/mic.0.28265-0.
- ↑Uchiyama J, Takemura-Uchiyama I, Sakaguchi Y, Gamoh K, Kato SI, Daibata M, Ujihara T, Misawa N, Matsuzaki S (2014 Mar 6). "Intragenus generalized transduction inStaphylococcusspp. by a novel giant phage. ".ISME J.doi:10.1038/ismej.2014.29.
- ↑21,021,121,221,321,421,5J. M. Macarulla, F. M. Goñi.Biomoléculas. Lecciones de Bioquímica Estructural.Reverté. pp. 119–120, 135.ISBN84-291-7328-5.
- ↑22,022,122,222,322,422,5Harvey Lodish, Arnold Berk, S Lawrence Zipursky, Paul Matsudaira, David Baltimore, and James Darnell (2000).Molecular Cell Biology (online)(4th ed.). New York: W. H. Freeman.ISBN0-7167-3136-3.
- ↑"«Acyclovir»".The American Society of Health-System Pharmacists.Consultado o 15-10-2015.
- ↑"Fluorouracil - Definición no Free Merriam-Webster Dictionary".Consultado o 15-10-2015.
- ↑Simpson L (1998)."A base called J".Proc Natl Acad Sci USA95(5): 2037–2038.Bibcode:1998PNAS...95.2037S.PMC33841.PMID9482833.doi:10.1073/pnas.95.5.2037.
- ↑Borst P, Sabatini R (2008)."Base J: discovery, biosynthesis, and possible functions".Annual review of microbiology62:235–51.PMID18729733.doi:10.1146/annurev.micro.62.081307.162750.
- ↑McNaught, A. D.; Wilkinson, A., eds. (1997).IUPAC Gold Book. Compendium of Chemical Terminology.Ver "attenuation", "extinction" e "absorbance" (2ª ed.). Oxford: Blackwell Scientific Publications.ISBN0-9678550-9-8.doi:10.1351/goldbook.
- ↑M. Mandel and J. Marmur (1968). "Use of Ultraviolet Absorbance-Temperature Profile for Determining the Guanine plus Cytosine Content of DNA".Methods in Enzymology12(2): 198–206.ISBN978-0-12-181856-2.doi:10.1016/0076-6879(67)12133-2.
- ↑Katritzky AR, Elguero J; et al. (1976).The Tautomerism of heterocycles.New York: Academic Press.ISBN0-1202-0651-X.
- ↑"Tautomerization".IUPAC Gold Book.
- ↑"Hidrogen bond".IUPAC Gold book.
- ↑International Human Genome Sequencing Consortium (2004). "Finishing the euchromatic sequence of the human genome".Nature(431 (7011)): 931–45.PMID15496913.
- ↑Dear, Paul H (2006). "Megabase Pair (Mbp)".eLS(Chichester: John Wiley & Sons).doi:10.1038/npg.els.0005710.
- ↑Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H (2000)."Mechanical stability of single DNA molecules".Biophys J78(4): 1997–2007.PMID10733978.
- ↑Ponnuswamy P, Gromiha M (1994). "On the conformational stability of oligonucleotide duplexes and tRNA molecules".J Theor Biol169(4): 419–32.PMID7526075.
- ↑36,036,1Alberts B, Johnson A, Lewis J; et al. (2002).Molecular Biology of the Cell.New York: Garland Science.
- ↑"Erwin Chargaff Papers".Arquivado dendeo orixinalo 15 de maio de 2005.Consultado o 29 de xuño de 2011.
- ↑Chalikian T, Völker J, Plum G, Breslauer K (1999)."A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques".Proc Natl Acad Sci U S A96(14): 7853–8.PMID10393911.
- ↑deHaseth P, Helmann J (1995)."Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA".Mol Microbiol16(5): 817–24.PMID7476180.
- ↑Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J (2004). "Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern".Biochemistry43(51): 15996–6010.PMID15609994.
- ↑41,041,1Evgenia N. Nikolova, Eunae Kim, Abigail A. Wise, Patrick J. O’Brien, Ioan Andricioaei, Hashim M. Al-Hashimi (2011). "Transient Hoogsteen base pairs in canonical duplex DNA".Nature470:498–502.doi:10.1038/nature09775.
- ↑Pinotsis N, Wilmanns M (October 2008)."Protein assemblies with palindromic structure motifs".Cell. Mol. Life Sci.65(19): 2953–6.PMID18791850.doi:10.1007/s00018-008-8265-1.
- ↑"Reverse Watson-Crick base pairs".·DNA Homepage.Consultado o 17-10-2015.
- ↑Hoogsteen K (1963). "The crystal and molecular structure of a hydrogen-bonded complex between 1-methylthymine and 9-methyladenine".Acta Crystallographica16:907–916.doi:10.1107/S0365110X63002437.
- ↑"Hoogsteen base pairing in DNA".Principles of Biochemistry.
- ↑"Hoogsteen and reverse Hoogsteen base pairs".3DNA Homepage.
- ↑Varani G, McClain W (2000)."The G × U wobble base pair. A fundamental building-block of RNA structure crucial to RNA function in diverse biological systems".EMBO Rep1(1): 18–23.PMC1083677.PMID11256617.doi:10.1093/embo-reports/kvd001.
- ↑Ebersberger I, Metzler D, Schwarz C, Pääbo S (June 2002)."Genomewide comparison of DNA sequences between humans and chimpanzees".Am. J. Hum. Genet.70(6): 1490–7.PMC379137.PMID11992255.doi:10.1086/340787.
- ↑49,049,149,249,3Hib, J. & De Robertis, E. D. P. 1998.Fundamentos de biología celular y molecular.El Ateneo, 3ª edición, 416 páxinas.ISBN 950-02-0372-3.ISBN 978-950-02-0372-2
- ↑50,050,150,2De Robertis, E.D.P. 1998. Biología celular y molecular. El Ateneo, 617 páxinas.ISBN 950-02-0364-2.ISBN 978-950-02-0364-7
- ↑51,051,1Dickerson RE, Drew HR, Conner BN, Wing RM, Fratini AV, Kopka ML (1982). "The anatomy of A-, B-, and Z-DNA".Science(216 (4545)): 475–85 @ doi =10.1126/science.7071593.PMID7071593.
- ↑Basu H, Feuerstein B, Zarling D, Shafer R, Marton L (1988). "Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies".J Biomol Struct Dyn6(2): 299–309.PMID2482766.
- ↑Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (1980). "Polymorphism of DNA double helices".J. Mol. Biol.143(1): 49–72.PMID7441761.
- ↑Wahl M, Sundaralingam M (1997)."Crystal structures of A-DNA duplexes".Biopolymers44(1): 45–63.PMID9097733.
- ↑Lu XJ, Shakked Z, Olson WK (2000). "A-form conformational motifs in ligand-bound DNA structures".J. Mol. Biol.300(4): 819–40.PMID10891271.
- ↑Rothenburg S, Koch-Nolte F, Haag F. "DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles".Immunol Rev184:286–98.PMID12086319.
- ↑Oh D, Kim Y, Rich A (2002)."Z-DNA-binding proteins can act as potent effectors of gene expression in vivo".Proc. Natl. Acad. Sci. U.S.A.99(26): 16666–71.PMID12486233.
- ↑Created fromNDB UD0017Arquivado07 de xuño de 2013 enWayback Machine.
- ↑59,059,1Greider C, Blackburn E (1985). "Identification of a specific telomere terminal transferase activity in Tetrahymena extracts".Cell43(2 Pt 1): 405–13.PMID3907856.
- ↑60,060,160,2Nugent C, Lundblad V (1998)."The telomerase reverse transcriptase: components and regulation".Genes Dev12(8): 1073–85.PMID9553037.
- ↑Wright W, Tesmer V, Huffman K, Levene S, Shay J (1997)."Normal human chromosomes have long G-rich telomeric overhangs at one end".Genes Dev11(21): 2801–9.PMID9353250.
- ↑62,062,1Burge S, Parkinson G, Hazel P, Todd A, Neidle S (2006)."Quadruplex DNA: sequence, topology and structure".Nucleic Acids Res34(19): 5402–15.PMID17012276.
- ↑Parkinson G, Lee M, Neidle S (2002). "Crystal structure of parallel quadruplexes from human telomeric DNA".Nature417(6891): 876–80.PMID12050675.
- ↑Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T (1999). "Mammalian telomeres end in a large duplex loop".Cell97(4): 503–14.PMID10338214.
- ↑Created fromPDB 1D65
- ↑66,066,1Watson, J. D.; Baker, T. A.; Bell, S. P.; Gann, A.; Levine, M.; Losick, R (2006).6. Las estructuras del DNA y el RNA.Biología Molecular del Gen(5ª Ed.) ed.) (Madrid: Médica Panamericana).ISBN84-7903-505-6.
- ↑Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R (1980). "Crystal structure analysis of a complete turn of B-DNA".Nature287(5784): 755–8.PMID7432492.
- ↑Pabo C, Sauer R (1984)."Protein-DNA recognition".Annu Rev Biochem53:293–321.PMID6236744.
- ↑Hüttenhofer A, Schattner P, Polacek N (2005)."Non-coding RNAs: hope or hype?".Trends Genet21(5): 289–97.PMID15851066.
- ↑Munroe S (2004). "Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns".J Cell Biochem93(4): 664–71.PMID15389973.
- ↑Makalowska I, Lin C, Makalowski W (2005). "Overlapping genes in vertebrate genomes".Comput Biol Chem29(1): 1–12.PMID15680581.
- ↑Johnson Z, Chisholm S (2004). "Properties of overlapping genes are conserved across microbial genomes".Genome Res14(11): 2268–72.PMID15520290.
- ↑Lamb R, Horvath C (1991). "Diversity of coding strategies in influenza viruses".Trends Genet7(8): 261–6.PMID1771674.
- ↑Benham C, Mielke S (2005). "DNA mechanics".Annu Rev Biomed Eng7:21–53.PMID16004565.
- ↑75,075,1Champoux J (2001)."DNA topoisomerases: structure, function, and mechanism".Annu Rev Biochem70:369–413.PMID11395412.
- ↑76,076,1Wang J (2002). "Cellular roles of DNA topoisomerases: a molecular perspective".Nat Rev Mol Cell Biol3(6): 430–40.PMID12042765.
- ↑Klose R, Bird A (2006). "Genomic DNA methylation: the mark and its mediators".Trends Biochem Sci31(2): 89–97.PMID16403636.doi:10.1016/j.tibs.2005.12.008.
- ↑Bird A (2002). "DNA methylation patterns and epigenetic memory".Genes Dev16(1): 6–21.PMID11782440.doi:10.1101/gad.947102.
- ↑Walsh C, Xu G (2006). "Cytosine methylation and DNA repair".Curr Top Microbiol Immunol301:283–315.PMID16570853.doi:10.1007/3-540-31390-7_11.
- ↑Ratel D, Ravanat J, Berger F, Wion D (2006). "N6-methyladenine: the other methylated base of DNA".Bioessays28(3): 309–15.PMID16479578.doi:10.1002/bies.20342.
- ↑Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P (1993). "beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei".Cell75(6): 1129–36.PMID8261512.doi:10.1016/0092-8674(93)90322-H.
- ↑Creado a partir de:PDB 1JDGArquivado22 de setembro de 2008 enWayback Machine.
- ↑Douki T, Reynaud-Angelin A, Cadet J, Sage E (2003). "Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation".Biochemistry42(30): 9221–6.PMID12885257.doi:10.1021/bi034593c.,
- ↑Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S (1999). "Hydroxyl radicals and DNA base damage".Mutat Res424(1–2): 9–21.PMID10064846.
- ↑Shigenaga M, Gimeno C, Ames B (1989)."Urinary 8-hydroxy-2′-deoxyguanosine as a biological marker ofin vivooxidative DNA damage ".Proc Natl Acad Sci U S A86(24): 9697–701.PMID2602371.doi:10.1073/pnas.86.24.9697.Arquivado dendeo orixinalo 07 de marzo de 2008.Consultado o 01 de xullo de 2011.
- ↑Cathcart R, Schwiers E, Saul R, Ames B (1984)."Thymine glycol and thymidine glycol in human and rat urine: a possible assay for oxidative DNA damage"(PDF).Proc Natl Acad Sci U S A81(18): 5633–7.PMID6592579.doi:10.1073/pnas.81.18.5633.Arquivado dendeo orixinal(PDF)o 25 de xuño de 2008.Consultado o 01 de xullo de 2011.
- ↑Valerie K, Povirk L (2003). "Regulation and mechanisms of mammalian double-strand break repair".Oncogene22(37): 5792–812.PMID12947387.doi:10.1038/sj.onc.1206679.
- ↑Ferguson L, Denny W (1991). "The genetic toxicology of acridines".Mutat Res258(2): 123–60.PMID1881402.
- ↑Jeffrey A (1985)."DNA modification by chemical carcinogens".Pharmacol Ther28(2): 237–72.PMID3936066.doi:10.1016/0163-7258(85)90013-0.
- ↑Stephens T, Bunde C, Fillmore B (2000). "Mechanism of action in thalidomide teratogenesis".Biochem Pharmacol59(12): 1489–99.PMID10799645.doi:10.1016/S0006-2952(99)00388-3.
- ↑Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (2001). "Intercalators as anticancer drugs".Curr Pharm Des7(17): 1745–80.PMID11562309.doi:10.2174/1381612013397113.
- ↑Roos, WP; Kaina, B (setembro 2006). "DNA damage-induced cell death by apoptosis.".Trends Mol Med.(12(9)): 440–50.PMID16899408.
- ↑Ridley, M. (2006).Genome.New York, NY: Harper Perennial.ISBN0-06-019497-9.
- ↑Thanbichler M, Wang S, Shapiro L (2005). "The bacterial nucleoid: a highly organized and dynamic structure".J Cell Biochem96(3): 506–21.PMID15988757.doi:10.1002/jcb.20519.
- ↑Creado a partir de:PDB 1MSW
- ↑Pennacchio, L. A.; Bickmore, W.; Dean, A.; Nobrega, M. A.; Bejerano, G. (2013). "Enhancers: Five essential questions".Nature Reviews Genetics14(4): 288–95.PMID23503198.doi:10.1038/nrg3458.
- ↑Crick, F (1970)."Central dogma of molecular biology."(PDF).Nature227(5258): 561–3.Bibcode:1970Natur.227..561C.PMID4913914.doi:10.1038/227561a0.
- ↑Wolfsberg T, McEntyre J, Schuler G (2001). "Guide to the draft human genome".Nature409(6822): 824–6.PMID11236998.doi:10.1038/35057000.
- ↑TheENCODEProject Consortium (2007). "Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project".Nature447(7146): 799–816.doi:10.1038/nature05874.
- ↑Gregory T (2005)."The C-value enigma in plants and animals: a review of parallels and an appeal for partnership".Ann Bot (Lond)95(1): 133–46.PMID15596463.doi:10.1093/aob/mci009.
- ↑Yale University."Yale Researchers Find “Junk DNA” May Have Triggered Key Evolutionary Changes in Human Thumb and Foot ".Arquivado dendeo orixinalo 12 de setembro de 2008.Consultado o 02 de xullo de 2011.Arquivado12 de setembro de 2008 enWayback Machine.
- ↑Pidoux AL, Allshire RC (2005)."The role of heterochromatin in centromere function".Philosophical Transactions of the Royal Society B360(1455): 569–79.PMC1569473.PMID15905142.doi:10.1098/rstb.2004.1611.
- ↑Harrison PM, Hegyi H, Balasubramanian S, Luscombe NM, Bertone P, Echols N, Johnson T, Gerstein M (2002)."Molecular Fossils in the Human Genome: Identification and Analysis of the Pseudogenes in Chromosomes 21 and 22".Genome Res12(2): 272–80.PMC155275.PMID11827946.doi:10.1101/gr.207102.
- ↑Harrison PM, Gerstein M (2002). "Studying genomes through the aeons: protein families, pseudogenes and proteome evolution".J Mol Biol318(5): 1155–74.PMID12083509.doi:10.1016/S0022-2836(02)00109-2.
- ↑Wang, W; Li, H; Chen, Z (2014). "Analysis of plastid and nuclear DNA data in plant phylogenetics-evaluation and improvement".Sci China Life Sci57(3): 280–6.PMID24554473.doi:10.1007/s11427-014-4620-7.
- ↑EP Salomon, CA Villee, PW Davis.Biología.México: Interamiericana. pp. 345–349.ISBN968-25-1225-5.
- ↑"Genetic Code".Brooklyn.cuny.edu.Consultado o 16 de outubro de 2015.
- ↑Allison, Lizabeth A (2007).Fundamental Molecular Biology.Blackwell Publishing. pp.112.ISBN978-1-4051-0379-4.
- ↑Albà M (2001)."Replicative DNA polymerases".Genome Biol2(1): reviews3002.1–reviews3002.4.PMC150442.PMID11178285.doi:10.1186/gb-2001-2-1-reviews3002.
- ↑Sandman K, Pereira S, Reeve J (1998)."Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome".Cell Mol Life Sci54(12): 1350–64.PMID9893710.doi:10.1007/s000180050259.
- ↑Dame RT (2005). "The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin".Mol. Microbiol.56(4): 858–70.PMID15853876.doi:10.1111/j.1365-2958.2005.04598.x.
- ↑Luger K, Mäder A, Richmond R, Sargent D, Richmond T (1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution".Nature389(6648): 251–60.PMID9305837.doi:10.1038/38444.
- ↑Jenuwein T, Allis C (2001). "Translating the histone code".Science293(5532): 1074–80.PMID11498575.doi:10.1126/science.1063127.
- ↑Ito T. "Nucleosome assembly and remodelling".Curr Top Microbiol Immunol274:1–22.PMID12596902.
- ↑Thomas J (2001). "HMG1 and 2: architectural DNA-binding proteins".Biochem Soc Trans29(Pt 4): 395–401.PMID11497996.doi:10.1042/BST0290395.
- ↑Grosschedl R, Giese K, Pagel J (1994)."HMG domain proteins: architectural elements in the assembly of nucleoprotein structures".Trends Genet10(3): 94–100.PMID8178371.doi:10.1016/0168-9525(94)90232-1.
- ↑Maeshima K, Laemmli UK. (2003). "A Two-Step Scaffolding Model for Mitotic Chromosome Assembly".Developmental Cell4.467-480.[1]
- ↑Tavormina PA, Côme MG, Hudson JR, Mo YY, Beck WT, Gorbsky GJ. (2002). "Rapid exchange of mammalian topoisomerase II alpha at kinetochores and chromosome arms in mitosis".J Cell Biol.158(1). 23-9.[2]
- ↑Iftode C, Daniely Y, Borowiec J (1999). "Replication protein A (RPA): the eukaryotic SSB".Crit Rev Biochem Mol Biol34(3): 141–80.PMID10473346.doi:10.1080/10409239991209255.
- ↑Firdos Alam Khan (2011).Biotechnology Fundamentals.CRC Press. p. 40.ISBN9781439897126.
- ↑Creado a partir dePDB1LMB.
- ↑Pabo C, Sauer R (1984)."Protein-DNA recognition".Annu Rev Biochem53:293–321.PMID6236744.doi:10.1146/annurev.bi.53.070184.001453.
- ↑Latchman DS (1997)."Transcription factors: an overview".Int. J. Biochem. Cell Biol.29(12): 1305–12.PMID9570129.doi:10.1016/S1357-2725(97)00085-X.
- ↑Myers L, Kornberg R (2000)."Mediator of transcriptional regulation".Annu Rev Biochem69:729–49.PMID10966474.doi:10.1146/annurev.biochem.69.1.729.
- ↑Spiegelman B, Heinrich R (2004). "Biological control through regulated transcriptional coactivators".Cell119(2): 157–67.PMID15479634.doi:10.1016/j.cell.2004.09.037.
- ↑Li Z, Van Calcar S, Qu C, Cavenee W, Zhang M, Ren B (2003)."A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells".Proc Natl Acad Sci U S A100(14): 8164–9.PMID12808131.doi:10.1073/pnas.1332764100.
- ↑Creado a partir dePDB 1RVA
- ↑"Nuclease".Encyclopedia Britannica.
- ↑Lehninger A. L. (1980).Bioquímica.Omega. pp. 329–330.ISBN84-282-0211-7.
- ↑Bickle TA, Krüger DH (1993)."Biology of DNA restriction".Microbiol Rev57(2): 434–50.PMC372918.PMID8336674.
- ↑Bickle T, Krüger D (1993)."Biology of DNA restriction".Microbiol Rev57(2): 434–50.PMID8336674.
- ↑David M. Glover; B. D. Hames, eds. (1995).DNA Cloning: Complex genomes.IRL Press.ISBN9780199634835.
- ↑Jay Aronson (2007).Genetic Witness: Science, Law, and Controversy in the Making of DNA Profiling.Rutgers University Press.ISBN9780813543833.
- ↑134,0134,1Doherty A, Suh S (2000)."Structural and mechanistic conservation in DNA ligases.".Nucleic Acids Res28(21): 4051–8.PMID11058099.doi:10.1093/nar/28.21.4051.
- ↑Schoeffler A, Berger J (2005). "Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism".Biochem Soc Trans33(Pt 6): 1465–70.PMID16246147.doi:10.1042/BST20051465.
- ↑Tuteja N, Tuteja R (2004). "Unraveling DNA helicases. Motif, structure, mechanism and function".Eur J Biochem271(10): 1849–63.PMID15128295.doi:10.1111/j.1432-1033.2004.04094.x.
- ↑Patel, S. S.; Donmez, I (2006). "Mechanisms of Helicases".Journal of Biological Chemistry281(27): 18265–18268.ISSN0021-9258.PMID16670085.doi:10.1074/jbc.R600008200.
- ↑Joyce C, Steitz T (1995)."Polymerase structures and function: variations on a theme?".J Bacteriol177(22): 6321–9.PMID7592405.
- ↑Hubscher U, Maga G, Spadari S (2002)."Eukaryotic DNA polymerases".Annu Rev Biochem71:133–63.PMID12045093.doi:10.1146/annurev.biochem.71.090501.150041.
- ↑Johnson A, O'Donnell M (2005)."Cellular DNA replicases: components and dynamics at the replication fork".Annu Rev Biochem74:283–315.PMID15952889.doi:10.1146/annurev.biochem.73.011303.073859.
- ↑Tarrago-Litvak L, Andréola M, Nevinsky G, Sarih-Cottin L, Litvak S (1994)."The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention".FASEB J8(8): 497–503.PMID7514143.
- ↑Martinez E (2002)."Multi-protein complexes in eukaryotic gene transcription".Plant Mol Biol50(6): 925–47.PMID12516863.doi:10.1023/A:1021258713850.
- ↑Creado a partir dePDB 1M6G
- ↑Cremer T, Cremer C (2001). "Chromosome territories, nuclear architecture and gene regulation in mammalian cells".Nat Rev Genet2(4): 292–301.PMID11283701.doi:10.1038/35066075.
- ↑Creighton, H. B.; McClintock, B (1931)."A Correlation of Cytological and Genetical Crossing-Over in Zea Mays".Proceedings of the National Academy of Sciences of the United States of America17(8): 492–497.PMC1076098.
- ↑Pál C, Papp B, Lercher M (2006). "An integrated view of protein evolution".Nat Rev Genet7(5): 337–48.PMID16619049.doi:10.1038/nrg1838.
- ↑O'Driscoll M, Jeggo P (2006). "The role of double-strand break repair - insights from human genetics".Nat Rev Genet7(1): 45–54.PMID16369571.doi:10.1038/nrg1746.
- ↑Vispé S, Defais M (1997)."Mammalian Rad51 protein: a RecA homologue with pleiotropic functions".Biochimie79(9-10): 587–92.PMID9466696.doi:10.1016/S0300-9084(97)82007-X.
- ↑Neale MJ, Keeney S (2006). "Clarifying the mechanics of DNA strand exchange in meiotic recombination".Nature442(7099): 153–8.PMID16838012.doi:10.1038/nature04885.
- ↑Dickman M, Ingleston S, Sedelnikova S, Rafferty J, Lloyd R, Grasby J, Hornby D (2002). "The RuvABC resolvasome".Eur J Biochem269(22): 5492–501.PMID12423347.doi:10.1046/j.1432-1033.2002.03250.x.
- ↑Joyce G (2002). "The antiquity of RNA-based evolution".Nature418(6894): 214–21.PMID12110897.doi:10.1038/418214a.
- ↑Orgel L."Prebiotic chemistry and the origin of the RNA world"(PDF).Crit Rev Biochem Mol Biol39(2): 99–123.PMID15217990.doi:10.1080/10409230490460765.
- ↑Davenport R (2001). "Ribozymes. Making copies in the RNA world".Science292(5520): 1278.PMID11360970.doi:10.1126/science.292.5520.1278a.
- ↑Szathmáry E (1992)."What is the optimum size for the genetic alphabet?"(PDF).Proc Natl Acad Sci U S A89(7): 2614–8.PMID1372984.doi:10.1073/pnas.89.7.2614.Arquivado dendeo orixinal(PDF)o 25 de xuño de 2008.Consultado o 05 de xullo de 2011.
- ↑Lindahl T (1993). "Instability and decay of the primary structure of DNA".Nature362(6422): 709–15.PMID8469282.doi:10.1038/362709a0.
- ↑Vreeland R, Rosenzweig W, Powers D (2000). "Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal".Nature407(6806): 897–900.PMID11057666.doi:10.1038/35038060.
- ↑Hebsgaard M, Phillips M, Willerslev E (2005)."Geologically ancient DNA: fact or artefact?".Trends Microbiol13(5): 212–20.PMID15866038.doi:10.1016/j.tim.2005.03.010.
- ↑Nickle D, Learn G, Rain M, Mullins J, Mittler J (2002)."Curiously modern DNA for a" 250 million-year-old "bacterium".J Mol Evol54(1): 134–7.PMID11734907.doi:10.1007/s00239-001-0025-x.
- ↑Birnbaum D, Coulier F, Pébusque MJ, Pontarotti P. (2000). ""Paleogenomics ": looking in the past to the future".J Exp Zool.288((1):). 21-2.[3]
- ↑Blanchette M, Green ED, Miller W, Haussler D. (2004). "Reconstructing large regions of an ancestral mammalian genome in silico".Genome Res.14((12):). 2412-23.Erratum in: Genome Res. 2005 Mar;15(3):451.[4]
- ↑Gaucher EA, Thomson JM, Burgan MF, Benner SA. (2003). "Inferring the palaeoenvironment of ancient bacteria on the basis of resurrected protein".Nature425((6955):). 285-8.[5]
- ↑Thornton JW. (2004). "Resurrecting ancient genes: experimental analysis of extinct molecules".Nat Rev Genet.5((5):). 366-75.[6]
- ↑Benner SA, Caraco MD, Thomson JM, Gaucher EA. (2002). "Planetary biology--paleontological, geological, and molecular histories of life".Science296((5569):). 864-8.[7]
- ↑Brenner SA, Carrigan MA, Ricardo A, Frye F. (2006). "Setting the stage: the history, chemistry and geobiology behind RNA". En Cold Spring Harbor Laboratory Press.The RNA World, 3rd Ed.ISBN 0-87969-739-3.[8]Arquivado10 de agosto de 2011 enWayback Machine.
- ↑165,0165,1165,2Griffiths, J.F. A.; et al. (2002). McGraw-Hill Interamericana, ed.Genética.ISBN 84-486-0368-0.
- ↑166,0166,1Watson, J, D.; Baker, T. A.; Bell, S. P.; Gann, A.; Levine, M.; Losick, R (2004). Benjamin Cummings, ed.Molecular Biology of the Gene(5ª ed.). San Francisco.ISBN0-321-22368-3.
- ↑Pettersson E, Lundeberg J, Ahmadian A (2009). "Generations of sequencing technologies".Genomics93(2): 105–11.PMID18992322.doi:10.1016/j.ygeno.2008.10.003.
- ↑Sanger F, Coulson AR (1975). "A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase".J. Mol. Biol.94(3): 441–8.PMID1100841.doi:10.1016/0022-2836(75)90213-2.
- ↑Sanger F, Coulson AR. "A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase".J Mol Biol. data=25 de maio de 1975(94(3)): 441–448.
- ↑Sanger F, Nicklen s, y Coulson AR (decembro de 1977). "DNA sequencing with chain-terminating inhibitors".Proc Natl Acad Sci U S A(74(12)): 5463–5467.
- ↑Bartlett, John M. S.; Stirling, David (2003). Bartlett, John M. S.; Stirling, David, eds.A Short History of the Polymerase Chain Reaction.Methods in Molecular Biology™(eninglés).Totowa, NJ: Humana Press. pp. 3–6.ISBN9781592593842.doi:10.1385/1-59259-384-4:3.
- ↑Prof. Juan Manuel González Mañas."Transferencia blotting)".Curso de Biomoléculas (Universidad del País Vasco).
- ↑Vera-Cabrera, L., Rendon, A., Diaz-Rodriguez, M., Handzel, V., & Laszlo, A (1999)."Dot Blot Assay for Detection of Antidiacyltrehalose Antibodies in Tuberculous Patients".Clinical and Diagnostic Laboratory Immunology6(5): 686–689.PMC95755.
- ↑Southern, E.M. (1975): "Detection of specific sequences among DNA fragments separated by gel electrophoresis",J Mol Biol.,98:503-517.PMID 1195397
- ↑Alwine JC, Kemp DJ, Stark GR (1977). "Method for detection of specific RNAs in agarose gels by transfer to diazobenzyloxymethyl-paper and hybridization with DNA probes".Proc. Natl. Acad. Sci. U.S.A.74(12): 5350–4.PMID414220.doi:10.1073/pnas.74.12.5350.
- ↑W. Neal Burnette (1981). Academic Press, ed."'Western blotting': electrophoretic transfer of proteins from sodium dodecyl sulfate — polyacrylamide gels to unmodified nitrocellulose and radiographic detection with antibody and radioiodinated protein A ".Analytical Biochemistry(United States)112(2): 195–203.ISSN0003-2697.PMID6266278.Arquivado dendeo orixinalo 14-05-2008.Consultado o 3-4-2008.
- ↑"DNA Microarray Technology".NIH - National Human Genome Research Institute.
- ↑"DNA microarray (animación)".Learn.Genetics - University of Utah.
- ↑Miller WL. (1979). "Use of recombinant DNA technology for the production of polypeptides".Adv Exp Med Biol.118:153–74.[9]
- ↑Leader B, Baca QJ, Golan DE. (2008). "Protein therapeutics: a summary and pharmacological classification".Nat Rev Drug Discov.7((1)). 21-39.[10]
- ↑Dingermann T. (2008). "Recombinant therapeutic proteins: production platforms and challenges".Biotechnol J.3((1)). 90-7.[11]
- ↑Voigt K, Izsvák Z, Ivics Z. (2008). "Targeted gene insertion for molecular medicine".J Mol Med.Jul 8. (Epub ahead of print).[12]Arquivado24 de setembro de 2019 enWayback Machine.
- ↑Houdebine L (2007). "Transgenic animal models in biomedical research".Methods Mol Biol360:163–202.PMID17172731.[13]
- ↑Soler E, Thépot D, Rival-Gervier S, Jolivet G, Houdebine LM. (2006 publicación = Reprod Nutr Dev.). "Preparation of recombinant proteins in milk to improve human and animal health"46((5)). 579-88.[14]Arquivado03 de xullo de 2009 enWayback Machine.
- ↑Chávez A, Muñoz de Chávez M. (2003). "Nutrigenomics in public health nutrition: short-term perspectives".Eur J Clin Nutr.57(Suppl 1). S97-100.[15]
- ↑Vasil IK. (2007). "Molecular genetic improvement of cereals: transgenic wheat (Triticum aestivum L.)".Plant Cell Rep.26((8)). 1133-54.[16]Arquivado24 de setembro de 2019 enWayback Machine.
- ↑Daniell H, Dhingra A (2002). "Multigene engineering: dawn of an exciting new era in biotechnology".Curr Opin Biotechnol13(2): 136–41.PMID11950565.doi:10.1016/S0958-1669(02)00297-5.
- ↑Job D (2002). "Plant biotechnology in agriculture".Biochimie84(11): 1105–10.PMID12595138.doi:10.1016/S0300-9084(02)00013-5.
- ↑Collins A, Morton N (1994)."Likelihood ratios for DNA identification"(PDF).Proc Natl Acad Sci USA91(13): 6007–11.PMID8016106.doi:10.1073/pnas.91.13.6007.Arquivado dendeo orixinal(PDF)o 27 de novembro de 2007.Consultado o 07 de xullo de 2011.
- ↑Weir B, Triggs C, Starling L, Stowell L, Walsh K, Buckleton J (1997)."Interpreting DNA mixtures".J Forensic Sci42(2): 213–22.PMID9068179.
- ↑Jeffreys A, Wilson V, Thein S (1985). "Individual-specific 'fingerprints' of human DNA".Nature316(6023): 76–9.PMID2989708.doi:10.1038/316076a0.
- ↑Colin Pitchfork — first murder conviction on DNA evidence also clears the prime suspectArquivado14 de decembro de 2006 enWayback Machine.Forensic Science Service Accessed 23 Dec 2006
- ↑National Institute of Justice, ed. (setembro de 2006)."DNA Identification in Mass Fatality Incidents".Arquivado dendeo orixinalo 12 de novembro de 2006.Consultado o 07 de xullo de 2011.Arquivado25 de febreiro de 2012 enWayback Machine.
- ↑Bhattacharya, Shaoni."Killer convicted thanks to relative's DNA".Arquivado12 de febreiro de 2007 enWayback Machine.newscientist.com(20 abril 2004). Consultado o 22 de decembro de 2006
- ↑Baldi, Pierre. Brunak, Soren (2001). MIT Press, ed.Bioinformatics: The Machine Learning Approach.ISBN978-0-262-02506-5.
- ↑Gusfield, Dan.Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology.Cambridge University Press, 15 January 1997.ISBN 978-0-521-58519-4.
- ↑Sjölander K (2004)."Phylogenomic inference of protein molecular function: advances and challenges".Bioinformatics20(2): 170–9.PMID14734307.doi:10.1093/bioinformatics/bth021.
- ↑Mount, DM (2004). Cold Spring Harbor Laboratory Press, ed.Bioinformatics: Sequence and Genome Analysis(2 ed.). Cold Spring Harbor, NY.ISBN0879697121.
- ↑Yin P, Hariadi RF, Sahu S, Choi HMT, Park SH, LaBean T, Reif JH. (2008). "Programming DNA Tube Circumferences".Science321((5890)).doi:10.1126/science.1157312.824 - 826.[17]
- ↑Andersen ES, Dong M, Nielsen MM, Jahn K, Subramani R, Mamdouh W, Golas MM, Sander B, Stark H, Oliveira CL, Pedersen JS, Birkedal V, Besenbacher F, Gothelf KV, Kjems J (2009). "Self-assembly of a nanoscale DNA box with a controllable lid". Nature 459 (7243): 73–6. Bibcode:2009Natur.459...73A. doi:10.1038/nature07971.PMID 19424153.
- ↑Ishitsuka Y, Ha T (2009). "DNA nanotechnology: a nanomachine goes live".Nat Nanotechnol4(5): 281–2.Bibcode:2009NatNa...4..281I.PMID19421208.doi:10.1038/nnano.2009.101.
- ↑Aldaye FA, Palmer AL, Sleiman HF (2008). "Assembling materials with DNA as the guide".Science321(5897): 1795–9.Bibcode:2008Sci...321.1795A.PMID18818351.doi:10.1126/science.1154533.
- ↑Wray G (2002)."Dating branches on the tree of life using DNA".Genome Biol3(1): REVIEWS0001.PMID11806830.doi:10.1046/j.1525-142X.1999.99010.x.
- ↑"Utilizing Phylogenetic Information".CIPRES.Arquivado dendeo orixinalo 17 de xullo de 2016.Consultado o 28 de outubro de 2015.Arquivado17 de xullo de 2016 enWayback Machine.
- ↑Goldman N, Bertone P, Chen S, Dessimoz C, LeProust EM, Sipos B, Birney E (23 January 2013)."Towards practical, high-capacity, low-maintenance information storage in synthesized DNA".Nature494(7435): 77–80.Bibcode:2013Natur.494...77G.PMC3672958.PMID23354052.doi:10.1038/nature11875.
- ↑Naik, Gautam (24 de xaneiro de 2013)."Storing Digital Data in DNA".Wall Street Journal.Consultado o 19-10-2015.
- ↑Dahm R (2008). "Discovering DNA: Friedrich Miescher and the early years of nucleic acid research".Hum. Genet.122(6): 565–81.PMID17901982.doi:10.1007/s00439-007-0433-0.
- ↑Levene P, (1 de decembro de 1919)."The structure of yeast nucleic acid".J Biol Chem40(2): 415–24. Arquivado dendeo orixinalo 29 de xuño de 2009.Consultado o 09 de xullo de 2011.Arquivado29 de xuño de 2009 enWayback Machine.
- ↑Astbury W, (1947). "Nucleic acid".Symp. SOC. Exp. Bbl1(66).
- ↑Valery N. Soyfer. The consequences of political dictatorship for Russian science.Nature Reviews Genetics2:723-729 (2001)
- ↑Lorenz MG, Wackernagel W (1 de setembro de 1994)."Bacterial gene transfer by natural genetic transformation in the environment".Microbiol. Rev.58(3): 563–602.PMC372978.PMID7968924.
- ↑Avery O, MacLeod C, McCarty M (1944)."Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Inductions of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III".J Exp Med79(2): 137–158.PMC2135445.PMID19871359.doi:10.1084/jem.79.2.137.
- ↑Hershey A, Chase M (1952)."Independent functions of viral protein and nucleic acid in growth of bacteriophage"(PDF).J Gen Physiol36(1): 39–56.PMC2147348.PMID12981234.doi:10.1085/jgp.36.1.39.
- ↑Watson J.D. and Crick F.H.C. (1953)."A Structure for Deoxyribose Nucleic Acid"(PDF).Nature171(4356): 737–738.Bibcode:1953Natur.171..737W.PMID13054692.doi:10.1038/171737a0.Consultado o 4 de maio de 2009.
- ↑O patrón de raios X do ADN Bá dereita desta imaxe ligadaArquivado10 de xullo de 2009 enWayback Machine.foi obtida porRosalind Frankline Raymond Gosling en maio de 1952 a altos niveis de resolución do ADN e foi etiquetada como "Foto 51"
- ↑Burton E. Tropp (2012).Principles of Molecular Biology.Jones & Bartlett Publishers. p. 15.ISBN9781449647919.
- ↑Nature ArchivesDouble Helix of DNA: 50 Years
- ↑Franklin, Rosalind and Gosling, Raymond (1953)."Molecular Configuration in Sodium Thymonucleate. Franklin R. and Gosling R.G"(PDF).Nature171(4356): 740–1.Bibcode:1953Natur.171..740F.PMID13054694.doi:10.1038/171740a0.
- ↑Osulibrary.oregonstate.edu (ed.)."Original X-ray diffraction image".Arquivado dendeo orixinalo 30-01-2009.Consultado o =06-01-2011.
- ↑Wilkins M.H.F., A.R. Stokes A.R. & Wilson, H.R. (1953)."Molecular Structure of Deoxypentose Nucleic Acids"(PDF).Nature171(4356): 738–740.Bibcode:1953Natur.171..738W.PMID13054693.doi:10.1038/171738a0.
- ↑The Nobel Prize in Physiology or Medicine 1962Nobelprize.org Acceso o 22 de decembro de 2006
- ↑Brenda Maddox (23 de xaneiro de 2003)."The double helix and the 'wronged heroine'"(PDF).Nature421(6921): 407–408.PMID12540909.doi:10.1038/nature01399.Arquivado dendeo orixinal(PDF)o 17 de outubro de 2016.Consultado o 09 de xullo de 2011.Arquivado17 de outubro de 2016 enWayback Machine.
- ↑Crick, F.H.C.On degenerate templates and the adaptor hypothesis (PDF).Arquivado01 de outubro de 2008 enWayback Machine.genome.wellcome.ac.uk (Lecture, 1955). Consultado o 22 de decembro de 2006.
- ↑Meselson M, Stahl F (1958)."The replication of DNA inEscherichia coli".Proc Natl Acad Sci USA44(7): 671–82.PMC528642.PMID16590258.doi:10.1073/pnas.44.7.671.
- ↑The Nobel Prize in Physiology or Medicine 1968Nobelprize.org Acceso o 22 de decembro de 2006
- ↑Ingram, Vernon M. (17 de outubro de 2002). "The birth of molecular biology".Nature(419): 669–670.doi:10.1038/419669a.
Véxase tamén
[editar|editar a fonte]| Wikimedia Commonsten máis contidos multimedia na categoría: Ácido desoxirribonucleico |
Bibliografía
[editar|editar a fonte]- Berry, Andrew; Watson, James. (2003). Alfred A. Knopf, ed.DNA: the secret of life.Nova York.ISBN0-375-41546-7.
- Calladine, Chris R.; Drew, Horace R.; Luisi, Ben F. and Travers, Andrew A. (2003). Elsevier Academic Press, ed.Understanding DNA: the molecule & how it works.Ámsterdam.ISBN0-12-155089-3.
- Dennis, Carina; Julie Clayton (2003). Palgrave Macmillan, ed.50 years of DNA.Basingstoke.ISBN1-4039-1479-6.
- Judson, Horace F (1979). Touchstone Books, ed.The Eighth Day of Creation: Makers of the Revolution in Biology(2.ª edición ed.).ISBN0-671-22540-5.
- Olby, Robert C. (1994). Dover Publications, ed.The path to the double helix: the discovery of DNA.Nova York.ISBN0-486-68117-3.,(primeira publicación en 1974 por MacMillan, revisado definitivamente en 1994)
- Micklas, David (2003). Cold Spring Harbor Press, ed.DNA Science: A First Course.ISBN978-0-87969-636-8.
- Ridley, Matt (2006). Eminent Lives, Atlas Books, ed.Francis Crick: discoverer of the genetic code.Ashland, OH.ISBN0-06-082333-X.
- Olby, Robert C. (2009). Cold Spring Harbor Laboratory Press, ed.Francis Crick: A Biography.Plainview, N.Y.ISBN0-87969-798-9.
- Rosenfeld, Israel (2010). Columbia University Press, ed.DNA: A Graphic Guide to the Molecule that Shook the World.ISBN978-0-231-14271-7.
- Schultz, Mark e Zander Cannon (2009). Hill and Wang, ed.The Stuff of Life: A Graphic Guide to Genetics and DNA.ISBN0-8090-8947-5.
- Stent, Gunther Siegmund; Watson, James. (1980). Norton, ed.The double helix: a personal account of the discovery of the structure of DNA.Nova York.ISBN0-393-95075-1.
- Watson, James (2004). Random House, ed.DNA: The Secret of Life.ISBN978-0-09-945184-6.
- Wilkins, Maurice (2003). University Press, ed.The third man of the double helix the autobiography of Maurice Wilkins.Cambridge, Eng.ISBN0-19-860665-6.
Outros artigos
[editar|editar a fonte]- ADN mitocondrial
- ADN cloroplástico
- Termodinámica dos ácidos nucleicos
- Rosalind Franklin
- James Watson
- Francis Crick
- Aduto do ADN
- Experimento de Meselson e Stahl
