Transcriptase inversa
| Transcriptase inversa (ADN polimerase ARN dependente) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| Identificadores | |||||||||
| Símbolo | RVT_1 | ||||||||
| Pfam | PF00078 | ||||||||
| Pfamclan | CL0027 | ||||||||
| InterPro | IPR000477 | ||||||||
| PROSITE | PS50878 | ||||||||
| SCOPe | 1hmv/SUPFAM | ||||||||
| CDD | cd00304 | ||||||||
| |||||||||
| ADN polimerase ARN dirixida | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Número EC | 2.7.7.49 | ||||||||
| Número CAS | 9068-38-6 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| EstruturasPDB | RCSB PDBPDBePDBjPDBsum | ||||||||
| Gene Ontology | AmiGO/EGO | ||||||||
| |||||||||
Atranscriptase inversa,[2][3]reversotranscriptaseouretrotranscriptase(RT) é unencimautilizado para xerar unha copia deADN complementario(ADNc) a partir dun molde deARN,proceso chamadotranscrición[4]inversa,reversotranscriciónouretrotranscrición.A transcrición inversa (ARN→ADN) é o proceso inverso datranscrición(ADN→ARN). A transcriptase inversa é necesaria para a replicación dosretrovirus(por exemplo oVIH), e os inhibidores da transcriptase inversa sondrogas antirretroviraismoi utilizadas no tratamento doVIH/SIDA.Noseucariotasexisten tamén encimas con actividade de transcriptase inversa que interveñen na replicación dos extremos doscromosomas(telomerase) e dalgúns elementos xenéticos móbiles (retrotransposóns). Tamén se encontrou actividade de transcriptase inversa nas bacterias nos chamadosRetron msr RNAs.
A transcriptase inversa retroviral presenta tres actividades bioquímicas secuenciais:
- ADN polimerase ARN dependente. A patir dun molde de ARN fai unha copiacomplementariade ADN.
- Ribonuclease H.Degrada ARN.
- ADN polimerase ADN dependente. A partir dun molde de ADN fai unha copia complementaria tamén de ADN.
Estas tres actividades úsanas os retrovirus para converter o seu ARN monocatenario xenómico en ADN bicatenario, que se pode integrar no xenoma do hóspede, xerando potencialmente unha infección de longa duración difícil de erradicar. A mesma secuencia de reaccións é moi usada no laboratorio para converter ARN en ADN para usalo naclonación molecular,secuenciación do ARN,reacción en cadea da polimerase(PCR), ouanálise xenómica.
Transcriptases inversas que foron ben estudadas son:
- Transcriptase inversa doVIH-1(virus da inmunodeficiencia humana 1) (PDB - 1HMV).
- Transcriptase inversa do M-MLV (virus da leucemia murina|virus da leucemia murina Moloney.
- Transcriptase inversa do AMV (virus da mieloblastose animal).
- Transcriptase inversa datelomerase(mantén ostelómerosdos cromosomas eucarióticos).
Historia
[editar|editar a fonte]A transcriptase inversa descubriunaHoward Teminna Universidade de Wisconsin–Madison en virións dovirus do sarcoma de Rous(RSV),[5]e illouna independentementeDavid Baltimoreen 1970 noMITa partir de dous virus tumorais de ARN:R-MLVe oRSV.[6]Por estes descubrimentos compartiron ambos os dous oPremio Nobel de Medicinade 1975 (xunto conRenato Dulbecco).
A idea da reversotranscrición foi ao principio impopular entre os científicos porque contradicía odogma central da bioloxía molecular,que establecía que o ADN se transcrbía a ARN e este se traducía a proteínas. Porén, en 1970 cando os científicosHoward TemineDavid Baltimoredescubriron o encima responsable da reversotranscrición, a posibilidade de que a información xenética puidese fluír desta maneira foi finalmente aceptada.[7]
Función nos virus
[editar|editar a fonte]O encima está codificado nos virus que realizan a reversotranscrición, que utilizan o encima durante a súa replicación. Os virus de ARN que fan a reversotranscrición, como osretrovirus,usan o encima para reversotranscribir o seuxenomade ARN a ADN, que é despois integrado no xenoma da súa célulahóspedee replicado ao replicarse este. O VIH é outro virus de ARN que infecta aos humanos grazas ao uso deste encima, e sen el o xenoma viral non podería incorporarse á célula hóspede e non se podería replicar. Existen tamén virus de ADN que fan a reversotranscrición, como oshepadnavirus,nos que o ARN pode servir como molde na ensamblaxe, e para facer cadeas de ADN.
Proceso da reversotranscrición
[editar|editar a fonte]A transcriptase inversa realiza a reversotranscrición ou transcrición inversa, que consite en formar ADN monocatenario a partir dun molde de ARN.
OsRetroviridaeademais poden facer despois a ese ADN bicatenario. Nas especies de virus que teñen transcriptase inversa pero que carece da actividade de ADN polimerase ADN dependente, a formación de ADN bicatenario pode realizala aADN polimerase δcodificada no xenoma do hóspede, que tomará o híbrido de ADN-ARN viral comoprimer(cebador) e sintetizará un ADN de dobre cadea por un mecanismo similar ao daeliminación do primer,no que o ADN acabado de sintetizar despraza o molde de ARN orixinal.
O proceso da reversotranscrición tende a cometer moitos erros de copia e durante esta fase poden orixinarse moitasmutacións.Tales mutacións poden dar lugar aresistencia ás drogas antirretrovirais.
Transcrición inversa retroviral
[editar|editar a fonte]Osretrovirus,tamén chamados virusssRNA-RTde clase VI, son virus de ARN que fan a transcrición inversa cun intermediato de ADN. Os seus xenomas constan de dúas moléculas de ARN monocatenario (ssRNA) desentido positivoque posúen extremos5' cape3' poliA.Exemplos de retrovirus son ovirus da inmunodeficiencia humana(HIV) e ovirus T linfotrópico humano(HTLV). A creación de ADN de dobre cadea ten lugar nocitosol.[8]
Rexións xénicas no ARN viral
[editar|editar a fonte]Para entendermos mellor as fases que debe seguir o proceso de reversotranscrición veremos primeiro as rexións de que consta o xenoma retroviral. Este ten os xenes codificantes no centro, e as partes importantes para o funcionamento da transcriptase inversa están nos dous extremos. O ARN retroviral ten un extremo 5' e outro 3'. O sitio onde oprimerse une (alinea ouanneals) ao ARN viral chámase sitio de unión aoprimer(PBS). O extremo 5’ do ARN no sitio PBS denomínase U5, e o extremo 3' do ARN do PBS chámase líder. Utilízase unprimerunido a un ARNt celular. Oprimerde ARNt está desenrolado ao longo de 14 a 22nucleótidose forma un dúplex cos pares de bases apareadas co ARN viral no PBS. O feito de que o PBS estea localizado preto do extremo 5’ do ARN viral é pouco común porque a transcriptase inversa sintetiza o ADN do extremo 3’ doprimeren dirección 5’→3’ (con respecto ao molde de ARN). Por tanto, oprimere a transcriptase inversa deben ser recolocados despois no extremo 3’ do ARN viral. Isto quere dicir, que o encima ten que cambiar dun extremo a outro do ARN molde durante a reversotranscrición. Para realizar este reposicionamento, necesítanse seguir varios pasos e varias actividades encimáticas como a deADN polimerase,ribonuclease H (RNase H) e desenrolamento depolinucleótidos.[9]
A transcriptase inversa do VIH tamén ten unha actividade deribonucleaseque degrada o ARN viral durante a síntese do ADNc, xunto coa súa actividade deADN polimerase ADN dependenteque copia a cadeasentidoADNc orixinando unha cadeaantisentidode ADN, formando así entre as dúas o intermediato de ADN bicatenario viral (ADNv) que se integrará no xenoma da célula.[10]
Fases da reversotranscrición
[editar|editar a fonte]
A actuación da transcriptase inversa faise seguindo unha serie de pasos (ver a imaxe), que son:
- UnARNtcelular específico actúa comoprimer(cebador) e hibrídase cunha parte complementaria do xenoma do virus chamada sitio de unión aoprimerou PBS, preto do extremo 5'.
- Despois fórmaseADN complementariosobre as rexións U5 (rexión non codificante) e R (unha secuencia repetida que se encontra a ambos os lados da molécula de ARN) do ARN viral.
- Un dominio do encima transcriptase inversa denominadoRNAse Hdegrada oextremo 5’do ARN, o que elimina as rexións U5 e R do ARN viral.
- Oprimer(que estaba no extremo 5') entón "salta" ao extremo 3’ do xenoma viral e a pequena cadea de ADN acabada de sintetizar hibrídase coa rexión R complementaria do ARN dese outro extremo (como dixemos, hai unha rexión R en cada extremo do ARN).
- A pequena cadea de ADN complementario (ADNc) alóngase en dirección ao outro extremo completando a súa síntese, e a maioría do ARN viral é degradado polo dominio RNAse H (só queda a rexión PP).
- Unha vez que a primeira cadea de ADN (ADNc) está completa, iníciase a síntese dunha segunda cadea de ADN cara ao extremo 5' do ARN, empezando desde o ARN viral que quedou (rexión PP).
- Prodúcese entón outro "salto" cara ao extremo 3' do ARN no que o PBS da segunda cadea se hibrida co PBS complementario da primeira cadea. Libérase oprimer/ARNt.
- Ambas as cadeas esténdense máis para completar o que lles falta, e orixínase un ADN bicatenario que está en condicións de poder incorporarse ao xenoma do hóspede polo encima víricointegrase.
Como se ve, a creación do ADN de dobre cadea implica atransferencia de cadea,na cal hai unha translocación do curto produto de ADN sintetizado (a partir da síntese inicial de ADN ARN dependente) a rexións molde aceptoras no outro extremo do xenoma viral de ARN, que son despois atinguidas e procesadas pola transcritse inversa na súa actividade de síntese de ADN dependente de ADN.[11]
En eucariotas
[editar|editar a fonte]- Artigos principais:RetrotransposóneTelomerase.
Certos tramos autorreplicantes dos xenomaseucarióticoschamadosretrotransposónsutilizan tamén unha transcriptase inversa para moverse dunha posición do xenoma e inserirse noutra por medio dun intermediato de ARN. Son moi abundantes nos xenomas de plantas e animais.
Atelomeraseé outro encima con actividade de reverso transcriptase que se encontra en moitos eucariotas, incluíndo os humanos, que leva o seu propio molde de ARN; este ARN utilizase como molde para areplicación do ADNnos extremos (telómeros) dos cromosomas.[12]
En procariotas
[editar|editar a fonte]As transcriptases inversas tamén se encontran nosRetron msr RNAsbacterianos,que son secuencias específicas de ARN que conteñen unha parte (r) que codifica unha transcriptase inversa, e se usan na síntese de ADNs monocatenatios multicopia (msDNA). Para iniciar a síntese do ADN, necesítase un cebador ouprimer.Nas bacterias, oprimersintetízase durante a replicación.[13]
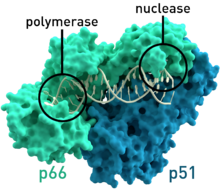
Estrutura
[editar|editar a fonte]Os encimas transcriptase inversa inclúen unha ADN polimerase ARN dependente e unha ADN polimerase ADN dependente, que funcionan conxuntamente para realizar a transcrición. Ademais da función de transcrición, as transcriptases inversas retrovirais teñen un dominio pertencente á familia dasRNase Hque é fundamenteal na súa replicación.
Fidelidade da replicación
[editar|editar a fonte]Durante o ciclo de replicaión dun retrovirus utilízanse diferentes sistemas de replicación. Primeiramente, a transcriptase inversa sintetiza o ADN viral a partir do ARN viral, e despois a partir da cadea de ADN acabada de sintetizar. O segundo proceso de replicación ten lugar cando a célula hóspede replica coa súaADN polimeraseo seu ADN e xunto con el o ADN viral integrado no seu xenoma. Finalmente, aARN polimerase IItranscribe o ADN retroviral a ARN, que será despois traducido e empaquetado nos virións. Por tanto, poden ocorrer mutacións durante todos estes pasos de replicación.[14]
A transcriptase inversa ten unha alta taxa de erros á hora de transcribir o ARN a ADN, xa que, a diferenza doutrasADN polimerases,non ten capacidade decorrección de probas.Esta alta taxa de erros permite que se acumulen asmutaciónsde forma rápida. Por exemplo, as transcriptases inversas dispoñibles comercialmente producidas porPromegateñen, segundo os seus manuais, unha taxa de erro de 1 en 17.000 bases para oAMVe de 1 en 30.000 bases para oM-MLV.[15]
Ademais de orixinarenpolimorfismo dun único nucleótido,as transcriptases inversas están implicadas tamén en procesos como afusión de transcritos,barallamento de exóns(exon shuffling) e na creación de transcritos artificiaisantisentido.[16][17]Especulouse que a actividade decambio de moldeda transcriptase inversa, que foi demostrada tantoin vitrocomain vivo,pode ser unha das causas de que se encontren varios miles de transcritos non anotados nos xenomas dos organismos modelos.[18]
Aplicacións
[editar|editar a fonte]
Drogas antivirais
[editar|editar a fonte]- Artigo principal:Inhibidor da transcriptase inversa.
Como oVIHutiliza a transcriptase inversa para copiar o seu material xenético e xerar novos virus, deseñáronse drogas específicas que interrompen este proceso e suprimen o crecemento da infección viral. En conxunto estas drogas chámanseinhibidores da transcriptase inversae inclúen os análogos denucleósidosenucleótidoszidovudina(nome comercial Retrovir),lamivudine(Epivir) etenofovir(Viread), e tamén inhibidores non análogos de nucleósidos comonevirapina(Viramune).
Bioloxía molecular
[editar|editar a fonte]A transcriptase inversa úsase correntemente en investigación para aplicar a técnica dareacción en cadea da polimeraseaoARN,o que se denominareacción en cadea da polimerase con transcriptase inversa(RT-PCR). A técnica da reacción en cadea da polimerase clásica pode aplicarse só ao ADN, pero, coa axuda da transcriptase inversa, o ARN pode transcribirse a ADN, facendo posible a análise por PCR de moléculas de ARN. A transcriptase inversa utilízase tamén para crearlibrarías de ADNca partir deARNm.A dispoñibilidade comercial da transcriptase inversa serviu para aumentar moito o coñecemento na área da bioloxía molecular, xa que, xunto con outros encimas, permitiu aos científicos clonar, secuenciar e caracterizar o ARN.
A transcriptase inversa empregouse tamén na produción deinsulina.Ao inserir en bacterias o ARNm eucariótico que codifica a insulina xunto con transcriptase inversa, o ARNm pode integrarse el mesmo no xenoma procariótico, e producir despois grandes cantidades de insulina, o que permitiu abandonar o método usado antes de extracción da insulina do páncreas deporco.Polo contrario, non funcionaría inserir nas bacterias directamente ADN eucariótico (en vez de ARNm) porque é un ADN que contén seccións non cofificantes chamadasintróns,e os seus transcritos non setraduciríancon éxito nosribosomasbacterianos. O ARNm eucariótico madura eliminando os intróns, polo que é máis adecuado para funcionar dentro das células procariotas, as cales carecen de intróns.
Notas
[editar|editar a fonte]- ↑PDB3KLF;Tu X, Das K, Han Q, Bauman JD, Clark AD Jr, Hou X, Frenkel YV, Gaffney BL, Jones RA, Boyer PL, Hughes SH, Sarafianos SG, Arnold E (setembro de 2010)."Structural basis of HIV-1 resistance to AZT by excision.".Nat. Struct. Mol. Biol.17(10): 1202–9.PMC2987654.PMID20852643.doi:10.1038/nsmb.1908.
- ↑BUSCatermostranscriptase, transcriptase inversa, inhibidor da transcriptase inversa
- ↑Definición deRNA-polimerasenoDicionario de GalegodeIr Indoe aXunta de Galicia.
- ↑Definicións noDicionario da Real Academia Galegae noPortal das Palabrasparatranscrición.
- ↑Temin HM, Mizutani S (1970). "RNA-dependent DNA polymerase in virions of Rous sarcoma virus".Nature226(5252): 1211–3.PMID4316301.doi:10.1038/2261211a0.
- ↑Baltimore D (1970). "RNA-dependent DNA polymerase in virions of RNA tumour viruses".Nature226(5252): 1209–11.PMID4316300.doi:10.1038/2261209a0.
- ↑"Central dogma reversed".Nature226(5252): 1198–9. 1970.PMID5422595.doi:10.1038/2261198a0.
- ↑Bio-Medicine.org - RetrovirusArquivado28 de abril de 2021 enWayback Machine.Consultado o 17 de febreiro de 2009
- ↑Bernstein A, Weiss R, Tooze J (1985). "RNA tumor viruses".Molecular Biology of Tumor Viruses(2nd ed.). Cold Spring Harbor, N.Y: Cold Spring Harbor Laboratory.
- ↑Doc Kaiser's Microbiology Home Page > IV. VIRUSES > F. ANIMAL VIRUS LIFE CYCLES > 3. The Life Cycle of HIVArquivado26 de xullo de 2010 enWayback Machine.Community College of Baltimore County. Updated: Jan., 2008
- ↑Telesnitsky A, Goff SP (1993). "Strong-stop strand transfer during reverse transcription". En Skalka, M. A., Goff, S.P.Reverse transcriptase(1st ed.). New York: Cold Spring Harbor. p.49.ISBN0-87969-382-7.
- ↑Krieger M, Scott MP, Matsudaira PT, Lodish HF, Darnell JE, Zipursky L, Kaiser C, Berk A (2004).Molecular cell biology.New York: W.H. Freeman and CO.ISBN0-7167-4366-3.
- ↑Hurwitz J, Leis JP (1972)."RNA-dependent DNA polymerase activity of RNA tumor viruses. I. Directing influence of DNA in the reaction".J. Virol.9(1): 116–29.PMC356270.PMID4333538.
- ↑Bbenek K, Kunkel AT (1993). "The fidelity of retroviral reverse transcriptases". En Skalka, M. A., Goff, P. S.Reverse transcriptase.New York: Cold Spring Harbor Laboratory Press. p.85.ISBN0-87969-382-7.
- ↑"Promega kit instruction manual (1999)"(PDF).Arquivado dendeo orixinal(PDF)o 21 de novembro de 2006.Consultado o 29 de marzo de 2013.
- ↑Houseley J, Tollervey D (2010)."Apparent non-canonical trans-splicing is generated by reverse transcriptase in vitro".PLoS ONE5(8): e12271.PMC2923612.PMID20805885.doi:10.1371/journal.pone.0012271.
- ↑Zeng XC, Wang SX (2002). "Evidence that BmTXK beta-BmKCT cDNA from Chinese scorpion Buthus martensii Karsch is an artifact generated in the reverse transcription process".FEBS Lett.520(1-3): 183–4; author reply 185.PMID12044895.doi:10.1016/S0014-5793(02)02812-0.
- ↑van Bakel H, Nislow C, Blencowe BJ, Hughes TR (2011). "Response to" The Reality of Pervasive Transcription"".PLoS Biology9(7): e1001102.doi:10.1371/journal.pbio.1001102.
Véxase tamén
[editar|editar a fonte]Outros artigos
[editar|editar a fonte]Ligazóns externas
[editar|editar a fonte]- MeshName - RNA+Transcriptase[1]
- animación da acción da reversotranscriptase e de tres dos seus inhibidores
- Molécula do mes de setembro de 2002Arquivado09 de abril de 2020 enWayback Machine.(consultado o 29 de marzo de 2013) en RCSB PDB
- Animación da replicación do VIH. (novembro 2008, consultado en marzo de 2013). Vídeo de Boehringer Ingelheim.
