Z-DNS
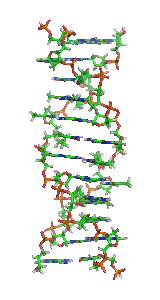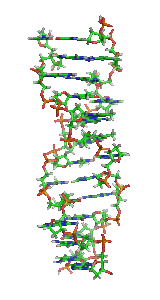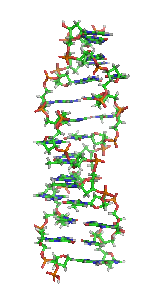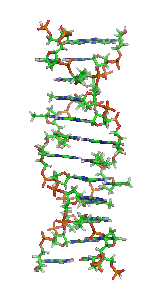
AZ-DNSa DNS egyik kettőshélix-szerkezete. E szerkezet balos, vagyis a hélix balra fordul cikcakkos mintában, nem jobbra, mint a gyakoribbB-DNSés azA-DNS.A Z-DNS feltehetően az A- és B-DNS-hez hasonlóan biológiailag aktív.
Története
[szerkesztés]A balra forgató DNS-t előszörRobert Wellsés társai javasolták azinozin–citozinpolimer szerkezetére.[1]„Fordított”körkörös dikroizmustészleltek e DNS-ekre, és ezt – helytelenül – a szálak balos egymásra fordulásának tekinteték. A Z- és a gyakoribb B-DNS kapcsolatát Pohl és Jovin mutatták ki,[2]akik kimutatták, hogy a poli(dG–dC) ultraibolya körkörös dikroizmusa 4 M-os NaCl-oldatban megfordult, és hogy a poli-d(I–C)·poli-d(I–C) szerkezete jobbra forgató D-DNS volt. Azt a feltételezést, hogy ez B-DNS-ről Z-DNS-re való átalakulás eredménye volt, az oldatok és a Z-DNS-kristályokRaman-spektrumánakvizsgálata igazolta.[3]Ezután a „Z-DNS” kristályszerkezetét is közölték, mely az első egykristály-röntgenszerkezet volt DNS-részletről (ad(CG)3önkomplementer DNS-hexameré). Erről kiderült, hogy ez balra forgató kettős hélix két antipárhuzamos lánccal, melyeket Watson–Crick-bázispárok tartottak össze. EztAndrew H. J. Wang,Alexander Richés társai mutatták ki 1979-ben aMIT-nél.[4]A B–Z-DNS kapcsolat 2005-ös kristályosítása[5]lehetővé tette a Z-DNS sejtekben játszott szerepének jobb megismerését. Z-DNS-szakasz keletkezésekor B–Z kapcsolatoknak kell lenni a két végén, a genom többi részén lévő B-DNS-hez kapcsolva azt.
2007-ben a Z-DNS RNS-változatát, aZ-RNS-t is előállították, melyet azA-RNSbalra forgatóvá alakított változataként írtak le.[6]Az A-ból Z-RNS-be való átmenetet azonban már 1984-ben leírták.[7]
Szerkezet
[szerkesztés]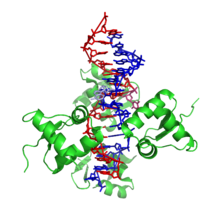
A Z-DNS jelentősen eltér a jobbra forgató változatoktól. Gyakran a Z-DNS-t a B-DNS-sel hasonlítják össze a főbb különbségek illusztrálásához. A Z-DNS-hélix balra forgat, szerkezete két bázispáronként ismétlődik. A nagy és kis bemélyedések szélessége az A- és B-DNS-től eltérően kevéssé tér el. E szerkezet keletkezése általában kedvezőtlen, de bizonyos helyzetek elősegíthetik, például váltakozópurin-pirimidinsorozatok (különösen a poli(dGC)2), a negatív DNS-szupertekeredés, a magas sótartalom és bizonyos kationok (fiziológiás hőmérsékleten (37 °C) és 7,3–7,4-es pH-n). A Z-DNS B-DNS-hez kapcsolódhat egy bázispár kiemelésével.[8]A Z-DNS-konformáció tanulmányozása nehéz, mivel a kettős hélixben nem stabil. Ehelyett ez átmeneti szerkezet, melyet néha a biológiai aktivitás indukál, majd gyorsan eltűnik.[9]
Z-DNS-szerkezet előrejelzése
[szerkesztés]Egy DNS-szekvencia Z-DNS-szerkezet-alkotásának valószínűsége előrejelezhető. A DNS B-ből Z-formába alakulásának valószínűségét jósló algoritmust, aZHuntotP. Sing Hoírta 1984-ben aMIT-nél.[10]Később eztTracy Camp,P. Christoph Champ,Sandor MauriceésJeffrey M. Vargasontovábbfejlesztették genomszintű elemzésre (Ho a vezető vizsgáló).[11]
Z-DNS keletkezése B-DNS-ből
[szerkesztés]A Z-DNS 1979-es felfedezése és kristályosítása óta nem ismert a B-DNS Z-DNS-sé alakulásának módja.[12]A B-DNS Z-DNS-sé alakulásának módja atomi szinten nem volt ismert, de 2010-ben számítógépes szimulációval igazolták a B→Z átalakulás korábban feltételezett összehangolt mechanizmusnál alacsonyabb aktivációs energiáját.[13]Ennek számítási bizonyítása után ez még kísérletileg is bizonyítandó a további igazoláshoz, Lee és társai cikkükben ezt írják: „A jelenlegi [számított] eredményegymolekulás FRET(smFRET) kísérletekkel ellenőrizhető később”.[13]2018-ban a B-DNS Z-DNS-sé való átalakulását smFRET-assay-kkel igazolták.[14]Ezt DNS-hez kötött donor és akceptor fluoreszcens festékek (fluoroforok) közti egymáshoz viszonyított intenzitás mérésével igazolták elektronátvitel közben.[15][16]A fluoroforok távolsága felhasználható a festékek helyzetének és a DNS változásainak mennyiségi elemzéséhez. Egy nagy affinitású Z-DNS-kötő fehérjét,a hZαADAR1-et[17]használtak különböző koncentrációban a B-DNS Z-DNS-sé alakulását.[14]Az smFRET-assay-k B* köztes állapotot mutattak, melyek a B-DNS-hez kötött hZαADAR1 növekedésekor stabilizálták azt.[14]Ez a nagy kapcsolati energia elkerülésére történik, így a B-DNS Z-DNS-sé tud alakulni nagy, zavaró energiaváltozás nélkül. Ez egybeesik Lee és társai munkájával, mely alapján a mechanizmus lépésenként történik, és célja a B-DNS Z-DNS-sé alakulásának aktivációs energiájának csökkentése.[13]A kötőfehérjék nem stabilizálják a Z-DNS-t keletkezése után, hanem csak a Z-DNS B* állapotból való kialakulását segítik, amelyet a nagy affinitású fehérjék B-DNS-hez kötődése alakít ki.[14]
Biológiai jelentősége
[szerkesztés]A Z-DNS az I-es típusú interferonválaszokat szabályozza, ezt 3 ismert ritka örökletes betegség tanulmányaiban (dyschromatosis symmetrica hereditaria(OMIM127400)),Aicardi–Goutières-szindróma(OMIM615010) éskétoldali striatalis nekrózis/disztónia) erősítették meg. A haploid ADAR-transzkriptom lehetővé teszi a Zα-változatok betegséghez kapcsolását, ez alapján a genetikai információt a DNS-ben a szekvencia és az alak is kódolja.[18]Az I. típusú interferonválaszok rákban való szabályzását az is megalapozza, hogy egy tumortípus 40%-a az ADAR enzimtől függött.[19]
Korábbi tanulmányok a Z-DNS-t összekapcsolták azAlzheimer-kórralés aszisztémás lupus erythematosusszal.Ennek illusztrálására a hippocampusban lévő normál, közepesen és erősen érintett DNS-t vizsgáltak. Körkörös dikroizmussal kimutatták a Z-DNS jelenlétét az erősen érintett DNS-ben.[20]A tanulmány szerint a közepesen érintett DNS-ben voltak nagyobb B–Z köztes konformációjú DNS-részek. Ez azért fontos, mert ebből kiderült, hogy a B→Z-DNS átmenet függ az Alzheimer-kór állapotával.[20]Ezenkívül a Z-DNS összefügg a szisztémás lupus erythematosusszal (SLE) a természetes antitestek révén. Jelentős mennyiségű anti-Z-DNS antitest található SLE-s betegekben, más reumatoid betegségekben ezek nem voltak jelen.[21]Ezen antitestek két típusban fordulnak elő. Radioimmunassay-ben kimutatták, hogy egyikük a Z- és a denaturált DNS felszínén lévő bázisokkal, a másik csak a cikcakkos Z-DNS-vázzal reagál. Az Alzheimer-kórhoz hasonlóan a betegség állapotától függnek az antitestek, a legaktívabb SLE-szakaszokban van a legtöbb ilyen antitest.
Z-DNS a transzkripcióban
[szerkesztés]A Z-DNS torziós feszültsége feltehetően csökkentranszkripciósorán, és a negatív szupertekeredéssel függ össze.[5][22]Azonban míg a szupertekeredés a transzkripcióval és a replikációval is összefügg, a Z-DNS-keletkezés a transzkripció sebességével függ össze.[23]
Ahumán 22. kromoszómatanulmányozása korrelációt mutatott a Z-DNS-képző részek és azI. magi faktorpromoterei közt. Így egyes humán gének transzkripcióját a Z-DNS-képzés és az I. magi faktor aktivációja szabályozhatja.[11]
A promoterek előtti Z-DNS stimulálja a transzkripciót. A legnagyobb aktivitásnövekedés a promoterszekvencia után 3 fordulattal lévő Z-DNS esetén van jelen. Továbbá micrococcalis nukleáz-keresztkötéssel[24]a Z-DNS valószínűleg nem hoz létrenukleoszómát,mely gyakran a Z-DNS-szakasz előtt vagy után van. Ezért a Z-DNS feltehetően a nukleoszóma-elhelyezés határát kódolja. Mivel a nukleoszómák helye befolyásolja atranszkripciós faktorokkötését, a Z-DNS fetehetően a transzkripció sebességét szabályozza.[24]
AzRNS-polimerázhozhasonlóan negatív szupertekeredéssel és aktív transzkripcióval létrehozott Z-DNS növeli a genetikai instabilitást, a promoterekhez közelimutagenezisvalószínűségét növelve.[25]EgyEscherichia colin végzett tanulmány szerint adeléciókspontán történnek a Z-DNS-alkotó szekvenciát tartalmazóplazmidrészeken.[26]Az emlőssejtekben az ilyen szekvenciák jelenléte nagy deléciókat okoz a kromoszomális kettősszál-törések miatt. Mindkét genetikai módosulásgéntranszlokációkkalhozható összefüggésbe, melyek a rákok, például aleukémiaés alimfómaesetén jellemzők, mivel a tumorsejtek törési helyei Z-DNS-alkotó szekvenciák körül vannak.[25]A baktériumplazmidok kisebb delécióireplikációs csúszástokoznak, míg az emlőssejtek nagyobb delécióit anem homológ végösszeillesztésokozza, mely hibákra hajlamos.[25][26]
Azetídium-bromidTrypanosoma-fajokra gyakorolt mérgező hatását akinetoplasztisz-DNS Z-formává alakítása okozza. Ezt az interkalációja okozza, melyet a DNS-szerkezet lazulása követ, mely a DNS szétválását, Z-formává alakulását és a replikáció gátlását okozza.[27]
A Zα domén felfedezése
[szerkesztés]Az első nagy affinitással Z-DNS-hez kapcsolódó domént azADAR1-ben fedezték fel Alan Herbert módszerével.[28][29]KrisztallográfiaiésNMR-tanulmányok megerősítették a biokémiai eredményeket, melyek szerint a domén nem szekvenciaspecifikusan kötődik Z-DNS-hez.[30][31][32]Hasonló doméneket fedeztek fel számos más fehérjébenszekvenciahomológiarévén.[29]A Zα domén azonosítása lehetővé tett más krisztallográfiai eredményeket, melyek a Z-RNS és a B–Z kapcsolat felismerését tették lehetővé. Feltehetően az ADAR1 Z-DNS-kötő doménje az új RNS szekvenciáját módosító enzimet aktív transzkripciós helyekre teheti.[33][34]A Zα, a Z-DNS és a Z-RNS genomvédelmi szerepe az Alu retroelemekkel szemben a dsRNS-sel szembeni nem specifikus immunválaszok szabályzásává fejlődött. A Zα-mutációk humán interferonopátiákatt, például Aicardi–Goutières-szindrómát okozhatnak.[35][18]Továbbá a Zα domének a stresszgranulákban is jelen lehetnek nem specifikus nukleinsavkötő képességük révén, és a különböző Zα domének eltérően kötődnek a Z-nukleinsavhoz, fontos lehetőségeket biztosítva a gyógyszerfejlesztésben.
A Z-DNSVaccinia-E3L-hez kapcsolódásának következményei
[szerkesztés]A Z-DNS intenzívebb kutatása során kiderült, hogy a Z-DNS Z-DNS-kötő fehérjékhez kapcsolódhat London-diszperziós erőkkel és hidrogénkötésekkel.[36]Az E3L gén terméke, azVaccinia-E3L fehérje például Z-DNS-kötő, működése egy Z-DNS-kötő emlősfehérjéhez hasonló.[37][38]Az E3L nemcsak köt a Z-DNS-hez, hanem felhasználható aVacciniahimlővírus virulenciájának méréséhez egerekben. A virulenciát meghatározó két fontos része az N- és a C-terminus. Az N-terminális szekvencia a Zα, más névenadenozin-deamináz z-α fehérjéhezhasonlít, míg a C-terminális kétszálú RNS-kötő részből áll.[37]Kim, Y. és társai által a Massachusettsi Műegyetemen végzett kutatások alapján az E3L fehérje N-terminusának Zα-ra való cseréje a vírus patogenitását kevéssé befolyásolta.[37]Ezzel szemben az összes Z-DNS-kötő aminosav eltávolítása a virulenciát csökkentette.[37]Ezek alapján a hasonló Z-DNS-kötő aminosavak a virulenciát meghatározó legfontosabb tényezők, a Z-DNS-kötésben részt nem vevő aminosavak kevéssé befolyásolják azt. Továbbá az E3LVaccinia-vírust tartalmazó vakcinák általi Z-DNS-affinitás-csökkentése csökkenti a humán negatív reakciókat is a vírusra.[37]
Ezenkívül Alexander Rich és Jin-Ah Kwon kimutatták, hogy az E3L a humán IL-6, NF-AT és p53transzaktivátora.Eredményeik alapján az E3L-tartalmúHeLasejtekben a humán IL-6, NF-AT és p53 gének jobban kifejeződnek, és egyes Z-DNS-kötő aminosavak mutációi vagy deléciói ezt csökkentik.[36]A Tyr 48 és a Pro 63 mutációi e gének transzaktivációját csökkentik a hidrogénkötés eltűnése és az E3L és a Z-DNS közti London-diszperziós erők miatt.[36]Így ezen eredmények alapján a Z-DNS és a Z-DNS-kötő fehérjék kölcsönhatásának csökkenése csökkenti a virulenciát és a génexpressziót, megmutatva a Z-DNS és az E3L-kötő fehérje közti kötések szükségességét.
A leggyakoribb DNS-ek összehasonlítása
[szerkesztés]

| Geometria | A-DNS | B-DNS | Z-DNS |
|---|---|---|---|
| Hélixállás | jobbra forgat | jobbra forgat | balra forgat |
| Ismétlődő egység | 1 bázispár | 1 bázispár | 2 bázispár |
| Forgás/bázispár | 32,7° | 34,3° | 60°/2 |
| Átlagos bp/fordulat | 11 | 10 | 12 |
| Bázispár-inklináció | +19° | −1,2° | −9° |
| Tengelymenti emelkedés/bp | 2,6 Å (0,26 nm) | 3,4 Å (0,34 nm) | 3,7 Å (0,37 nm) |
| Fordulatonkénti emelkedés | 28,6 Å (2,86 nm) | 35,7 Å (3,57 nm) | 45,6 Å (4,56 nm) |
| Átlagos csavarodás | +18° | +16° | 0° |
| Glikozilszög | anti | anti | pirimidin: anti,
purin: szin |
| Nukleotidok foszfát-foszfát távolsága | 5,9 Å | 7,0 Å | C: 5,7 Å, G: 6,1 Å |
| Cukorbemélyedés | C3'-endo | C2'-endo | C: C2'-endo, G: C3'-endo |
| Átmérő | 23 Å (2,3 nm) | 20 Å (2,0 nm) | 18 Å (1,8 nm) |
Jegyzetek
[szerkesztés]- ↑(1970) „Physical and enzymatic studies on poly d(I–C)·poly d(I–C), an unusual double-helical DNA”.Nature228(5277), 1166–1169. o.DOI:10.1038/2281166a0.PMID4321098.
- ↑(1972) „Salt-induced co-operative conformational change of a synthetic DNA: equilibrium and kinetic studies with poly(dG-dC)”.Journal of Molecular Biology67(3), 375–396. o.DOI:10.1016/0022-2836(72)90457-3.PMID5045303.
- ↑(1981) „High salt form of poly(dG–dC)·poly(dG–dC) is left handed Z-DNA: raman spectra of crystals and solutions”.Nucleic Acids Research9(20), 5443–5457. o.DOI:10.1093/nar/9.20.5443.PMID7301594.PMC327531.
- ↑(1979) „Molecular structure of a left-handed double helical DNA fragment at atomic resolution”.Nature282(5740), 680–686. o.DOI:10.1038/282680a0.PMID514347.
- ↑ab(2005) „Crystal structure of a junction between B-DNA and Z-DNA reveals two extruded bases”.Nature437(7062), 1183–1186. o.DOI:10.1038/nature04088.PMID16237447.
- ↑(2007) „A left-handed RNA double helix bound by the Z Alpha domain of the RNA-editing enzyme ADAR1”.Structure15(4), 395–404. o.DOI:10.1016/j.str.2007.03.001.PMID17437712.PMC2082211.
- ↑(1984. október 1.) „'Z-RNA'—a left-handed RNA double helix”.Nature311(5986), 584–586. o.DOI:10.1038/311584a0.PMID6482970.
- ↑(2010. május 1.) „Crystal structure of a junction between two Z-DNA helices”.Proceedings of the National Academy of Sciences107(20), 9088–9092. o.DOI:10.1073/pnas.1003182107.PMID20439751.PMC2889044.
- ↑(2006) „Reversible B/Z-DNA transition under the low salt condition and non-B-form poly(dA)poly(dT) selectivity by a cubane-like europium-l-aspartic acid complex”.Biophysical Journal90(9), 3203–3207. o. [2008. október 12-i dátummal azeredetibőlarchiválva].DOI:10.1529/biophysj.105.078402.PMID16473901.PMC1432110.(Hozzáférés: 2023. október 25.)
- ↑(1986) „A computer aided thermodynamic approach for predicting the formation of Z-DNA in naturally occurring sequences”.EMBO Journal5(10), 2737–2744. o.DOI:10.1002/j.1460-2075.1986.tb04558.x.PMID3780676.PMC1167176.
- ↑ab(2004) „Distributions of Z-DNA and nuclear factor I in human chromosome 22: a model for coupled transcriptional regulation”.Nucleic Acids Research32(22), 6501–6510. o.DOI:10.1093/nar/gkh988.PMID15598822.PMC545456.
- ↑(1979. december 1.) „Molecular structure of a left-handed double helical DNA fragment at atomic resolution”.Nature282(5740), 680–686. o.DOI:10.1038/282680a0.ISSN0028-0836.PMID514347.
- ↑abc(2010. augusztus 5.) „Transition between B-DNA and Z-DNA: Free Energy Landscape for the B−Z Junction Propagation”.The Journal of Physical Chemistry B114(30), 9872–9881. o.DOI:10.1021/jp103419t.ISSN1520-6106.PMID20666528.
- ↑abcd(2018. március 23.) „Unveiling the pathway to Z-DNA in the protein-induced B–Z transition”.Nucleic Acids Research46(8), 4129–4137. o.DOI:10.1093/nar/gky200.ISSN0305-1048.PMID29584891.PMC5934635.
- ↑(2013. június 3.) „Photobleaching Lifetimes of Cyanine Fluorophores Used for Single-Molecule Förster Resonance Energy Transfer in the Presence of Various Photoprotection Systems”.ChemBioChem14(9), 1075–1080. o.DOI:10.1002/cbic.201300030.ISSN1439-4227.PMID23733413.PMC3871170.
- ↑Didenko, Vladimir V. (2001. november 1.). „DNA Probes Using Fluorescence Resonance Energy Transfer (FRET): Designs and Applications”.BioTechniques31(5), 1106–1121. o.DOI:10.2144/01315rv02.ISSN0736-6205.PMID11730017.PMC1941713.
- ↑(1997. augusztus 5.) „A Z-DNA binding domain present in the human editing enzyme, double-stranded RNA adenosine deaminase”.Proceedings of the National Academy of Sciences94(16), 8421–8426. o.DOI:10.1073/pnas.94.16.8421.ISSN0027-8424.PMID9237992.PMC22942.
- ↑abHerbert, A. (2019). „Mendelian disease caused by variants affecting recognition of Z-DNA and Z-RNA by the Zα domain of the double-stranded RNA editing enzyme ADAR.”.European Journal of Human Genetics8(1), 114–117. o.DOI:10.1038/s41431-019-0458-6.PMID31320745.PMC6906422.
- ↑Herbert, A. (2019). „ADAR and Immune Silencing in Cancer.”.Trends in Cancer5(5), 272–282. o.DOI:10.1016/j.trecan.2019.03.004.PMID31174840.
- ↑ab(2002) „First Evidence to Show the Topological Change of DNA from B-DNA to Z-DNA Conformation in the Hippocampus of Alzheimer's Brain”.NeuroMolecular Medicine2(3), 289–298. o.DOI:10.1385/nmm:2:3:289.ISSN1535-1084.PMID12622407.
- ↑(1983. február 1.) „Z-DNA-specific antibodies in human systemic lupus erythematosus.”.Journal of Clinical Investigation71(2), 314–321. o.DOI:10.1172/jci110771.ISSN0021-9738.PMID6822666.PMC436869.
- ↑(2003) „Timeline: Z-DNA: the long road to biological function”.Nature Reviews Genetics4(7), 566–572. o.DOI:10.1038/nrg1115.PMID12838348.
- ↑(1991) „Transcription is associated with Z-DNA formation in metabolically active permeabilized mammalian cell nuclei”.Proceedings of the National Academy of Sciences88(6), 2259–2263. o.DOI:10.1073/pnas.88.6.2259.PMID2006166.PMC51210.
- ↑ab(2007) „Characterization of Z-DNA as a nucleosome-boundary element in yeastSaccharomyces cerevisiae”.Proceedings of the National Academy of Sciences104(7), 2229–2234. o.DOI:10.1073/pnas.0611447104.PMID17284586.PMC1892989.
- ↑abc(2006) „Z-DNA-forming sequences generate large-scale deletions in mammalian cells”.Proceedings of the National Academy of Sciences108(8), 2677–2682. o.DOI:10.1073/pnas.0511084103.PMID16473937.PMC1413824.
- ↑ab(1989) „Z-DNA-forming sequences are spontaneous deletion hot spots”.Proceedings of the National Academy of Sciences86(19), 7465–7469. o.DOI:10.1073/pnas.86.19.7465.PMID2552445.PMC298085.
- ↑(2010. december 1.) „The killing of African trypanosomes by ethidium bromide”.PLOS Pathogens6(12), e1001226. o.DOI:10.1371/journal.ppat.1001226.PMID21187912.PMC3002999.
- ↑(1993) „A method to identify and characterize Z-DNA binding proteins using a linear oligodeoxynucleotide”.Nucleic Acids Research21(11), 2669–2672. o.DOI:10.1093/nar/21.11.2669.PMID8332463.PMC309597.
- ↑ab(1997) „A Z-DNA binding domain present in the human editing enzyme, double-stranded RNA adenosine deaminase.”.Proceedings of the National Academy of Sciences94(16), 8421–8426. o.DOI:10.1073/pnas.94.16.8421.PMID9237992.PMC22942.
- ↑(1998) „The Zα domain from human ADAR1 binds to the Z-DNA conformer of many different sequences”.Nucleic Acids Research26(15), 2669–2672. o.DOI:10.1093/nar/26.15.3486.PMID9671809.PMC147729.
- ↑(1999) „Crystal structure of the Zα domain of the human editing enzyme ADAR1 bound to left-handed Z-DNA”.Science284(5421), 1841–1845. o.DOI:10.1126/science.284.5421.1841.PMID10364558.
- ↑(1999) „The solution structure of the Zα domain of the human RNA editing enzyme ADAR1 reveals a prepositioned binding surface for Z-DNA”.Proceedings of the National Academy of Sciences96(22), 2465–2470. o.DOI:10.1073/pnas.96.22.12465.PMID10535945.PMC22950.
- ↑(2001) „The role of binding domains for dsRNA and Z-DNA in thein vivoediting of minimal substrates by ADAR1”.Proceedings of the National Academy of Sciences98(21), 12132–12137. o.DOI:10.1073/pnas.211419898.PMID11593027.PMC59780.
- ↑Halber, D.:Scientists observe biological activities of 'left-handed' DNA.MIT News Office, 1999. szeptember 11. (Hozzáférés: 2008. szeptember 29.)
- ↑(2019) „Z-DNA and Z-RNA in human disease”.Communications Biology2,7. o.DOI:10.1038/s42003-018-0237-x.PMID30729177.PMC6323056.
- ↑abc(2005. augusztus 26.) „Biological function of the vaccinia virus Z-DNA-binding protein E3L: Gene transactivation and antiapoptotic activity in HeLa cells”.Proceedings of the National Academy of Sciences102(36), 12759–12764. o.DOI:10.1073/pnas.0506011102.ISSN0027-8424.PMID16126896.PMC1200295.
- ↑abcde(2003. május 30.) „A role for Z-DNA binding in vaccinia virus pathogenesis”.Proceedings of the National Academy of Sciences100(12), 6974–6979. o.DOI:10.1073/pnas.0431131100.ISSN0027-8424.PMID12777633.PMC165815.
- ↑(2004. február 2.) „Evidence that vaccinia virulence factor E3L binds to Z-DNA in vivo: Implications for development of a therapy for poxvirus infection”.Proceedings of the National Academy of Sciences101(6), 1514–1518. o.DOI:10.1073/pnas.0308260100.ISSN0027-8424.PMID14757814.PMC341766.
Fordítás
[szerkesztés]Ez a szócikk részben vagy egészben aZ-DNAcímű angol Wikipédia-szócikkezen változatánakfordításán alapul.Az eredeti cikk szerkesztőit annak laptörténete sorolja fel. Ez a jelzés csupán a megfogalmazás eredetét és a szerzői jogokat jelzi, nem szolgál a cikkben szereplő információk forrásmegjelöléseként.

