DNA
L'acido desossiribonucleicoodeossiribonucleico(siglaDNA,dall'ingleseDeoxyriboNucleic Acid[1][2][3]) è l'acido nucleicoche contiene leinformazioni geneticheper lo sviluppo, l'omeostasie la riproduzione di tutti gliesseri viventiconosciuti. Costituisce inoltre la molecola base del funzionamento di una piccola minoranza deivirus.[4]
Dal punto di vista chimico, il DNA è unpolimeroorganico a doppia catena i cuimonomerisono chiamatinucleotidi(desossiribonucleotidi).[5]I nucleotidi sono costituiti da tre componenti fondamentali: ungruppo fosfato,unozucchero(ildeossiribosio) e unabase azotata,legata alglicidecon unlegame N-glicosidico.Le basi azotate che entrano nella formazione dei nucleotidi sono quattro:adenina,timina,citosinaeguanina(nell'RNAal posto della timina è presente l'uracile). La doppia catena polinucleotidica del DNA ha struttura antiparallela, spiralizzata e complementare poiché le basi azotate di una catena si accoppiano con l'altra conlegami idrogenosecondo lo schema:A-TeG-C.
La sequenza dei nucleotidi costituisce ilcodice genetico,che è tradotto negliamminoacidicorrispondenti. La sequenza amminoacidica prodotta forma le proteine. Il processo dellasintesi proteicaavviene tramite la sintesi di una molecolaintermediadiRNA(RNA messaggero), che è formata per complementarità con le quattro basi dei nucleotidi del DNA in un processo noto cometrascrizione.Il DNA non genera solo filamenti di RNA destinati alla traduzione in aminoacidi, ma anche frammenti in grado di svolgere svariate funzioni biologiche, ad esempio all'interno deiribosomi,dove l'RNA ha una funzione strutturale.
Neglieucarioti,il DNA si complessa all'interno delnucleoin strutture chiamatecromosomi.Negli altri organismi, privi di nucleo, esso può essere organizzato in cromosomi o meno (nei batteri è presente un'unica molecola di DNA circolare a doppia catena). All'interno dei cromosomi, le proteine dellacromatinacome gliistoni,lecoesinee le condensine organizzano il DNA e lo avvolgono in strutture ordinate. Queste strutture guidano l'interazione tra il codice genetico e le proteine responsabili della trascrizione, contribuendo al controllo della trascrizione genica. Nel corso dellamitosiavviene lareplicazione del DNA,con cui l'informazione genica è trasmessa integralmente nel passaggio tra diverse generazioni cellulari.
Storia
[modifica|modifica wikitesto]Il DNA fu inizialmente isolato dal biochimicosvizzeroFriedrich Miescher,il quale, nel 1869, individuò una sostanza microscopica contenuta nelpusdi bende chirurgiche utilizzate. Dal momento che tale molecola aveva la sua localizzazione nelnucleo,egli la chiamònucleina.[6]Nel 1919Phoebus Leveneindividuò la struttura del nucleotide, composta da base azotata, zucchero e fosfato.[7]Levene suggerì che il DNA consistesse in un filamento di nucleotidi legati tra loro attraverso i fosfati. Egli, però, era convinto che tale filamento fosse corto e che le basi fossero disposte secondo un preciso ordine ripetuto. Nel 1937William Astburypresentò i primi risultati di alcuni studi didiffrazione a raggi X,i quali dimostrarono che il DNA ha una struttura estremamente regolare[8].Nel 1944Erwin Schrödingerasserì che, visto che secondo la fisica quantistica i sistemi di pochi atomi hanno un comportamento disordinato, il materiale genetico doveva essere costituito da una grande molecola non ripetitiva, sufficientemente stabile da mantenere l'informazione genetica, chiamata "cristallo aperiodico".[9]
Nel 1928Frederick Griffithscoprì che icaratteridella formasmooth( "liscia" ) diPneumococcuspotevano essere trasferiti alla formarough( "rugosa" ), miscelando i resti dibatterismoothmorti con batteriroughvivi.[10]Questo sistema, pur non fornendo nessuna evidenza su quale fosse la sostanza che determinava il cambiamento, mostrava che qualcosa potesse trasportare l'informazione genetica dai resti dei batteri morti a quelli vivi. Si parlò quindi di unprincipio trasformantein grado di modificare i batteri vivi. Nel 1943Oswald Theodore Averydimostrò, in uncelebre esperimentoinsieme aColin MacLeodeMaclyn McCarty,che il DNA è ilprincipio trasformantealla base di questo fenomeno.[11]Il ruolo del DNA nell'ereditarietàè stato provato, infine, nel 1953 daAlfred HersheyeMartha Chaseattraverso un altroclassico esperimento,che dimostrò che il materiale genetico delfago T2è effettivamente il DNA.[12]
Il 1953 è anche l'anno in cui, attraverso ulteriori immagini da diffrazione a raggi X[13]realizzate daRosalind Franklin,chimica-fisica inglese,James WatsoneFrancis Crickpresentarono[13],sulla rivistaNature,quello che è oggi accertato come il primo modello accurato della struttura del DNA,[14]ovvero il modello adoppia elica.A disegnarne il bozzetto fuOdile Speed,pittrice e moglie di Crick. Le evidenze sperimentali a supporto del modello di Watson e Crick furono riportate in una serie di cinque articoli pubblicati sullo stesso numero di Nature.[15]Tra questi figurava l'articolo dellaFrankline diRaymond Gosling,che conteneva i dati di diffrazione a raggi X, fondamentale per sostenere il modello.[16][17]Tale numero conteneva anche un articolo sulla struttura del DNA scritto daMaurice Wilkins.[18]Nel 1962, dopo la morte di Rosalind Franklin (a causa di un tumore provocato, probabilmente, dalle alte dosi di raggi X a cui si era esposta nel corso dei suoi esperimenti), Watson, Crick e Wilkins ricevettero congiuntamente ilPremio Nobel per la medicina.[19]Dal momento che la scoperta del modello si basò essenzialmente sui dati di Rosalind Franklin, ancora oggi esistono pareri molto eterogenei nella comunità scientifica su chi avrebbe dovuto ricevere tale premio.
In un'importante presentazione del 1957, Crick propose ildogma centrale della biologia molecolare,che fissa le relazioni tra DNA, RNA e proteine.[20]La conferma finale del meccanismo di replicazione basato sulla struttura a doppia elica fu fornita nel 1958 dall'esperimento di Meselson-Stahl.[21]Un successivo lavoro di Crick dimostrò come il codice genetico fosse basato su triplette di basi non sovrapposte, permettendo aHar Gobind Khorana,Robert HolleyeMarshall Warren Nirenbergdi decifrarlo.[22]Queste scoperte sono alla base della modernabiologia molecolare.
Nel 1961Marshall NirenbergeSevero Ochoascoprono che ogni tripletta dinucleotidicodifica per uno specifico amminoacido.
Struttura
[modifica|modifica wikitesto]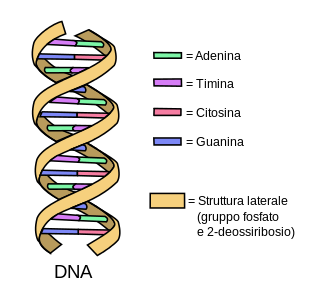
Il DNA è un lungopolimerocostituito da unità ripetute dinucleotidi.[23][24]La catena del DNA è larga tra i 22 e i 26ångström(da 2,2 a 2,6nanometri) e ogni unità nucleotidica è lunga 3,3 ångstrom (0,33 nanometri).[25]Sebbene ogni unità occupi uno spazio decisamente ridotto, la lunghezza dei polimeri di DNA può essere sorprendentemente elevata, dal momento che ogni filamento può contenere diversi milioni di nucleotidi. Ad esempio, il più grandecromosomaumano (ilcromosoma 1) contiene quasi 250 milioni dipaia di basi.[26]
Negli organismi viventi, il DNA non è quasi mai presente sotto forma di singolo filamento, ma come una coppia di filamenti saldamente associati tra loro.[14][27]Essi si intrecciano tra loro a formare una struttura definitadoppia elica.Ogni nucleotide è costituito da unoscheletro laterale,che ne permette illegame covalentecon i nucleotidi adiacenti, e da una base azotata, che instauralegami idrogenocon la corrispondente base azotata presente sul filamento opposto. Il composto formato da una base azotata legata allo zucchero è definitonucleoside;unnucleotideè invece un nucleoside a cui sono legati uno o più gruppi fosfato.[28]
La struttura laterale del DNA è composta da unità ripetute e alternate di gruppi fosfato e di 2-deossiribosio,[29]uno zuccheropentoso(a cinqueatomidicarbonio) che si lega ai fosfati adiacenti attraversolegami fosfodiestericipresso il terzo e il quinto carbonio; in pratica, ogni molecola di fosfato forma un ponte molecolare collegando, attraverso legami fosfodiesterici, il carbonio in posizione 3′ di una molecola di deossiribosio con quello in posizione 5′ dello zucchero successivo. Conseguenza di questi legami asimmetrici è che ogni filamento di DNA ha un senso, determinato dalla direzione dei legami fosfodiesterici. Le basi azotate, invece, si uniscono in posizione 1' dello zucchero desossiribosio con legami N-glicosidici. In una doppia elica, il senso di un filamento è opposto a quello del filamento complementare. Per tale motivo, i due filamenti che costituiscono una doppia elica sono detti antiparalleli. Le estremità asimmetriche di un filamento di DNA sono definiteestremità5′(cinque primo) edestremità3′(tre primo). La principale differenza tra il DNA e l'RNA è lo zucchero pentoso utilizzato: l'RNA, infatti, utilizza ilribosio.[27]
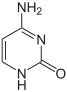
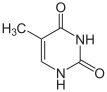
La doppia elica del DNA è stabilizzata dai legami idrogeno che si instaurano tra le basi azotate presenti sui due filamenti.[30]Le quattro basi che sono presenti nel DNA sono l'adenina(abbreviata con la lettera A), lacitosina(C), laguanina(G) e latimina(T). Tutte e quattro le basi hanno strutturaeterociclica,ma adenina e guanina sono, dal punto di vista strutturale, derivate dellapurina,e pertanto dettebasi puriniche,mentre citosina e timina sono correlate allapirimidinae dettebasi pirimidiniche.[27]Esiste una quinta base, di tipo pirimidinico, chiamatauracile(U), ma essa non è di norma presente nelle catene di DNA. L'uracile è altresì presente nei filamenti di RNA al posto della timina, dalla quale si differenzia per la mancanza di ungruppo metile.L'uracile è presente nel DNA solo come prodotto della degradazione della citosina. Solo nelbatteriofagoPBS1 tale base può essere utilizzata all'interno del DNA.[31]Al contrario, è molto più frequente individuare la timina all'interno di molecole di RNA, a causa dellametilazioneenzimaticadi diversi uracili. Questo evento avviene solitamente a carico di RNA con funzione strutturale o enzimatica (rRNAetRNA).[32]
La doppia elica è una spirale destrorsa. Con l'avvitarsi su sé stessi dei due filamenti, restano esposti dei solchi tra i diversi gruppi fosfato. Ilsolco maggioreè largo 22 Å, mentre ilsolco minoreè largo 12 Å.[33]La differente ampiezza dei due solchi si traduce concretamente in una differente accessibilità delle basi, a seconda che si trovino nel solco maggiore o minore. Proteine che legano il DNA, come i fattori di trascrizione, dunque, solitamente prendono contatto con le basi presenti nel solco maggiore.[34][35]
Appaiamento delle basi
[modifica|modifica wikitesto]Ogni tipo di base presente su un filamento forma un legame con la base posta sul filamento opposto. Tale evento è noto comeappaiamentocomplementare.Le basi puriniche formano legami idrogeno con le basi pirimidiniche: A può legare solo T e G può legare solo C. L'associazione di due basi viene comunemente chiamatapaio di basied è l'unità di misura maggiormente utilizzata per definire la lunghezza di una molecola di DNA. Dal momento che i legami idrogeno non sonocovalenti,essi possono esser rotti e riuniti in modo relativamente semplice, poiché questi sono legami ad alta energia. I due filamenti possono essere allontanati tra loro, come avviene per unacerniera,sia dalle altetemperaturesia da un'azione meccanica (come avviene durante lareplicazione del DNA).[36]Conseguenza di questa complementarità è che tutte le informazioni contenute nella doppia elica possono essere duplicate a partire da entrambi i filamenti, evento fondamentale per una corretta replicazione del DNA.[23]
I due tipi di paia di basi formano un numero differente di legami idrogeno: A e T ne formano due, G e C tre. Per tale motivo, la stabilità del legame GC è decisamente maggiore di quello AT. Di conseguenza, la stabilità complessiva di una molecola di DNA è direttamente correlata alla frequenza di GC presenti nella molecola stessa, nonché alla lunghezza dell'elica: una molecola di DNA è dunque tanto più stabile quanto più contiene GC ed è lunga.[37]Un'altra conseguenza di tale evento è il fatto che le regioni di DNA che devono essere separate facilmente contengono un'elevata concentrazione di A e T, come avviene ad esempio per ilPribnow boxdeipromotoribatterici, la cui sequenza è infatti TATAAT.[38]
In laboratorio, la stabilità dell'interazione tra filamenti è misurata attraverso la temperatura necessaria a rompere tutti i legami idrogeno, chiamatatemperatura di melting(oTm). Quando tutti i legami idrogeno sono rotti, i singoli filamenti si separano e possono assumere strutture molto variegate.[39]
La stabilizzazione della doppia elica, in ogni caso, non è dovuta ai soli legami idrogeno, ma anche ainterazioni idrofobichee dipi stacking.[40]
Senso e antisenso
[modifica|modifica wikitesto]
Unasequenza di DNAè definitasensose la sua sequenza è la stessa del relativomRNA.La sequenza posta sul filamento opposto è invece dettaantisenso.Dal momento che leRNA polimerasilavorano producendo una copia complementare, il filamento necessario per latrascrizioneè l'antisenso. Sia nei procarioti sia negli eucarioti vengono prodotte numerose molecole diRNA antisensoa partire dalle sequenzesenso.La funzione di questiRNA non codificantinon è stata ancora completamente chiarita.[41]Si ritiene che gli RNA antisenso possano giocare un ruolo nella regolazione dell'espressione genica.[42]
Esistono alcune sequenze di DNA, sia in procarioti sia in eucarioti (ma soprattutto neiplasmidie neivirus) in cui la differenza tra sequenze senso e antisenso è meno chiara, dal momento che le sequenze di alcuni geni si sovrappongono tra loro.[43]In questi casi, dunque, alcune sequenze rivestono un doppio compito: codificare una proteina se lette in direzione 5'→3′ su un filamento; codificarne un'altra se lette sull'altro (sempre in direzione 5'→3′). Nei batteri, questa sovrapposizione genica è spesso coinvolta nella regolazione della trascrizione,[44]mentre nei virus il fenomeno è dovuto alla necessità di contenere in un piccologenomaun'elevata quantità di informazioni.[45]Un altro modo di ridurre le dimensioni genomiche è quello individuato da altri virus, che contengono molecole di DNA lineare o circolare a singolo filamento.[46][47]
Superavvolgimento
[modifica|modifica wikitesto]Il DNA può essere distorto come avviene per una corda attraverso un processo definitosuperavvolgimento.Quando il DNA è in uno statorilassato,un filamento percorre un giro completo intorno all'asse ogni 10,4 paia di basi. Se invece il DNA è distorto, il numero di basi può aumentare o diminuire.[48]Lo stato di superavvolgimento in cui si trova una molecola di DNA è definitotopologia.Se il DNA si avvolge nella direzione dell'elica, si parla disuperavvolgimento positivo,con le basi strette tra loro in modo più marcato. In caso contrario, si parla disuperavvolgimento negativo.In natura, la maggior parte delle molecole di DNA presentano un lieve superavvolgimento negativo, introdotto da enzimi definititopoisomerasi.[49]Questi enzimi sono anche necessari in processi come la trascrizione e la replicazione del DNA, dal momento che sono in grado di risolvere gli stress topologici indotti dai processi stessi.[50]
Strutture alternative della doppia elica
[modifica|modifica wikitesto]
Il DNA esiste in diversi tipi diconformazioni.Esse sono denominate A-DNA, B-DNA, C-DNA, D-DNA,[51]E-DNA,[52]H-DNA,[53]L-DNA,[51]P-DNA[54]e Z-DNA.[29][55]In ogni caso, solo le conformazioni A-DNA, B-DNA e Z-DNA sono state osservate nei sistemi biologici naturali. La conformazione del DNA può dipendere dalla sequenza, dal superavvolgimento, dalla presenza di modificazioni chimiche delle basi o dalle condizioni del solvente, come la concentrazione diionimetallici.[56]Di tali conformazioni, la conformazioneBè la più frequente nelle condizioni standard delle cellule.[57]Le due conformazioni alternative sono differenti dal punto di vista dellageometriae delle dimensioni.
La formaAè un'ampia spirale destrorsa (il solco minore è largo ma poco profondo, quello maggiore è più stretto e profondo), con un passo di 2,9 nm (circa 11bp) e un diametro di 2,5 nm. Tale conformazione è presente in condizioni non fisiologiche, quando il DNA viene disidratato. In condizioni fisiologiche, questa conformazione caratterizza glieteroduplexdi DNA e RNA e i complessi formati dalle associazioni DNA-proteina.[58][59]
La conformazioneZè tipica invece delle sequenze che presentano modificazioni chimiche come lametilazione,e dei tratti di DNA ricchi di basi C e G. Essa assume un andamento sinistrorso, opposto rispetto alla conformazione B.[60]Ha un passo di 4,6 nm e un diametro di 1,8 nm, il solco maggiore più superficiale e quello minore più stretto; deve il suo nome all'andamento a zig-zag che la caratterizza. Queste strutture inusuali possono essere riconosciute da specifiche Z-DNA-binding proteins,con conseguenze notevoli nella regolazione della trascrizione, anche se non ne sono stati trovati esempi in natura.[61]
Strutture alternative alla doppia elica
[modifica|modifica wikitesto]
Le regioni terminali deicromosomilineari sono sequenze ripetute dettetelomeri.La funzione principale di tali regioni è quella di permettere alla cellula di replicare le estremità dei cromosomi senza che ci sia perdita di informazioni geniche, dal momento che leDNA polimerasicoinvolte nella replicazione del DNA non sono in grado di replicare le estremità 3' dei cromosomi.[63]Se un cromosoma non avesse telomeri, infatti, diventerebbe un po' più corto a ogni replicazione, con il rischio di perdere sequenze codificanti. Attraverso un particolare tipo di DNA polimerasi (dettotelomerasi), invece, i telomeri mantengono costantemente la loro lunghezza, proteggendo così la parte interna del cromosoma. Nelle cellule umane, i telomeri sono composti da alcune migliaia di ripetizioni di una semplice sequenza costituita da TTAGGG.[64]
Questa sequenza ricca in guanina può stabilizzare le estremità dei cromosomi formando strutture insolite, composte da unità di quattro basi azotate al posto delle canoniche due. Ciò è dovuto all'interazione tra quattro guanine, che formano una struttura planare che si impila sopra ad altre strutture dello stesso tipo, a ottenere un filamento stabile definitoG-quadruplexstructure.[65]Tali strutture sono stabilizzate dalla formazione di legami idrogeno che si instaurano tra le sommità delle basi e dallachelazionecon uno ione metallico, situato al centro di ogni unità di quattro basi.[66]
Oltre a queste, i telomeri generano anche strutture circolari, chiamatetelomere-loopsoT-loops.In questo caso, il singolo filamento di DNA si piega a formare ampie circonferenze, stabilizzate da proteine specifiche che legano i telomeri.[67]Al termine delT-loop,il singolo filamento di DNA prende contatto con un doppio filamento, che si apre e forma una struttura atripla elica.Questa struttura è chiamatadisplacement-loopoD-loop.[65]
Modificazioni chimiche
[modifica|modifica wikitesto]Modificazioni di basi
[modifica|modifica wikitesto]L'espressione genicadi un determinatolocusè influenzata dalla struttura che lacromatinaassume presso il locus stesso. Regionieterocromatiniche(caratterizzate da un'espressione scarsa o assente) sono estesamentemetilatesullecitosine.La metilazione della citosina, ad esempio, è fondamentale per l'inattivazione del cromosoma X.[68]Il livello medio di metilazione è molto variabile tra i diversi organismi:Caenorhabditis elegansnon presenta metilazione delle citosine, mentre ivertebratimostrano livelli maggiori, con circa l'1% delgenomacontenente 5-metilcitosina.[69]La 5-metilcitosina, essendo suscettibile di deaminazione spontanea, è una base presso cui l'incidenza di mutazioni è elevatissima.[70]
Ulteriori modificazioni di basi sono la metilazione dell'adenina (presente nei batteri) e laglicosilazionedell'uracile che produce le cosiddettebasi Jneicinetoplastidi.[71][72]
Danni al DNA
[modifica|modifica wikitesto]
Il DNA può essere alterato dall'azione di numerosi agenti, genericamente definitimutageni;è fondamentale notare però come una mutazione -ovverosia un cambiamento raro, casuale, che alteri la sequenza di basi azotate- non sia necessariamente un evento pernicioso ma anzi sia alla base dell'evoluzione: suddetta mutazione dovrà però farsi spazio nella fittissima rete cibernetica cellulare nonché nell'ambiente nel quale vive e opera l'organismo vivente in questione; qualora vengano superati questi punti di restrizione (altamente selettivi vista la loro complessità intrinseca, la stragrande maggioranza delle mutazioni difatti si rivela non vantaggiosa o anche neutra), si avrà un organismo arricchito dalla mutazione. Tra gli agenti alteranti figurano ad esempioagenti ossidanti,agenti alchilantie ancheradiazioniad alta energia, come iraggi Xe gliUV.
Il tipo di danno causato al DNA dipende dal tipo di agente: gli UV, ad esempio, danneggiano il DNA generando la formazione didimeri di timina,costituiti da ponti aberranti che si instaurano tra basi pirimidiniche adiacenti.[74]Agenti ossidanti come iradicali liberio ilperossido di idrogeno,invece, producono danni di tipo più eterogeneo, come modificazioni di basi (in particolare di guanine) o rotture del DNA a doppio filamento.[75]Secondo diversi studi, in ogni cellula umana almeno 500 basi al giorno sono sottoposte a danni ossidativi.[76][77]Di tali lesioni, le più pericolose sono le rotture a doppio filamento, dal momento che tali danni sono i più difficili da riparare e costituiscono l'origine primaria dellemutazioni puntiformieframeshiftche si accumulano sulle sequenze genomiche, nonché delletraslocazionicromosomiche.[78]
Molti agenti devono il loro potere mutageno alla capacità diintercalarsitra due basi azotate consecutive. Gli intercalanti sono tipicamente molecole planari earomatiche,come l'etidio,ladaunomicina,ladoxorubicinao latalidomide.Perché un intercalante possa trovare posto tra le due basi, occorre che la doppia elica si apra e perda la sua conformazione standard. Tali modifiche strutturali inibiscono sia la trascrizione sia la replicazione del DNA e aumentano la possibilità di insorgenza di mutazioni. Per tale motivo, gli intercalanti sono considerati molecolecancerogene,come dimostrato da numerosi studi su molecole come ilbenzopirene,l'acridina,l'aflatossinae ilbromuro di etidio.[79][80][81]In ogni caso, proprio grazie alla loro capacità di inibire trascrizione e replicazione, tali molecole sono anche utilizzate inchemioterapiaper inibire la rapida crescita delle cellule neoplastiche.[82]
Localizzazione del DNA
[modifica|modifica wikitesto]Neglieucarioti,il DNA è solitamente presente all'interno dicromosomilineari (circolari neiprocarioti). La somma di tutti i cromosomi di una cellula ne costituisce ilgenoma;ilgenoma umanoconta circa 3 miliardi dipaia di basicontenute in 46 cromosomi.[83]
La disposizione finale a cromosomi segue precise regole gerarchiche diimpacchettamento.Nelle cellule, infatti, il doppio filamento di DNA non può essere dispostoa casaccio,ma deve seguire precise regole di ordinamento. Tali accorgimenti si rivelano necessari perché la lunghezza dei filamenti di DNA è solitamente molto elevata e creerebbe seri problemi alla cellula ospite. Ad esempio, il cromosoma diEscherichia coli,il procariote più studiato nella storia della biomedicina, misura circa 1 mm. In una cellula lunga solo 2 µm, come quella di E. coli, la disposizione casuale di un cromosoma del genere potrebbe generare problemi. Se una molecola di questa lunghezza si disponesse casualmente, infatti, ci sarebbe bisogno di una cellula grande almeno1000volte tanto. Le modalità diimpacchettamentosono differenti tra gli organismiprocariotie quellieucarioti.
Procarioti
[modifica|modifica wikitesto]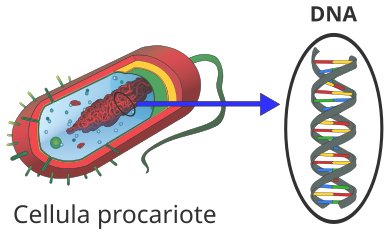
Nella maggior parte delle cellule batteriche il DNA è disposto su un unico cromosoma circolare (e presenta, come molti altri batteri, un'unicaorigine di replicazione), come previsto da numerosi esperimenti di linkage e infine evidenziato in cellule cresciute contiminamarcata contritio.
I meccanismi messi in atto dalla cellula procariote per ridurre lo spazio necessario consistono anzitutto nelmascheramentodelle cariche negative presenti sul DNA attraverso la sua associazione conpoliamminecariche positivamente, come lasperminae laspermidina.Oltre a queste, il DNA procariote prende contatto anche con numerose piccole proteine, che compattano la struttura complessiva del DNA. Tra di esse, figuraH-NS,un dimero con funzioni molto simili agli istoni eucariotici. In ogni cellula di E. coli esistono in media20000molecole di H-NS, che si dispongono lungo il DNA a distanza di circa 400 bp.
Il DNA di E. coli è inoltre moltosuperavvolto.Tale fenomeno contribuisce ulteriormente al compattamento del DNA, permettendo a esso di disporsi comodamente all'interno della cellula.
Eucarioti
[modifica|modifica wikitesto]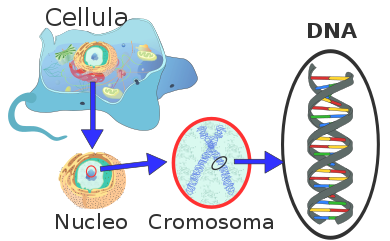
Negli eucarioti l'impacchettamento è ottenuto attraverso diversi accorgimenti. Il DNA è associato a un gran numero di proteine: l'associazione complessiva DNA-proteine è definitacromatina,la cui struttura è ampiamente conservata tra tutti gli organismi eucarioti.
Le proteine cromatiniche più abbondanti sono gliistoni,una famiglia di polipeptidibasicipresenti nel nucleo. Le principali proteine istoniche sonoH1,H2A,H2B,H3e H4. La basicità degli istoni è dovuta alla grande quantità di amminoacidi carichi positivamente (lisinaearginina), in grado di instaurare interazioni elettrostatiche con i gruppi fosfato del DNA. Le proteine istoniche sono anche pesantemente modificate, proprio sui residui carichi, damodificazioni post-traduzionali,tra cui l'aggiunta diacetili,difosfatie dimetili,che neutralizzano la carica positiva o la rendono negativa.
Le sequenze amminoacidiche di quattro dei cinque istoni (H2A, H2B, H3 e H4) sono altamente conservate, anche tra specie molto diverse. La sequenza di H1 presenta invece maggiori variazioni lungo l'evoluzione: in alcuni organismi, H1 non è nemmeno presente in tutti i tessuti (ad esempio neglieritrocitidegliuccelliH1 è sostituita da un sesto istone, chiamato H5). La presenza di differenti H1, in ogni caso, non modifica sostanzialmente la struttura complessiva dell'apparato istonico (definitonucleosoma), che resta ampiamente conservato nell'architettura nella quasi totalità degli eucarioti.
Funzioni biologiche
[modifica|modifica wikitesto]Nel genoma, l'informazione è conservata in sequenze di DNA chiamategeni.La trasmissione dell'informazione contenuta nei geni è garantita dalla presenza di sequenze di basi azotate complementari. Infatti, durante latrascrizione,l'informazione può essere facilmente copiata in un filamento complementare di RNA. Solitamente, tale copia di RNA è utilizzata per sintetizzare una proteina, attraverso un processo definitotraduzione(o sintesi proteica). In alternativa, una cellula può semplicemente duplicare l'informazione genetica attraverso un processo definitoreplicazione del DNA.
Struttura del genoma
[modifica|modifica wikitesto]Negli organismi eucarioti, il DNA genomico è localizzato all'interno del nucleo cellulare, nonché in piccole quantità all'interno dimitocondriecloroplasti.Nei procarioti, il DNA è invece racchiuso in un organello irregolare, privo di membrana, contenuto nel citoplasma, chiamatonucleoide.[84]L'informazione è contenuta all'interno dei geni, unitàereditariein grado di influire sulfenotipodell'organismo. Ogni gene contiene unopen reading frame(regione in grado di essere trascritta a RNA) e una regione regolatoria, costituita sia da unpromotoresia daenhancers.
In moltespecie,solo una piccola frazione della sequenza totale di un genoma può essere trascritta e tradotta. Ad esempio, solo l'1,5% del genoma umano è costituito daesonicodificanti una proteina, mentre più del 50% consiste disequenze ripetutediDNA non codificante.[85]La ragione per cui ci sia una tale quantità di DNA non codificante non è tuttora completamente chiara ed è stata definita comeEnigma del C-value.[86]In ogni caso, le sequenze di DNA che non codificano una proteina possono essere trascritte inRNA non codificante,coinvolto nella regolazione dell'espressione genica.[87]

Alcune sequenze non codificanti ricoprono un ruolo strutturale per i cromosomi. Le regionitelomericheecentromerichecontengono solitamente pochissimi geni, ma sono necessarie per la funzione e la stabilità dei cromosomi.[89]Nell'uomo, grandi quantità di DNA non codificante si ritrovano neglipseudogeni,copie di geni rese inattive dalla presenza di una mutazione.[90]Queste sequenze sono considerate comefossilimolecolari, anche se esistono evidenze secondo le quali si può ipotizzare che siano una sorta dimateriale grezzonecessario per la creazione di nuovi geni attraverso i processi diduplicazione genicae dievoluzione divergente.[91]
Trascrizione e traduzione
[modifica|modifica wikitesto]Un gene è una sequenza di DNA che contiene le informazioni in grado di influire sulle caratteristiche delfenotipodell'organismo. All'interno di un gene, la sequenza di basi di DNA è utilizzata come stampo per la sintesi di una molecola di RNA che, nella maggior parte dei casi, è tradotta in una molecola peptidica.
Il meccanismo attraverso il quale la sequenza nucleotidica di un gene è copiata in un filamento di RNA è dettotrascrizionee avviene per mezzo dell'enzimaRNA polimerasi.Il filamento di RNA può andare incontro a destini differenti: alcune molecole di RNA hanno funzioni di tipo strutturale (come quelle che si trovano all'interno delribosoma) o catalitica (molecole note comeribozimi); la funzione più nota è tuttavia quella dellatraduzione in proteinetramite la produzionemRNA.Il processo di traduzione avviene nel citoplasma, dove gli mRNA si associano ai ribosomi, ed è mediato dalcodice genetico.Il ribosoma permette la lettura sequenziale dei codoni del mRNA, favorendone il riconoscimento e l'interazione con specificitRNA,molecole che trasportano gli amminoacidi corrispondenti a ogni singolo codone.
Il codice genetico
[modifica|modifica wikitesto]Ilcodice geneticoconsiste diparoledi tre lettere chiamatecodoni,costituite dalla sequenza di tre nucleotidi (ad esempio ACU, CAG, UUU), presenti sull'mRNA,ognuna delle quali è associata a un particolare amminoacido. Ad esempio la timina ripetuta in una serie di tre (UUU) codifica lafenilalanina.Utilizzando gruppi di tre lettere si possono avere fino a 64 combinazioni diverse (), in grado di codificare i venti diversi amminoacidi esistenti. Poiché esistono 64 triplette possibili e solo 20 amminoacidi, ilcodice geneticoè dettoridondante(odegenere): alcuni amminoacidi possono infatti essere codificati da più triplette diverse. Non è invece vero il contrario: a ogni tripletta corrisponderà un solo amminoacido (senza possibilità di ambiguità). Esistono infine tre triplette che non codificano alcun amminoacido, ma rappresentano codoni distop(ononsense), ovvero indicano il punto in cui, all'interno del gene, termina la sequenza che codifica la proteina corrispondente: si tratta dei codoni UAA, UGA e UAG.
Replicazione
[modifica|modifica wikitesto]
La divisione cellulare, necessaria a un organismo per crescere e vivere, richiede una duplicazione del DNA cellulare, in modo che le cellule figlie possano avere la stessa informazione genetica della cellula madre. La struttura a doppia elica del DNA permette un meccanismo estremamente semplice per la replicazione del DNA. I due filamenti, infatti, sono separati e da ognuno viene creato un filamento complementare, per opera di un enzima chiamatoDNA polimerasi.Con questo meccanismo, le basi presenti sul filamento figlio sono determinate da quelle presenti sul filamento parentale: è proprio attraverso questo meccanismo che le cellule figlie presentano genoma identico alla cellula madre (salvo errori avvenuti durante il processo, che portano alla comparsa di mutazioni). Tale tipo di replicazione, che porta a doppie eliche costituite da un filamento preesistente e uno neoformato è dettasemiconservativa.
Per iniziare la replicazione, occorre anzitutto l'apertura dellaforca replicativa,attraverso la parziale denaturazione del DNA a doppia elica, portata a termine dalle elicasi e dallesingle-strand-binding proteins(SSBPs): leelicasisono enzimi che separano attivamente i due filamenti usando l'energia dell'ATP;le SSBPs sono in grado di mantenere la denaturazione del DNA legandosi esclusivamente alle porzioni a singolo filamento e stabilizzandole. Nelle molecole di DNA circolari deiprocariotisi ha una sola regione diorigine della replicazionedalla quale partono due forche replicative (la struttura prende il nome dibolla di replicazione). Quando le due forche si incontrano dal lato opposto la replicazione è completata. Neglieucariotila replicazione di ogni cromosoma inizia invece in più punti.
LeDNA polimerasi,enzimi capaci di costruire una nuova catena solo in direzione 5'-3', sono stati individuati per la prima volta daArthur Kornberg,il quale, grazie a un suo famoso esperimento,[92]identificò la DNA polimerasi I inEscherichia coli.La reazione della DNA polimerasi èdiretta dallo stampo,perché produce un nuovo filamento di DNA esattamente complementare a uno preesistente che funge, appunto, da stampo. La DNA polimerasi non è in grado di iniziare la sintesi di un filamentoex novo,mentre può allungare un filamento polinucleotidico preesistente. In una cellula in replicazione, dunque, è indispensabile la presenza di un filamento preesistente (dettoprimer), che consiste solitamente in un breve segmento di RNA complementare allo stampo, sintetizzato da enzimi specifici dettiprimasi.
Dal momento che le DNA polimerasi sono in grado di svolgere la loro attività solo in direzione 5'-3', esse hanno messo a punto diversi meccanismi per copiare i due filamenti della doppia elica.[93]Un filamento (chiamatofilamento guida) può essere replicato in modo quasi continuo, man mano che viene esposto, l'altro (filamento lento) risulta invece disseminato da brevi filamenti di DNA di nuova sintesi (iframmenti di Okazaki), ognuno dei quali presenta un innesco iniziale di RNA. I nuovi filamenti devono essere quindi completati mediante la rimozione degli inneschi da parte diendonucleasie il riempimento degli spazi rimasti per opera di polimerasi diriparazione.Successivamente tutti questi frammenti di DNA di nuova sintesi delfilamento lentovengono legati dalleDNA ligasi.
Interazioni con proteine
[modifica|modifica wikitesto]Tutte le funzioni del DNA dipendono dalle sue interazioni con specifiche proteine. Tali interazioni possono sia essere aspecifiche, sia prevedere un legame estremamente specifico della proteina a una singola sequenza di DNA. Sono numerosi anche gli enzimi che possono legare il DNA e, tra questi, sono particolarmente importanti le polimerasi che copiano le sequenze nella trascrizione e nella replicazione del DNA.
Proteine che legano il DNA
[modifica|modifica wikitesto]Le proteine strutturali che legano il DNA sono esempi delle interazioni aspecifiche tra DNA e proteine. All'interno dei cromosomi, il DNA è associato a complessi di natura proteica, che si organizzano tra loro a formare una struttura compatta chiamatacromatina.Negli eucarioti, questa struttura presuppone il legame del DNA a piccoli complessi proteici basici chiamatiistoni;nei procarioti, invece, sono coinvolti diversi tipi di differenti proteine.[94][95]Gli istoni formano un complesso a forma di disco chiamatonucleosoma,che instaura interazioni di tipo ionico (tra i residui basici degli istoni e lo scheletro fosforico acido del DNA) con circa duecento paia di basi di DNA, che si avvolgono intorno al disco formando due giri completi, indipendentemente dalla sequenza che li caratterizza.[96]Questi residui basici possono subiremetilazioni,fosforilazionieacetilazioni:[97]tali modificazioni chimiche alterano l'interazione tra gli istoni e il DNA, rendendolo così più o meno accessibile aifattori di trascrizionee modulando la velocità della trascrizione stessa.[98]
AltreDNA-binding proteins(DNAbp) di tipo aspecifico, anch'esse presenti nellacromatina,sono lehigh-mobility group proteins,che legano preferenzialmente il DNA ripiegato o distorto.[99]Queste proteine hanno un ruolo fondamentale nel ripiegamento delle file di nucleosomi e nel loro impacchettamento all'interno di strutture cromatiniche più complesse.[100]
Un ulteriore gruppo di DNAbp sono lesingle-strand-binding proteins(SSBP), che si legano esclusivamente a una molecola di DNA a singolo filamento. Nell'uomo, la RPA (replication protein A) è il membro meglio caratterizzato di questa famiglia ed è essenziale per la maggior parte dei processi che richiedono una separazione della doppia elica, tra cui la replicazione del DNA, la sua ricombinazione e la suariparazione.[101]Queste DNAbp sono in grado di stabilizzare la forma a singolo filamento, impedendo che la molecola si ripieghi a formarestem loopso venga degradata dall'azione dellenucleasi.

A differenza di quelle finora presentate, esistono anche numerose proteine che legano specificamente determinate sequenze di DNA. Quelle maggiormente studiate sono ifattori di trascrizione(TF), proteine in grado di regolare la trascrizione. Ognuna di esse si lega a una o più sequenze specifiche, poste solitamente nei pressi delpromotoredi un gene, attivando o inibendo la trascrizione del gene stesso. Tale processo viene svolto attraverso due tipi di meccanismo: anzitutto, i TF sono in grado di legare, direttamente o attraverso proteine adattatrici, la RNA polimerasi responsabile della trascrizione, localizzandola presso il sito di inizio della trascrizione e favorendone dunque l'avvio;[103]un'altra modalità consiste nel legame tra i TF ed enzimi in grado di modulare metilazioni e acetilazioni degli istoni presenti presso il promotore, modificando dunque l'accessibilità di quella regione alla polimerasi.[104]
Dal momento che un TF può avere numerose sequenze bersaglio, cambiamenti di attività di un TF possono avere effetti sull'espressione di migliaia di geni.[105]Di conseguenza, queste proteine sono spesso i bersagli finali delle cascate ditrasduzione del segnale,che mediano le risposte cellulari agli stimoli interni ed esterni alla cellula. La specificità dei TF per il DNA è legato ai contatti multipli che si instaurano tra la proteina e il solco maggiore, dove le basi azotate sono maggiormente accessibili.[34]
Enzimi che modificano il DNA
[modifica|modifica wikitesto]Nucleasi e ligasi
[modifica|modifica wikitesto]
Lenucleasisonoenzimiin grado di tagliare filamenti di DNA, dal momento che catalizzano l'idrolisidellegame fosfodiesterico.Le nucleasi che idrolizzano il DNA partendo dai nucleotidi situati alle estremità dei filamenti sono definiteesonucleasi.Sonoendonucleasi,invece, quelle che tagliano direttamente all'interno del filamento. Le nucleasi più utilizzate inbiologia molecolare,detteenzimi di restrizione,tagliano il DNA in corrispondenza di specifiche sequenze. L'enzimaEcoRV,ad esempio, riconosce la sequenza di sei basi 5′-GAT|ATC-3′ ed effettua il taglio presso la linea verticale. In natura, questo enzima protegge ibatteridalle infezionifagiche,digerendo il DNA del fago quando esso fa il suo ingresso nella cellula batterica.[107]Generalmente, le nucleasi di restrizione riconoscono particolari sequenze nucleotidichepalindromiche,note comesiti di restrizione,nelle quali la stessa sequenza si ripete in direzioni opposte sulle due eliche complementari, e quindi producono dei tagli su entrambi i filamenti. Tali enzimi sono utilizzati ampiamente nelle tecniche che prevedono ilsubclonaggiodi DNA all'interno divettori.Si crede che le sequenze palindromiche possano essere, oltre a siti di restrizione, anche segnali di riconoscimento per alcune proteine di regolazione oppure segnalare il sito di avvio della replicazione del DNA.
LeDNA ligasisono enzimi in grado di riunire filamenti di DNA precedentemente tagliati o spezzati, utilizzando energia chimica proveniente daATPo daNAD.[108]Le ligasi sono particolarmente importanti nella replicazione delfilamento lento,dal momento che esse riuniscono iframmenti di Okazakiin un filamento unico. Esse rivestono un ruolo importante anche nellariparazione del DNAe nellaricombinazione genetica.[108]
Topoisomerasi ed elicasi
[modifica|modifica wikitesto]Letopoisomerasisono enzimi che presentano sia un'attivitànucleasicasia unaligasica.Queste proteine sono in grado di modificare le proprietàtopologichedel DNA. Alcune di esse svolgono tale funzione tagliando l'elica di DNA e permettendole di ruotare, riducendo il suo grado di superavvolgimento, per poi procedere alla ligazione delle due estremità.[49]Altre topoisomerasi sono invece in grado di tagliare l'elica e far passare attraverso il sito di rottura una seconda elica, prima di ligare il filamento spezzato.[109]Le topoisomerasi sono necessarie per molti processi che coinvolgono il DNA, come ad esempio la replicazione del DNA e la trascrizione.[50]
Leelicasisono proteine in grado di utilizzare l'energia chimicapresente neinucleosidi trifosfato,soprattuttoATP,per rompere i legami idrogeno che si instaurano tra le basi azotate, permettendo l'apertura della doppia elica di DNA in singoli filamenti.[110]Questi enzimi sono essenziali per la maggior parte dei processi biologici che coinvolgono enzimi che richiedono un diretto contatto con le basi del DNA.
Polimerasi
[modifica|modifica wikitesto]Lepolimerasisono enzimi che sintetizzano catene polinucleotidiche a partire dalnucleoside trifosfato.Esse funzionano aggiungendo nucleotidi al 3′-OHdel precedente nucleotide presente sul filamento. Come conseguenza di ciò, tutte le polimerasi lavorano in direzione5′-3′.[111]Nel sito attivo di questi enzimi, il nucleoside trifosfato si appaia a un nucleotide presente su un filamento usato come stampo: ciò permette alle polimerasi di sintetizzare in modo accurato filamenti fedelmente complementari agli stampi. Le polimerasi sono classificate sulla base del tipo di stampo che utilizzano.
Lareplicazione del DNArichiede unaDNA polimerasi DNA-dipendente,in grado cioè di realizzare una perfetta copia di una sequenza di DNA. L'accuratezza è fondamentale in questo processo, motivo per cui molte di queste polimerasi presentano anche un'attività diproofreading(dall'inglese,correzione di bozze). Esse sono infatti in grado di rilevare un errore di appaiamento (omismatch) tra basi azotate e attivare un'azione 3' o 5' esonucleasica per rimuovere la base scorretta.[112]Nella maggior parte degli organismi, le DNA polimerasi funzionano all'interno di un più ampio complesso proteico definitoreplisoma,che consiste anche di numerose subunità accessorie come ad esempio leelicasi.[113]
LeDNA polimerasi RNA-dipendentisono una classe di polimerasi specializzate nella sintesi di una copia di DNA, usando come stampo un frammento di RNA. Tra di esse figurano latrascrittasi inversa,un enzima virale coinvolto nell'infezione deiretrovirus,e latelomerasi,necessaria per la replicazione deitelomeri.[63][114]La telomerasi è una polimerasi inusuale, dal momento che contiene una sequenza stampo di RNA all'interno della propria struttura.
La trascrizione è invece svolta daRNA polimerasi DNA-dipendenti,che legano il DNA presso ilpromotoredi un gene e separano i due filamenti. Successivamente, l'enzima genera una molecola dimRNAfino al raggiungimento delterminatore,dove si interrompe la trascrizione e l'enzima si distacca dal DNA. Come avviene per le DNA polimerasi DNA-dipendenti, anche queste polimerasi operano all'interno di un ampio complesso proteico, composto di molecole accessorie e regolatorie.[115]
Ricombinazione genetica
[modifica|modifica wikitesto]

|

|
Un filamento di DNA solitamente non interagisce con altri segmenti di DNA e, nelle cellule umane, i differenti cromosomi occupano addirittura regioni separate del nucleo (territori cromosomici).[117]Tale separazione fisica è fondamentale per permettere al DNA di essere un archivio stabile e sicuro dell'informazione genetica. L'interazione tra diversi segmenti di DNA è invece possibile e frequente attraverso il fenomeno delcrossing-over,che permette laricombinazione geneticaattraverso la rottura di due eliche, lo scambio di segmenti tra di esse e il ricongiungimento finale.
La ricombinazione permette ai cromosomi di scambiare informazioni genetiche e produrre nuove combinazioni di geni, con il risultato di aumentare l'efficienza dellaselezione naturalee di facilitare l'evoluzione di nuove proteine.[118]La ricombinazione genetica può anche essere coinvolta nella riparazione del DNA, in particolare come risposta cellulare in seguito a rotture a doppio filamento.[119]
La principale forma di crossing-over cromosomico è laricombinazione omologa,nella quale i due cromosomi coinvolti condividono sequenze molto simili. Le ricombinazioni non omologhe, invece, possono essere dannose per la cellula, perché in grado di produrretraslocazioni cromosomichee anomalie genetiche. La reazione di ricombinazione è catalizzata da enzimi noti comericombinasi.[120]Il primo passaggio del processo di ricombinazione consiste nella rottura a singolo filamento provocata da un'endonucleasi o da un danno al DNA.[121]Una serie di passaggi successivi, in parte catalizzati dalla ricombinasi, porta all'unione tra due eliche attraverso la formazione di unagiunzione di Holliday,nella quale un segmento a singolo filamento di ogni elica è appaiato al filamento complementare presente sull'altra elica. La reazione di ricombinazione è quindi interrotta dalla rottura della giunzione e dalla re-ligazione del DNA così ottenuto.[122]L'esistenza della giunzione di Holliday è stata dimostrata da fotografie al microscopio elettronico di molecole di DNA in ricombinazione.
Evoluzione del metabolismo del DNA
[modifica|modifica wikitesto]Il DNA contiene l'informazione genetica che permette a tutti gli organismi viventi (esclusi i virus, la cui ammissibilità tra i viventi è tuttavia ampiamente dibattuta) di funzionare, crescere e riprodursi. In ogni caso, non è stato ancora chiarito in quale momento dellastoria della vitail DNA abbia assunto tale ruolo fondamentale. È generalmente accettato dalla comunità scientifica, infatti, l'ipotesiche il DNA non sia stato il primo acido nucleico a essere utilizzato dai viventi: tale ruolo spetterebbe infatti all'RNA.[123][124]L'RNA potrebbe aver giocato un ruolo centrale del metabolismo cellulare ancestrale, dal momento che può avere sia un ruolo nella conservazione dell'informazione genetica, sia uno catalitico (ad esempio attraverso iribozimi), sia uno strutturale (all'interno deiribosomi).[125]Ilmondo a RNA,basato su un unico tipo di molecola avente funzioni genetiche, catalitiche e strutturali, potrebbe avere avuto un'influenza sullo sviluppo dell'attuale codice genetico, basato proprio su quattro nucleotidi. Tale numero potrebbe essere un compromesso tra la necessità da una parte di ridurre la quantità di basi possibili, per migliorare l'accuratezza della replicazione, e dall'altra di aumentarla, per incrementare l'efficacia catalitica dei ribozimi.[126]
Non si conoscono fossili di DNA risalenti all'origine della vita (dopo il mondo a RNA) per poterne studiare l'evoluzione molecolare, dal momento che è impossibile recuperare DNA dai fossili. Ciò è dovuto al fatto che il DNA può sopravvivere nell'ambiente per meno di un milione di anni e, se in soluzione, si degrada rapidamente in piccoli frammenti.[127]Sebbene ci siano stati diversi annunci di scoperte di DNA antichissimo, tra cui quella relativa all'isolamento di un batterio vivo da un cristallo di sale risalente a 250 milioni di anni fa,[128]queste affermazioni sono controverse.[129][130]
Utilizzi del DNA
[modifica|modifica wikitesto]Ingegneria genetica
[modifica|modifica wikitesto]La moderna biologia e biochimica fa un uso intensivo del DNA. Con il termine diDNA ricombinanteci si riferisce a segmenti di DNA realizzati e assemblati artificialmente. Essi possono essere inseriti all'interno di organismi viventi sotto forma diplasmidio mediante altri tipi divettori.[131]Gli organismi così prodotti sono dettigeneticamente modificatie possono essere utilizzati per la produzione di proteine ricombinanti, necessarie per la ricerca biomedica,[132]o per le coltivazioniagricole.[133][134]
Medicina forense
[modifica|modifica wikitesto]Lamedicina forensesi serve del DNA, generalmente isolato dalsangue,dallapelle,dallasaliva,daicapellie da altri tessuti e fluidi biologici, per identificare i responsabili di atti criminosi, come omicidi o violenze. Il processo utilizzato è ilfingerprinting genetico:tale tecnica consiste nel comparare la lunghezza delle sezioni variabili delDNA ripetitivo,come leshort tandem repeatse iminisatelliti,che possono risultare molto diverse tra un individuo e l'altro. La comparazione tra due campioni di DNA in esame, non si basa perciò sull'analisi di tutta la sequenza desossiribonucleotidica, ma solo su tali sezioni. Infatti, due individui non legati da rapporti di parentela hanno in comune ben il 99,9% di sequenza di DNA. Tale metodo è solitamente molto affidabile,[135]anche se a volte l'identificazione dei criminali può risultare complicata qualora la scena sia contaminata dal DNA di diverse persone.[136]Questo metodo, sviluppato nel 1984 dalgenetistabritannicoSirAlec Jeffreys,[137]fu usato per la prima volta nel 1988 per incriminare Colin Pitchfork. Nella pratica attuale, spesso i sospettati sono invitati a fornire un campione di DNA per il confronto con eventuali reperti biologici presenti sulla scena del delitto. Il fingerprinting genetico può essere utilizzato anche per identificare le vittime di incidenti di massa.
L'acquisizione del DNA senza consenso viene indicato con il termine digene theft.
Bioinformatica
[modifica|modifica wikitesto]Labioinformaticaè una branca dellabiologiache comprende la manipolazione, la ricerca e ildata miningdei dati relativi a sequenze di DNA. Lo sviluppo di tecniche utili a immagazzinare e ricercare sequenze di DNA, infatti, ha condotto ad ampi sviluppi dell'informatica applicata alla biologia molecolare, specialmente per quanto riguarda gli algoritmi di ricerca di stringhe e l'apprendimento automatico.[138]Gli algoritmi di ricerca (o appaiamento) di stringhe, in grado di individuare la presenza di una sequenza di lettere all'interno di sequenze molto più ampie, furono inizialmente sviluppati per la ricerca di specifiche sequenze nucleotidiche.[139]
Esistono da molto tempo, ovviamente, semplicialgoritmiin grado di affrontare questi problemi (quelli presenti, ad esempio, neglieditor di testo), ma l'analisi del DNA, che si presenta come composto di sole quattro lettere, richiede programmi più elaborati. Il problema immediatamente correlato dell'allineamento di sequenzesi pone come obiettivo quello di identificare le sequenzeomologhee individuare le specifichemutazioniche le rendono differenti. Queste tecniche, in particolare l'allineamento di sequenze multiple,sono utilizzate per studiare lerelazioni filogenetichee la funzione delle proteine.[140]Esistono anche algoritmi di ricerca genetica.
La grande quantità di dati ottenuta da progetti come ilprogetto genoma umanoè infatti di difficile utilizzo senza una prima analisi che permetta di localizzare i geni e le regioni regolatorie sui cromosomi. Tali algoritmi, dunque, sono in grado di riconoscere regioni putative di presenza di geni codificanti RNA o proteine.[141]
DNA in informatica e nanotecnologie
[modifica|modifica wikitesto]
Il DNA è stato utilizzato in informatica per la prima volta per risolvere un semplice problema dicammino hamiltoniano,unproblema NP-completo.[143]Il calcolo attraverso il DNA è più vantaggioso rispetto a quelloclassicoper via elettronica sia dal punto di vista dell'energia consumata, sia da quello dello spazio utilizzato: strutture di questo genere sono infatti in grado di svolgerecalcoli in modalità paralleleche permettono di risolvere agevolmente numerosi altri problemi quali la simulazione dimacchine astratte,ilproblema di soddisfacibilità booleanae la versioneboundeddelproblema del commesso viaggiatore.[144]Grazie alla sua compattezza, il DNA presenta anche un ruolo (almeno teorico) nel campo dellacrittografia,nella quale permetterebbe in particolare la costituzione e l'utilizzo efficiente dicifrari di Vernamsicuri.[145]
Il DNA è utilizzato anche nel campo dellenanotecnologiepoiché presenta proprietà diriconoscimento molecolareche lo rendono in grado di auto-assemblarsi in strutture complesse di tipo bidimensionale opoliedrico.Tali assemblati sono utilizzati con funzioni essenzialmente strutturali e non come vettori di informazione biologica.[146]
Storia e antropologia
[modifica|modifica wikitesto]
Dal momento che il DNA è sottoposto nel tempo a mutazioni che vengono ereditate, esso contiene informazioni preziose che possono essere utilizzate dai genetisti per studiare l'evoluzione degli organismi e la lorofilogenesi.[148]Sulla base delle diverse mutazioni presenti in geni estremamente conservati tra gli organismi (oppure, tramite algoritmi comparativi bioinformatici più avanzati, confrontando direttamente interi genomi) i genetisti sono in grado di ricostruirealberi filogeneticiin grado di descrivere l'evoluzione di diverse specie anche molto diverse tra loro.[149][150]Studiando le mutazioni accumulatesi nel tempo, è anche possibile ricostruire alberi che descrivano l'evoluzione all'interno di famiglie di proteine.
Comparando le sequenze di DNA all'interno di una stessa specie, inoltre, è possibile studiare la storia genetica di particolaripopolazioni.Ciò presenta una notevole rilevanza sia per analisiecologichesia per studiantropologici:il DNA è stato usato, ad esempio, per ricostruire la vicenda delledieci tribù perdute d'Israele.[151]
DNA ambientale eDNA
[modifica|modifica wikitesto]Nel 1987Tamar Barkaydescrisse il metodo diestrazione diretta del DNA ambientale,eDNA[152].
Note
[modifica|modifica wikitesto]- ^ADN,inTreccani.it –Vocabolario Treccanion line,Roma, Istituto dell'Enciclopedia Italiana.URL consultato il 27 aprile 2017.
- ^Lemma "ADN".inTullio De Mauro,Dizionario italiano.
- ^Cfr. "DNA o ADN."nell'enciclopediaSapere.
- ^(EN)Genetics dictionary - DNA,suNational Cancer Institute,20 luglio 2012.URL consultato il 21 ottobre 2023.
- ^Bruce Alberts,Molecular biology of the cell,Sixth edition, Garland Science, Taylor and Francis Group, 2015,ISBN978-0-8153-4432-2.
- ^Dahm R,Friedrich Miescher and the discovery of DNA,inDev Biol,vol. 278, n. 2, 2005, pp. 274–88,PMID15680349.
- ^Levene P,,The structure of yeast nucleic acid,inJ Biol Chem,vol. 40, n. 2, 1919, pp. 415–24.URL consultato il 2 settembre 2007(archiviato dall'url originaleil 29 giugno 2009).
- ^Astbury W,,Nucleic acid,inSymp. SOC. Exp. Bbl,vol. 1, n. 66, 1947.
- ^Erwin Schrödinger -What is Life? The Physical Aspect of the Living Cell,Cambridge University Press, Cambridge 1944
- ^Lorenz MG, Wackernagel W,Bacterial gene transfer by natural genetic transformation in the environment,inMicrobiol. Rev.,vol. 58, n. 3, 1994, pp. 563–602,PMID7968924.
- ^Avery O, MacLeod C, McCarty M,Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Inductions of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III,inJ Exp Med,vol. 79, n. 2, 1944, pp. 137–158.
- ^Hershey A, Chase M,Independent functions of viral protein and nucleic acid in growth of bacteriophage(PDF), inJ Gen Physiol,vol. 36, n. 1, 1952, pp. 39–56,PMID12981234.
- ^abWatson J.D. and Crick F.H.C."A Structure for Deoxyribose Nucleic Acid".(PDF).(PDF)Nature171, 737–738 (1953). Ultimo accesso: 13 febbraio 2007.
- ^abWatson J, Crick F,Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid(PDF), inNature,vol. 171, n. 4356, 1953, pp. 737–8,PMID13054692.
- ^Nature ArchivesDouble Helix of DNA: 50 Years.
- ^Molecular Configuration in Sodium Thymonucleate. Franklin R. and Gosling R.G.Nature 171, 740–741 (1953)Nature Archives Full Text (PDF)(PDF).
- ^Original X-ray diffraction image.
- ^Molecular Structure of Deoxypentose Nucleic Acids. Wilkins M.H.F., A.R. Stokes A.R. & Wilson, H.R. Nature 171, 738–740 (1953)Nature Archives (PDF)(PDF).
- ^The Nobel Prize in Physiology or Medicine 1962.Nobelprize.org. Ultimo accesso: 22 dicembre 2006
- ^Crick, F.H.C.On degenerate templates and the adaptor hypothesis (PDF).(PDF)(archiviato dall'url originaleil 1º ottobre 2008).genome.wellcome.ac.uk (Lecture, 1955). Ultimo accesso: 22 dicembre 2006.
- ^Meselson M, Stahl F,The replication of DNA in Escherichia coli,inProc Natl Acad Sci U S A,vol. 44, n. 7, 1958, pp. 671–82,PMID16590258.
- ^The Nobel Prize in Physiology or Medicine 1968.Nobelprize.org. Ultimo accesso: 22 dicembre 2006.
- ^abBruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walters,Molecular Biology of the Cell; Fourth Edition,New York and London, Garland Science, 2002,ISBN0-8153-3218-1.
- ^Butler, John M. (2001)Forensic DNA Typing"Elsevier". pp. 14–15.ISBN 978-0-12-147951-0.
- ^Mandelkern M, Elias J, Eden D, Crothers D,The dimensions of DNA in solution,inJ Mol Biol,vol. 152, n. 1, 1981, pp. 153–61,PMID7338906.
- ^Gregory S,et al.,The DNA sequence and biological annotation of human chromosome 1,inNature,vol. 441, n. 7091, 2006, pp. 315–21,PMID16710414.
- ^abcBerg J., Tymoczko J. and Stryer L. (2002)Biochemistry.W. H. Freeman and CompanyISBN 0-7167-4955-6.
- ^Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents.IUPAC-IUB Commission on Biochemical Nomenclature (CBN). Ultimo accesso: 3 gennaio 2006.
- ^abGhosh A, Bansal M,A glossary of DNA structures from A to Z,inActa Crystallogr D Biol Crystallogr,vol. 59, Pt 4, 2003, pp. 620–6,PMID12657780.
- ^(EN)IUPAC Gold Book, "deoxyribonucleic acids (DNA)".
- ^Takahashi I, Marmur J.,Replacement of thymidylic acid by deoxyuridylic acid in the deoxyribonucleic acid of a transducing phage for Bacillus subtilis,inNature,vol. 197, 1963, pp. 794–5,PMID13980287.
- ^Agris P,Decoding the genome: a modified view,inNucleic Acids Res,vol. 32, n. 1, 2004, pp. 223–38,PMID14715921.
- ^Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R,Crystal structure analysis of a complete turn of B-DNA,inNature,vol. 287, n. 5784, 1980, pp. 755–8,PMID7432492.
- ^abPabo C, Sauer R,Protein-DNA recognition,inAnnu Rev Biochem,vol. 53, 1984, pp. 293–321,PMID6236744.
- ^Tra le eccezioni a questa regola, si ricordano le proteine condominiodi legame al DNA di tipoHMG-box,qualeHMGB1.
- ^Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H,Mechanical stability of single DNA molecules,inBiophys J,vol. 78, n. 4, 2000, pp. 1997–2007,PMID10733978.
- ^Chalikian T, Völker J, Plum G, Breslauer K,A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques,inProc Natl Acad Sci U S A,vol. 96, n. 14, 1999, pp. 7853–8,PMID10393911.
- ^deHaseth P, Helmann J,Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA,inMol Microbiol,vol. 16, n. 5, 1995, pp. 817–24,PMID7476180.
- ^Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J,Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern,inBiochemistry,vol. 43, n. 51, 2004, pp. 15996–6010,PMID15609994.
- ^Ponnuswamy P, Gromiha M,On the conformational stability of oligonucleotide duplexes and tRNA molecules,inJ Theor Biol,vol. 169, n. 4, 1994, pp. 419–32,PMID7526075.
- ^Hüttenhofer A, Schattner P, Polacek N,Non-coding RNAs: hope or hype?,inTrends Genet,vol. 21, n. 5, 2005, pp. 289–97,PMID15851066.
- ^Munroe S,Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns,inJ Cell Biochem,vol. 93, n. 4, 2004, pp. 664–71,PMID15389973.
- ^Makalowska I, Lin C, Makalowski W,Overlapping genes in vertebrate genomes,inComput Biol Chem,vol. 29, n. 1, 2005, pp. 1–12,PMID15680581.
- ^Johnson Z, Chisholm S,Properties of overlapping genes are conserved across microbial genomes,inGenome Res,vol. 14, n. 11, 2004, pp. 2268–72,PMID15520290.
- ^Lamb R, Horvath C,Diversity of coding strategies in influenza viruses,inTrends Genet,vol. 7, n. 8, 1991, pp. 261–6,PMID1771674.
- ^Davies J, Stanley J,Geminivirus genes and vectors,inTrends Genet,vol. 5, n. 3, 1989, pp. 77–81,PMID2660364.
- ^Berns K,Parvovirus replication,inMicrobiol Rev,vol. 54, n. 3, 1990, pp. 316–29,PMID2215424.
- ^Benham C, Mielke S,DNA mechanics,inAnnu Rev Biomed Eng,vol. 7, 2005, pp. 21–53,PMID16004565.
- ^abChampoux J,DNA topoisomerases: structure, function, and mechanism,inAnnu Rev Biochem,vol. 70, 2001, pp. 369–413,PMID11395412.
- ^abWang J,Cellular roles of DNA topoisomerases: a molecular perspective,inNat Rev Mol Cell Biol,vol. 3, n. 6, 2002, pp. 430–40,PMID12042765.
- ^abHayashi G, Hagihara M, Nakatani K,Application of L-DNA as a molecular tag,inNucleic Acids Symp Ser (Oxf),vol. 49, 2005, pp. 261–262,PMID17150733.
- ^Vargason JM, Eichman BF, Ho PS,The extended and eccentric E-DNA structure induced by cytosine methylation or bromination,inNature Structural Biology,vol. 7, 2000, pp. 758–761,PMID10966645.
- ^Wang G, Vasquez KM,Non-B DNA structure-induced genetic instability,inMutat Res,vol. 598, 1–2, 2006, pp. 103–119,PMID16516932.
- ^Allemand, et al,Stretched and overwound DNA forms a Pauling-like structure with exposed bases,inPNAS,vol. 24, 1998, pp. 14152-14157,PMID9826669.
- ^Palecek E,Local supercoil-stabilized DNA structures,inCritical Reviews in Biochemistry and Molecular Biology,vol. 26, n. 2, 1991, pp. 151–226,PMID1914495.
- ^Basu H, Feuerstein B, Zarling D, Shafer R, Marton L,Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies,inJ Biomol Struct Dyn,vol. 6, n. 2, 1988, pp. 299–309,PMID2482766.
- ^Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL,Polymorphism of DNA double helices,inJ. Mol. Biol.,vol. 143, n. 1, 1980, pp. 49–72,PMID7441761.
- ^Wahl M, Sundaralingam M,Crystal structures of A-DNA duplexes,inBiopolymers,vol. 44, n. 1, 1997, pp. 45–63,PMID9097733.
- ^Lu XJ, Shakked Z, Olson WK,A-form conformational motifs in ligand-bound DNA structures,inJ. Mol. Biol.,vol. 300, n. 4, 2000, pp. 819-40,PMID10891271.
- ^Rothenburg S, Koch-Nolte F, Haag F,DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles,inImmunol Rev,vol. 184, pp. 286–98,PMID12086319.
- ^Oh D, Kim Y, Rich A,Z-DNA-binding proteins can act as potent effectors of gene expression in vivo,inProc. Natl. Acad. Sci. U.S.A.,vol. 99, n. 26, 2002, pp. 16666-71,PMID12486233.
- ^Immagine realizzata daNDB UD0017(archiviato dall'url originaleil 7 giugno 2013).
- ^abGreider C, Blackburn E,Identification of a specific telomere terminal transferase activity in Tetrahymena extracts,inCell,vol. 43, 2 Pt 1, 1985, pp. 405–13,PMID3907856.
- ^Wright W, Tesmer V, Huffman K, Levene S, Shay J,Normal human chromosomes have long G-rich telomeric overhangs at one end,inGenes Dev,vol. 11, n. 21, 1997, pp. 2801–9,PMID9353250.
- ^abBurge S, Parkinson G, Hazel P, Todd A, Neidle S,Quadruplex DNA: sequence, topology and structure,inNucleic Acids Res,vol. 34, n. 19, 2006, pp. 5402–15,PMID17012276.
- ^Parkinson G, Lee M, Neidle S,Crystal structure of parallel quadruplexes from human telomeric DNA,inNature,vol. 417, n. 6891, 2002, pp. 876–80,PMID12050675.
- ^Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T,Mammalian telomeres end in a large duplex loop,inCell,vol. 97, n. 4, 1999, pp. 503–14,PMID10338214.
- ^Klose R, Bird A,Genomic DNA methylation: the mark and its mediators,inTrends Biochem Sci,vol. 31, n. 2, 2006, pp. 89–97,PMID16403636.
- ^Bird A,DNA methylation patterns and epigenetic memory,inGenes Dev,vol. 16, n. 1, 2002, pp. 6–21,PMID11782440.
- ^Walsh C, Xu G,Cytosine methylation and DNA repair,inCurr Top Microbiol Immunol,vol. 301, pp. 283–315,PMID16570853.
- ^Ratel D, Ravanat J, Berger F, Wion D,N6-methyladenine: the other methylated base of DNA,inBioessays,vol. 28, n. 3, 2006, pp. 309–15,PMID16479578.
- ^Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P,beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei,inCell,vol. 75, n. 6, 1993, pp. 1129–36,PMID8261512.
- ^Immagine realizzata a partire dalla entryPDB 1JDG(archiviato dall'url originaleil 22 settembre 2008).
- ^Douki T, Reynaud-Angelin A, Cadet J, Sage E,Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation,inBiochemistry,vol. 42, n. 30, 2003, pp. 9221–6,PMID12885257.
- ^Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S,Hydroxyl radicals and DNA base damage,inMutat Res,vol. 424, 1–2, 1999, pp. 9–21,PMID10064846.
- ^Shigenaga M, Gimeno C, Ames B,Urinary 8-hydroxy-2′-deoxyguanosine as a biological marker ofin vivooxidative DNA damage,inProc Natl Acad Sci U S A,vol. 86, n. 24, 1989, pp. 9697–701,PMID2602371.
- ^Cathcart R, Schwiers E, Saul R, Ames B,Thymine glycol and thymidine glycol in human and rat urine: a possible assay for oxidative DNA damage(PDF), inProc Natl Acad Sci U S A,vol. 81, n. 18, 1984, pp. 5633–7,PMID6592579.
- ^Valerie K, Povirk L,Regulation and mechanisms of mammalian double-strand break repair,inOncogene,vol. 22, n. 37, 2003, pp. 5792–812,PMID12947387.
- ^Ferguson L, Denny W,The genetic toxicology of acridines,inMutat Res,vol. 258, n. 2, 1991, pp. 123–60,PMID1881402.
- ^Jeffrey A,DNA modification by chemical carcinogens,inPharmacol Ther,vol. 28, n. 2, 1985, pp. 237–72,PMID3936066.
- ^Stephens T, Bunde C, Fillmore B,Mechanism of action in thalidomide teratogenesis,inBiochem Pharmacol,vol. 59, n. 12, 2000, pp. 1489–99,PMID10799645.
- ^Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A,Intercalators as anticancer drugs,inCurr Pharm Des,vol. 7, n. 17, 2001, pp. 1745–80,PMID11562309.
- ^Venter J,et al.,The sequence of the human genome,inScience,vol. 291, n. 5507, 2001, pp. 1304–51,PMID11181995.
- ^Thanbichler M, Wang S, Shapiro L,The bacterial nucleoid: a highly organized and dynamic structure,inJ Cell Biochem,vol. 96, n. 3, 2005, pp. 506–21,PMID15988757.
- ^Wolfsberg T, McEntyre J, Schuler G,Guide to the draft human genome,inNature,vol. 409, n. 6822, 2001, pp. 824–6,PMID11236998.
- ^Gregory T,The C-value Enigma in plants and animals: a review of parallels and an appeal for partnership,inAnn Bot (Lond),vol. 95, n. 1, 2005, pp. 133–46,PMID15596463.
- ^The ENCODE Project Consortium,Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project,inNature,vol. 447, n. 7146, 2007, pp. 799-816,DOI:10.1038/nature05874,ISSN0028-0836.
- ^Realizzata a partire dall'entryPDB1MSW.
- ^Pidoux A, Allshire R,The role of heterochromatin in centromere function,inPhilos Trans R Soc Lond B Biol Sci,vol. 360, n. 1455, 2005, pp. 569–79,PMID15905142.
- ^Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, Gerstein M,Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22,inGenome Res,vol. 12, n. 2, 2002, pp. 272–80,PMID11827946.
- ^Harrison P, Gerstein M,Studying genomes through the aeons: protein families, pseudogenes and proteome evolution,inJ Mol Biol,vol. 318, n. 5, 2002, pp. 1155–74,PMID12083509.
- ^Animazione esplicativa dell'esperimento di Kornberg.del1958)
- ^Albà M,Replicative DNA polymerases,inGenome Biol,vol. 2, n. 1, 2001, pp. REVIEWS3002,PMID11178285.
- ^Sandman K, Pereira S, Reeve J,Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome,inCell Mol Life Sci,vol. 54, n. 12, 1998, pp. 1350–64,PMID9893710.
- ^Dame RT,The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin,inMol. Microbiol.,vol. 56, n. 4, 2005, pp. 858-70,PMID15853876.
- ^Luger K, Mäder A, Richmond R, Sargent D, Richmond T,Crystal structure of the nucleosome core particle at 2.8 A resolution,inNature,vol. 389, n. 6648, 1997, pp. 251–60,PMID9305837.
- ^Jenuwein T, Allis C,Translating the histone code,inScience,vol. 293, n. 5532, 2001, pp. 1074–80,PMID11498575.
- ^Ito T,Nucleosome assembly and remodelling,inCurr Top Microbiol Immunol,vol. 274, pp. 1–22,PMID12596902.
- ^Thomas J,HMG1 and 2: architectural DNA-binding proteins,inBiochem Soc Trans,vol. 29, Pt 4, 2001, pp. 395–401,PMID11497996.
- ^Grosschedl R, Giese K, Pagel J,HMG domain proteins: architectural elements in the assembly of nucleoprotein structures,inTrends Genet,vol. 10, n. 3, 1994, pp. 94–100,PMID8178371.
- ^Iftode C, Daniely Y, Borowiec J,Replication protein A (RPA): the eukaryotic SSB,inCrit Rev Biochem Mol Biol,vol. 34, n. 3, 1999, pp. 141–80,PMID10473346.
- ^Realizzato a partire dalla entryPDB 1LMB.
- ^Myers L, Kornberg R,Mediator of transcriptional regulation,inAnnu Rev Biochem,vol. 69, 2000, pp. 729–49,PMID10966474.
- ^Spiegelman B, Heinrich R,Biological control through regulated transcriptional coactivators,inCell,vol. 119, n. 2, 2004, pp. 157-67,PMID15479634.
- ^Li Z, Van Calcar S, Qu C, Cavenee W, Zhang M, Ren B,A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells,inProc Natl Acad Sci U S A,vol. 100, n. 14, 2003, pp. 8164–9,PMID12808131.
- ^Immagine realizzata a partire dalla entry PDB1RVA.
- ^Bickle T, Krüger D,Biology of DNA restriction,inMicrobiol Rev,vol. 57, n. 2, 1993, pp. 434–50,PMID8336674.
- ^abDoherty A, Suh S,Structural and mechanistic conservation in DNA ligases.,inNucleic Acids Res,vol. 28, n. 21, 2000, pp. 4051–8,PMID11058099.
- ^Schoeffler A, Berger J,Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism,inBiochem Soc Trans,vol. 33, Pt 6, 2005, pp. 1465–70,PMID16246147.
- ^Tuteja N, Tuteja R,Unraveling DNA helicases. Motif, structure, mechanism and function[collegamento interrotto],inEur J Biochem,vol. 271, n. 10, 2004, pp. 1849–63,DOI:10.1111/j.1432-1033.2004.04094.x,PMID15128295.
- ^Joyce C, Steitz T,Polymerase structures and function: variations on a theme?,inJ Bacteriol,vol. 177, n. 22, 1995, pp. 6321–9,PMID7592405.
- ^Hubscher U, Maga G, Spadari S,Eukaryotic DNA polymerases,inAnnu Rev Biochem,vol. 71, 2002, pp. 133–63,PMID12045093.
- ^Johnson A, O'Donnell M,Cellular DNA replicases: components and dynamics at the replication fork,inAnnu Rev Biochem,vol. 74, 2005, pp. 283–315,PMID15952889.
- ^Tarrago-Litvak L, Andréola M, Nevinsky G, Sarih-Cottin L, Litvak S,The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention,inFASEB J,vol. 8, n. 8, 1994, pp. 497–503,PMID7514143.
- ^Martinez E,Multi-protein complexes in eukaryotic gene transcription,inPlant Mol Biol,vol. 50, n. 6, 2002, pp. 925–47,PMID12516863.
- ^Immagine realizzata a partire dalla entryPDB1M6G.
- ^Cremer T, Cremer C,Chromosome territories, nuclear architecture and gene regulation in mammalian cells,inNat Rev Genet,vol. 2, n. 4, 2001, pp. 292–301,PMID11283701.
- ^Pál C, Papp B, Lercher M,An integrated view of protein evolution,inNat Rev Genet,vol. 7, n. 5, 2006, pp. 337–48,PMID16619049.
- ^O'Driscoll M, Jeggo P,The role of double-strand break repair - insights from human genetics,inNat Rev Genet,vol. 7, n. 1, 2006, pp. 45–54,PMID16369571.
- ^Vispé S, Defais M,Mammalian Rad51 protein: a RecA homologue with pleiotropic functions,inBiochimie,vol. 79, n. 9-10, 1997, pp. 587-92,PMID9466696.
- ^Neale MJ, Keeney S,Clarifying the mechanics of DNA strand exchange in meiotic recombination,inNature,vol. 442, n. 7099, 2006, pp. 153-8,PMID16838012.
- ^Dickman M, Ingleston S, Sedelnikova S, Rafferty J, Lloyd R, Grasby J, Hornby D,The RuvABC resolvasome,inEur J Biochem,vol. 269, n. 22, 2002, pp. 5492–501,PMID12423347.
- ^Joyce G,The antiquity of RNA-based evolution,inNature,vol. 418, n. 6894, 2002, pp. 214–21,PMID12110897.
- ^Orgel L,Prebiotic chemistry and the origin of the RNA world(PDF), inCrit Rev Biochem Mol Biol,vol. 39, n. 2, pp. 99–123,PMID15217990.
- ^Davenport R,Ribozymes. Making copies in the RNA world,inScience,vol. 292, n. 5520, 2001, pp. 1278,PMID11360970.
- ^Szathmáry E,What is the optimum size for the genetic Alpha bet?(PDF), inProc Natl Acad Sci U S A,vol. 89, n. 7, 1992, pp. 2614–8,PMID1372984.
- ^Lindahl T,Instability and decay of the primary structure of DNA,inNature,vol. 362, n. 6422, 1993, pp. 709–15,PMID8469282.
- ^Vreeland R, Rosenzweig W, Powers D,Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal,inNature,vol. 407, n. 6806, 2000, pp. 897–900,PMID11057666.
- ^Hebsgaard M, Phillips M, Willerslev E,Geologically ancient DNA: fact or artefact?,inTrends Microbiol,vol. 13, n. 5, 2005, pp. 212–20,PMID15866038.
- ^Nickle D, Learn G, Rain M, Mullins J, Mittler J,Curiously modern DNA for a "250 million-year-old" bacterium,inJ Mol Evol,vol. 54, n. 1, 2002, pp. 134–7,PMID11734907.
- ^Goff SP, Berg P,Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells,inCell,vol. 9, 4 PT 2, 1976, pp. 695–705,PMID189942.
- ^Houdebine L,Transgenic animal models in biomedical research,inMethods Mol Biol,vol. 360, pp. 163–202,PMID17172731.
- ^Daniell H, Dhingra A,Multigene engineering: dawn of an exciting new era in biotechnology,inCurr Opin Biotechnol,vol. 13, n. 2, 2002, pp. 136–41,PMID11950565.
- ^Job D,Plant biotechnology in agriculture,inBiochimie,vol. 84, n. 11, 2002, pp. 1105–10,PMID12595138.
- ^Collins A, Morton N,Likelihood ratios for DNA identification(PDF), inProc Natl Acad Sci U S A,vol. 91, n. 13, 1994, pp. 6007–11,PMID8016106.
- ^Weir B, Triggs C, Starling L, Stowell L, Walsh K, Buckleton J,Interpreting DNA mixtures,inJ Forensic Sci,vol. 42, n. 2, 1997, pp. 213–22,PMID9068179.
- ^Jeffreys A, Wilson V, Thein S,Individual-specific 'fingerprints' of human DNA.,inNature,vol. 316, n. 6023, pp. 76–9,PMID2989708.
- ^Baldi, Pierre. Brunak, Soren.Bioinformatics: The Machine Learning ApproachMIT Press (2001)ISBN 978-0-262-02506-5
- ^Gusfield, Dan.Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology.Cambridge University Press, 15 January 1997.ISBN 978-0-521-58519-4.
- ^Sjölander K,Phylogenomic inference of protein molecular function: advances and challenges,inBioinformatics,vol. 20, n. 2, 2004, pp. 170-9,PMID14734307.
- ^Mount DM,Bioinformatics: Sequence and Genome Analysis,2ª ed., Cold Spring Harbor, NY, Cold Spring Harbor Laboratory Press, 2004,ISBN0-87969-712-1.
- ^Immagine prelevata daStrong M. Protein nanomachines. PLoS Biol. 2004 Mar;2(3):E73. Epub 2004 Mar 16.Entrez PubMed15024422
- ^Adleman L,Molecular computation of solutions to combinatorial problems,inScience,vol. 266, n. 5187, 1994, pp. 1021–4,PMID7973651.
- ^Parker J,Computing with DNA.,inEMBO Rep,vol. 4, n. 1, 2003, pp. 7–10,PMID12524509.
- ^Ashish Gehani, Thomas LaBean and John Reif.DNA-Based Cryptography..Proceedings of the 5th DIMACS Workshop on DNA Based Computers, Cambridge, MA, USA, 14–15 June 1999.
- ^Russel P. Goodman, Schaap, Iwan A. T.; Tardin, C. F.; Erben, Christof M.; Berry, Richard M.; Schmidt, C.F.; Turberfield, Andrew J.,Rapid chiral assembly of rigid DNA building blocks for molecular nanofabrication,inScience,vol. 310, n. 5754, 9 dicembre 2005, pp. 1661–1665,Bibcode:2005Sci...310.1661G,DOI:10.1126/science.1120367,PMID16339440.
- ^T. Hodge and M.J.T.V. Cope (2000). "A Myosin Family Tree". Journal of Cell Science 113: 3353-3354Entrez PubMed10984423
- ^Wray G,Dating branches on the tree of life using DNA,inGenome Biol,vol. 3, n. 1, 2002, pp. REVIEWS0001,PMID11806830.
- ^I Letunic,Interactive Tree Of Life (iTOL): an online tool for phylogenetic tree display and annotation.,inBioinformatics,23(1), 2007, pp. 127-8.
- ^FD Ciccarelli,Toward automatic reconstruction of a highly resolved tree of life.,inScience,311(5765), 2006, pp. 1283-7.
- ^Lost Tribes of Israel,NOVA, messa in onda sullaPBS:22 febbraio 2000. Testo disponibile suPBS.org.(ultimo accesso: 4 marzo 2006)
- ^https:// technologyreview.it/come-il-dna-nellambiente-sta-offrendo-agli-scienziati-un-nuovo-modo-di-capire-il-nostro-mondo/
Bibliografia
[modifica|modifica wikitesto]- (EN) Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, Peter Walters,Molecular Biology of the Cell,New York e Londra, Garland Science, 2002,ISBN0-8153-3218-1.
- (EN) John M. Butler,Forensic DNA Typing,Elsevier, 2001,ISBN978-0-12-147951-0.
- (EN) Berg J., Tymoczko J., Stryer L.,Biochemistry,W. H. Freeman and Company, 2002,ISBN0-7167-4955-6.
- (EN) Baldi, Pierre. Brunak, Soren,Bioinformatics: The Machine Learning Approach,MIT Press, 2001,ISBN978-0-262-02506-5.
- Gusfield, Dan,Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology,Cambridge University Press, 15 gennaio 1997,ISBN 978-0-521-58519-4.
- (EN) Mount DM,Bioinformatics: Sequence and Genome Analysis,2ª ed., Cold Spring Harbor, NY, Cold Spring Harbor Laboratory Press, 2004,ISBN0-87969-712-1.
- (EN) Andrew Berry;James D. Watson,DNA: the secret of life,New York, Alfred A. Knopf, 2003,ISBN0-375-41546-7.
- (EN)James D. Watson,DNA: The Secret of Life,Random House, 2004,ISBN978-0-09-945184-6.
- (EN) Robert Olby,Francis Crick: A Biography,Plainview, N.Y, Cold Spring Harbor Laboratory Press, 2009,ISBN0-87969-798-9.
- (EN)Gunther Stent;James D. Watson,The Double Helix,New York, Norton, 1980,ISBN0-393-95075-1.
- John W. Pelley,Biochimica,Elsevier, 2008,ISBN978-88-214-3020-6.
- Giuliano Ricciotti,Biochimica di base,Zanichelli, 1987,ISBN88-08-01182-8.
Voci correlate
[modifica|modifica wikitesto]- Acido nucleico
- Complementarità
- Computer a DNA
- Conformazioni del DNA
- Cromosoma
- DNA a tripla elica
- DNA clamp
- DNA complementare
- DNA girasi
- DNA hachimoji
- DNA metiltransferasi
- DNA mitocondriale
- DNA non codificante
- DNA nucleotidil-esotrasferasi
- DNA polimerasi (DNA-dipendente)
- DNA polimerasi (RNA-dipendente)
- DNA ricombinante in botanica
- DNA ricombinante
- DNA satellite
- DNA-binding protein
- Dogma centrale della biologia molecolare
- Frammentazione del DNA
- Impronta genetica
- Macchina di DNA
- Nanotecnologia del DNA
- Prodotto genico
- Progetto genoma
- RNA
- Sequenza ripetuta di DNA
- Superavvolgimento del DNA
- Tecnologia del DNA ricombinante
- Traduzione (biologia)
- Trascrizione (biologia)
- Virus a DNA
- DNA Nucleare
Altri progetti
[modifica|modifica wikitesto] Wikiquotecontiene citazioni di o suDNA
Wikiquotecontiene citazioni di o suDNA Wikibookscontiene testi o manuali suDNA
Wikibookscontiene testi o manuali suDNA Wikizionariocontiene il lemma di dizionario «DNA»
Wikizionariocontiene il lemma di dizionario «DNA» Wikimedia Commonscontiene immagini o altri file suDNA
Wikimedia Commonscontiene immagini o altri file suDNA- Wikibooks - Analisi del proprio genoma
Collegamenti esterni
[modifica|modifica wikitesto]- DNA,suTreccani.it – Enciclopedie on line,Istituto dell'Enciclopedia Italiana.
- (EN)DNA,suEnciclopedia Britannica,Encyclopædia Britannica, Inc.
- (EN)DNA,suStarTrek,CBS Studios.
- (EN)DNA Interactive,sudnai.org.
| Controllo di autorità | Thesaurus BNCF5481·LCCN(EN)sh85037008·GND(DE)4070512-2·BNE(ES)XX527602(data)·BNF(FR)cb119649178(data)·J9U(EN,HE)987007548275805171·NDL(EN,JA)00561484 |
|---|