Splicing alternativo
Splicing alternativoé um processo pelo qual, durante aexpressão génica,éxonsde umtranscrito primáriosão clivados em locais diferentes na molécula de RNA recém sintetizada, as enzimas que exercem essa função de splicing são sustentadas pelo domínio do braço de carboxila do complexo da RNA polimerase II, deste modo com as diferentes composições dosíntronsque foram removidos, os mRNA maduros são compostos de bases com sequências diferentes, implicando assim em códons diferentes e consequentemente em polipeptídeos com sequências de aminoácidos distintas, o que constitui proteínas de diferentes funções que foram codificadas por um mesmo gene.[1][2] Em muitos casos, o processo desplicingpode criar váriasproteínasúnicas por variações no splicing do mesmotranscrito primário.Esta descoberta fez avançar a pesquisa de doenças que antes era norteada pelo "paradigma monogênico (uma doença, uma mutação)"[3]e passou para o poligênico "onde uma mutação genética significariam desde então várias proteínas defeituosas e várias correlações com diversas doenças, ou várias novas características (no caso de mutações positivas).
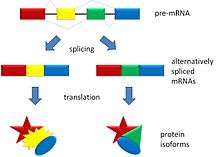
A descoberta de apenas 30 000genesemhumanosnão reflete a dimensão correta do número possível de proteínas a serem sintetizadas. O splicing alternativo permite que uma única fita deRNA mensageirorecém-sintetizada sofra diversas possibilidades deprocessamento,aumentando consideravelmente o número total possível de proteínas.
A produção de RNAm através do splicing alternativo é regulada por um sistema de proteínas que se ligam a sítios de ação durante a transcrição primária. Essas proteínas incluem ativadores do splicing, que promovem o uso de um sítio específico de splicing, e também incluem inibidores de splicing, que reduzem o uso de um sítio específico. Os mecanismos do splicing alternativo variam muito e novos exemplos têm sido constantemente descobertos.
Bibliografia
editarRicardo Godinho, Ivan Keogh e Roland Eavey (2003) [pt Perda auditiva genética]., Revista Brasileira de Otorrinolaringologia,69(1).doi:10.1590/S0034-72992003000100016.
Referências
- ↑Crabtree, G. R.; Kant, J. A. (novembro de 1982).«Organization of the rat gamma-fibrinogen gene: alternative mRNA splice patterns produce the gamma A and gamma B (gamma ') chains of fibrinogen».Cell(1): 159–166.ISSN0092-8674.PMID6897622.doi:10.1016/0092-8674(82)90415-9.Consultado em 2 de março de 2021
- ↑Strachan, Tom; Read, Andrew (2011).Human Molecular Genetics(em inglês) 4ª ed. New York: Garland Science. p. 373-374. 781 páginas.ISBN978-0-815-34149-9
- ↑«Conhecer o próprio genoma envolve surpresas e decepções».Folha de S.Paulo.10 de março de 2019.Consultado em 2 de março de 2021