Grupo externo (cladística)
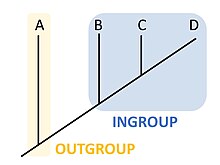
Grupo externo(muitas vezesoutgroup) é a designação dada emcladísticaoufilogenéticaao um grupo de organismos mais distantemente relacionado que serve como grupo de referência para determinar asrelações evolutivasdo grupo interno, ou seja do conjunto de organismos em estudo.[1]O conceito é distinto de «grupo externo» ou «exogrupo» utilizado em sociologia ou etnografia.
Descrição
[editar|editar código-fonte]O grupo externo é usado como ponto de comparação para o grupo interno e permite especificamente que aárvore filogenéticado grupo em estudo seja enraizada. Como a polaridade (direcção) da mudança de caracteres pode ser determinada apenas numa filogenia enraizada, a escolha do grupo externo é essencial para o entendimento da evolução das características ao longo de uma filogenia.[2]
Embora o conceito de grupo externo tenha sido usado desde os primórdios da cladística, acredita-se que o termo «outgroup» tenha sido cunhado no início dos anos 1970 noAmerican Museum of Natural History.[3]Antes da generalização do conceito e do termo, vários outros termos foram usados pelos biólogos evolucionários, incluindo «exogrupo», «grupo relacionado» e «grupos exteriores».[3]
Escolha do grupo externo
[editar|editar código-fonte]Na base da selecção do «grupo externo» está a hipótese de que este esteja menos filogeneticamente próximo do grupo interno (o grupo em estudo) do que qualquer dos membros do grupo interno entre si. A conclusão evolucionária dessas relações é que as espécies pertencentes ao grupo externo têm um ancestral comum com o grupo interno que é mais antigo que o ancestral comum que une os membros do grupo interno. Em consequência, a escolha do grupo externo pode modificar atopologiada filogenia que a partir dele é determinada.[4]
Em consequência da potencial arbitrariedade introduzida pela escolha do grupo externo, os filogeneticistas geralmente usam mais de um grupo externo na análise cladística. O uso de múltiplos grupos externos é preferível porque fornece uma filogenia mais robusta, protegendo contra os candidatos de grupos desfavoráveis e testando amonofiliahipotética do grupo interno.[3][5][6]
Para poder ser qualificado como um bom grupo externo, umtáxondeve satisfazer cumulativamente as duas características seguintes:
- Não deve ser um membro do grupo interno (do grupo em estudo);
- Deve estar relacionado com o grupo interno, com uma relação filogenética suficientemente próxima para permitir comparações significativas com os caracteres definidores do grupo interno.
Portanto, um grupo externo apropriado deve estar inequivocamente fora docladode interesse no estudo filogenético. Um grupo externo que esteja aninhado dentro do grupo interno, quando usado para radicar a filogenia, resultará inevitavelmente em conclusões incorrectas sobre as relações filogenéticas e a evolução das características dos membros do grupo em estudo.[7]No entanto, o nível óptimo de relacionamento do grupo externo com o grupo interno depende da profundidade da análise filogenética. Escolher um outgroup intimamente relacionado em relação ao grupo interno é mais útil quando se olha para diferenças subtis, enquanto escolher um grupo externo indevidamente distante pode resultar em confundir o fenómeno daevolução convergentecom um relacionamento evolutivo directo devido à partilha de umancestral comum.[8][9]
Para analises filogenéticas superficiais, por exemplo para resolver as relações evolutivas de um clado dentro de um género, um grupo externo apropriado seria um membro doclado irmão.[10]No entanto, para uma análise filogenética mais profunda, os táxons menos próximos podem ser usados. Por exemplo, para investigar os primeiros ramos da filogenia das aves foram usados humanos e crocodilos como grupos externos.[11]
Emfilogenética molecular,satisfazer o requisito de existência de um ancestral comum próximo tipicamente significa quesequências de DNA,ou deproteínas,do grupo externo possam seralinhadascom sucesso para comparação com sequências do grupo interno. Embora existamabordagens algorítmicaspara identificar os grupos externos com amáxima parcimóniaglobal, estes grupos são frequentemente limitados por não reflectir a natureza quantitativa contínua de certos estados de caracter em estudo.[12]Os estados de caracter são traços, ancestrais ou derivados, que afectam a construção de padrões de ramificação numaárvore filogenética.[13]
Exemplos
[editar|editar código-fonte]| Ingroup | Outgroup |
|---|---|
| Grandes macacos[14] | Gibões |
| Mamíferos plancentários[15] | Marsupiais |
| Cordados[16] | Equinodermes |
| Angiospérmicas[17] | Gimnospérmicas |
Em cada exemplo, umafilogeniade organismos no grupo interno pode ser enraizada por referência aos mesmos estados de cada característica (cada traço) em relação a um ou mais membros do grupo externo.
Ver também
[editar|editar código-fonte]- Apomorfia,uma característica derivada de um organismo.
- Grupo irmão,um grupo que pode estar intimamente relacionado com um grupo interno.
- Plesiomorfia,uma característica ancestral de um organismo.
- Primitivo (filogenética),um termo para designar características ancestrais.
Referências
[editar|editar código-fonte]- ↑Grimaldi, David; Engel, Michael S.; Engel, Michael S. (16 de maio de 2005).Evolution of the Insects.[S.l.: s.n.]ISBN9780521821490
- ↑Farris, J. S. (1982). «Outgroups and Parsimony».Systematic Biology.31(3): 328–334.ISSN1063-5157.doi:10.1093/sysbio/31.3.328
- ↑abcNixon, Kevin; Carpenter, James (dezembro de 1993). «On Outgroups».Cladistics.9(4): 413–426.doi:10.1111/j.1096-0031.1993.tb00234.x
- ↑Giribet, G.; Ribera, C. (junho de 1998). «The position of arthropods in the animal kingdom: a search for a reliable outgroup for internal arthropod phylogeny».Molecular Phylogenetics and Evolution.9(3): 481–488.PMID9667996.doi:10.1006/mpev.1998.0494
- ↑Barriel, V.; Tassy, P. (junho de 1998). «Rooting with Multiple Outgroups: Consensus Versus Parsimony».Cladistics.14(2): 193–200.doi:10.1111/j.1096-0031.1998.tb00332.x
- ↑de la Torre-Barcena, Jose Eduardo; Kolokotronis, S.O.; Lee, Ernest; Stevenson, Dennis; Brenner, Eric; Katari, Manpreet; Coruzzi, Gloria; DeSalle, Rob (2009).«The Impact of Outgroup Choice and Missing Data on Major Seed Plant Phylogenetics Using Genome-Wide EST Data».PLOS ONE.4(6): e5764.PMC2685480
 .PMID19503618.doi:10.1371/journal.pone.0005764
.PMID19503618.doi:10.1371/journal.pone.0005764
- ↑Maddison, Wayne; et al. (1984).«Outgroup Analysis and Parsimony»(PDF).Systematic Zoology.33(1): 83–103.JSTOR2413134.doi:10.2307/2413134
- ↑Wilberg, Eric W. (1 de julho de 2015).«What's in an Outgroup? The Impact of Outgroup Choice on the Phylogenetic Position of Thalattosuchia (Crocodylomorpha) and the Origin of Crocodyliformes».Systematic Biology.64(4): 621–637.ISSN1063-5157.PMID25840332.doi:10.1093/sysbio/syv020
- ↑O'BRIEN, MICHAEL J.; LYMAN, R.LEE; SAAB, YOUSSEF; SAAB, ELIAS; DARWENT, JOHN; GLOVER, DANIEL S. (2002). «Two Issues in Archaeological Phylogenetics: Taxon Construction and Outgroup Selection».Journal of Theoretical Biology.215(2): 133–150.PMID12051970.doi:10.1006/jtbi.2002.2548
- ↑David A. Baum; Stacey D. Smith (2013).Tree Thinking: An Introduction to Phylogenetic Biology.[S.l.]: Roberts. p. 175.ISBN978-1-936221-16-5
- ↑Jarvis, E.; et al. (dezembro de 2014).«Whole-genome analyses resolve early branches in the tree of life of modern birds».Science.346(6215): 1320–1331.PMC4405904
 .PMID25504713.doi:10.1126/science.1253451
.PMID25504713.doi:10.1126/science.1253451
- ↑Stevens, P. F. (1991). «Character States, Morphological Variation, and Phylogenetic Analysis: A Review».Systematic Botany.16(3): 553–583.JSTOR2419343.doi:10.2307/2419343
- ↑Rineau, Valentin; Grand, Anaïs; Zaragüeta, René; Laurin, Michel (1 de maio de 2015).«Experimental systematics: sensitivity of cladistic methods to polarization and character ordering schemes».Contributions to Zoology.84(2): 129–148 – via EBSCOhost
- ↑Prado-Martinez, Javier; Marques-Bonet, Tomas (2013).«Great ape genetic diversity and population history».Nature.499(7459): 471–475.PMC3822165
 .PMID23823723.doi:10.1038/nature12228
.PMID23823723.doi:10.1038/nature12228
- ↑Murphy, William; Pringle, Thomas; Crider, Tess; Springer, Mark; Miller, Webb (2007).«Using genomic data to unravel the root of the placental mammal phylogeny».Genome Research.17(4): 413–421.PMC1832088
 .PMID17322288.doi:10.1101/gr.5918807
.PMID17322288.doi:10.1101/gr.5918807
- ↑Cameron, Chris; Garey, James; Swalla, Billie (2000).«Evolution of the chordate body plan: New insights from phylogenetic analyses of deuterostome phyla».PNAS.97(9): 4469–4474.PMC18258
 .PMID10781046.doi:10.1073/pnas.97.9.4469
.PMID10781046.doi:10.1073/pnas.97.9.4469
- ↑Matthews, Sarah; Donoghue, Michael (1999). «The Root of Angiosperm Phylogeny Inferred from Duplicate Phytochrome Genes».Science.286(5441): 947–950.doi:10.1126/science.286.5441.947
