Jakobea
| Jakobea Jakobida | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
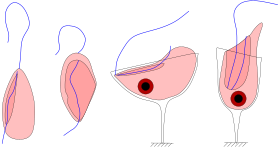 Quatro espécies de jacobídeos, mostrand o sulco e os flagelos:Jakoba libera(vista ventral),Stygiella incarcerata(vista ventral),Reclinomonas americana(vista dorsal) eHistiona aroides(vista ventral). | |||||||||||
| Classificação científica | |||||||||||
| |||||||||||
| Famílias | |||||||||||
| Sinónimos | |||||||||||
Jakobeaé umcladodemicroorganismosheterotróficosflagelados,geralmente considerado nonível taxonómicodeclasseou defilo,pertencentes aosupergrupoExcavata.Os membros do agrupamento são pequenos eucariotas devida livre(menos de 15 μm) encontrados em ambientesaeróbicoseanaeróbicos.[3][4][5]Estão validamente descritas pelo menos 22 espécies.
Descrição[editar|editar código-fonte]
A ordem Jakobida, que se acredita sermonofilética,consiste em apenas 20-22 espécies, tendo sido classificada como um grupo em 1993.[3][5][6]Existem ainda algumas incertezas sobre osgenomas mitocondriaisdos jakobídeos, que são extraordinariamente grandes e semelhantes abactérias,evidência de que os jakobídeos podem ser importantes para a história evolutiva doseucariotas.[4][7]
Evidências obtidas com recurso às tecnicas dafilogenética molecularsugerem fortemente que os jakobídeos estão fortemente relacionados comHeterolobosea(Percolozoa) eEuglenozoa[8]do que com qualquer outroagrupamento taxonómico.
Morfologia[editar|editar código-fonte]
Os jakobídeos apresentam doisflagelos,inseridos na extremidade anterior da célula, e, como outros membros do agrupamentoExcavata,têm um sulco de alimentação ventral associado a umcitoesqueletode suporte.[9]O flagelo posterior possui uma palheta dorsal e está alinhado dentro do sulco ventral, onde gera uma corrente que a célula utiliza para a ingestão de alimentos.[7][9]
Onúcleoestá geralmente na parte anterior da célula e possui umnucléolo.A maioria dos jakobídeos conhecidos tem umamitocôndria,novamente localizada anteriormente, que em diferentes géneros pode tercristasachatadas, tubulares ou ausentes. Osvacúolosalimentares estão localizados principalmente na parte posterior da célula e, na maioria dos jacobídeos, oretículo endoplasmáticoestá distribuído por toda a célula.[6]
OsHistionidae,que são organismossésseiseloricados,e ocasionalmente os nadadores livres da espécieJakoba libera(Jakobidae), apresentamextrussomassob a membrana dorsal, organelos que teoricamente são estruturas defensivas.[3][6]
DNA mitocondrial[editar|editar código-fonte]
Como os Jokobea não têm uso económico conhecido, a maioria da investigação sobre o grupo esteve concentrada sobre o seu significado evolutivo. ODNA mitocondrialdos jakobídeos é o mais parecido com uma bactéria de todos os DNA mitocondriais eucarióticos conhecidos, sugerindo que osgenomasmitocondriais dos jakobídeos podem estar próximos do genoma mitocondrial ancestral.[6]
O DNA mitocondrial de Jakobea é substancialmente diferente da maioria dos outros eucariotas, especialmente em termos de número de genes (quase 100 em algumas espécies) e na presença de elementos semelhantes a genes de bactérias nos seus genomas.[5][6]Nove dos genes nunca foram encontrados no DNA mitocondrial eucariótico. Excepcionalmente, os genomas mitocondriais dos jakobídeos codificam para o tipo bacteriano daRNA polimerase,em oposição ao típicoRNA polimeraseeucariótico mitocondrial, referido como “tipo fago”, que parece ser de origem viral.[6]
Essas características não significam necessariamente que os jakobídeos são basais para a filogenia dos eucariotos. Enquanto as mitocôndrias dos jakobídeos têm características genéticas que parecem terem sido desenvolvidas a partir de bactérias, e aparentemente não possuem RNA do tipo fago, é possível que outros clados eucarióticos tenham independentemente perdido as características bacterianas.[10]
Foram propostas várias possibilidades para explicar as características bacterianas do DNA mitocondrial deste grupo. Uma delas é que os jakobídeos divergiram muito cedo do resto dos eucariotas. Essa hipótese depende de se comprovar que os jakobídeos são realmente basais para todos os eucariotas vivos, mas ainda não há evidências para apoiar essa sugestão.[6]
Outra hipótese é que a RNA polimerase do tipo fago passou de um grupo de eucariotas para outro viatransferência lateral de genes,substituindo a enzima do tipo bacteriano, e simplesmente não atingiu os jakobídeos. Isso não dependeria de os jakobídeos serem basais para os eucariotas como um todo, mas a questão não foi amplamente estudada e permenece incerta.[6]
Uma terceira possibilidade é o inverso das outras, sugerindo que a RNA polimerase do tipo fago é a basal. Nesse cenário, os jakobídeos adquiriram a RNA polimerase do tipo bacteriano muito mais recentemente e que então se espalharam por transferência lateral de genes.[6]No entanto, o arranjo génico do DNA mitocondrial dos jakobídeos sugere uma origem ancestral da polimerase de RNA do tipo bacteriano como umadivergênciamais recente.[5][6]
Um dos cenários propostos sugere que oancestral comumdos eucariotas tinha duas polimerases de RNA mitocondrial, tanto do tipo fago quanto do tipo bactéria, e os jakobídeos perderam a polimerase do tipo fago, enquanto o restante dos eucariotas perdeu o tipo de bactéria, possivelmente várias vezes.[6][11]Tal modelo elimina a necessidade de os jakobídeos serem verdadeiramente basais. Um estudo propôs que as polimerases do tipo fago e do tipo bactéria, quando presentes na mesma mitocôndria, desempenhavam funções diferentes, da mesma forma que osorganelosdas plantas terrestres têm duas enzimas RNA polimerase diferentes que transcrevem genes diferentes.[6]
Ecologia[editar|editar código-fonte]
Os jakobídeos têm uma ampladistribuição natural,tendo sido encontrados nosolo,água docee em diversoshabitatsmarinhos, mas geralmente não são comuns.[4][7][6][12]Contudo, estudos deDNA ambientalsugerem que osStygiellidaesão abundantes em habitats marinhos anóxicos.[6][13]
Alguns membros deste agrupamento são capazes de sobreviver em ambientes hipersalinos e anóxicos, embora os histionídeos tenham sido encontrados apenas em ecossistemas de água doce, onde se ligam aalgasouzooplâncton.[6]Fora das espécies sésseis obrigatórias, muitas espécies de jakobídeos podem-se fixar temporariamente sobre superfícies, usando um dos dois flagelos ou o próprio corpo celular.[13]
Todos os jakobídeos conhecidos são organimsos heterotróficos que vivem como alimentadores de suspensão.[4][6]Acredita-se que as suas presas primárias sejam bactérias, embora uma espécie tenha sido observada ingerindo células eucarióticas extremamente pequenas (< 1 µm).[5][14]Os jakobídeos são geralmente nadadores lentos, com baixas taxas de depuração em relação a organismos semelhantes.[6]Nenhum estudo sugeriu que os jakobídeos possam ser patogénicos ou tóxicos para humanos e para a macrofauna.[6]
Taxonomia[editar|editar código-fonte]
Jakobídeos são um grupomonofiléticoe estão filegeneticamente mais intimamente relacionados com osEuglenozoaeHeterolobosea.[5][6][10]Na sua presentecircunscrição taxonómica,o agrupamento Jakobida contém cinco famílias que consistem principalmente em géneros de natação livre:Jakobidae,Moramonadidae,AndaluciidaeeStygiellidae.[6]A sexta família,Histionidae,é amplamente dominada por génerossésseisloricadose inclui os primeiros jacobídeos que foram descritos.[6]Uma possível taxonomia ecladogramapara o grupo são os seguintes:
| ||||||||||||||||||||||||||||||||||||||||||||||||
- ClasseJakobeaCavalier-Smith 1999
- OrdemJakobidaCavalier-Smith 1993
- SubordemOphirininaYabuki et al. 2018
- FamíliaOphirinidaeYabuki et al. 2018
- GéneroOphirinaYabuki et al. 2018
- EspécieOphirina amphinemaYabuki et al. 2018
- GéneroOphirinaYabuki et al. 2018
- FamíliaOphirinidaeYabuki et al. 2018
- SubordemAndaluciinaCavalier-Smith 2013
- FamíliaAndaluciidaeCavalier-Smith 2013
- GéneroAndaluciaLara et al. 2006
- EspécieAndalucia godoyiLara et al. 2006
- GéneroAndaluciaLara et al. 2006
- FamíliaStygiellidaePánek, Táborský & Čepička 2015[13]
- GéneroVelundellaPánek, Táborský & Čepička 2015
- EspécieV. nautaPánek, Táborský & Čepička 2015
- EspécieV. trypanoidesPánek, Táborský & Čepička 2015
- GéneroStygiellaPánek, Táborský & Čepička 2015 non Bruand 1853
- EspécieS. incarcerata(Bernard, Simpson & Patterson 2000) Pánek, Táborský & Čepička 2015[Jakoba incarcerataBernard, Simpson & Patterson 2000;Andalucia incarcerata(Bernard, Simpson & Patterson 2000) Lara et al. 2006]
- EspécieS. agilisPánek, Táborský & Čepička 2015
- EspécieS. crypticaPánek, Táborský & Čepička 2015
- EspécieS. adhaerensPánek, Táborský & Čepička 2015
- GéneroVelundellaPánek, Táborský & Čepička 2015
- FamíliaAndaluciidaeCavalier-Smith 2013
- SubordemHistoninaCavalier-Smith 1993
- Espécie?Jakoba echidnaO'Kelly 1991
- FamíliaMoramonadidaeStrassert et al. 2016
- GéneroMoramonasStrassert et al. 2016
- EspécieMoramonas marocensisStrassert et al. 2016
- GéneroSeculamonasMarx et al. 2003 nomen nudum
- EspécieSeculamonas ecuadoriensisMarx et al. 2003 nomen nudum
- GéneroMoramonasStrassert et al. 2016
- FamíliaJakobidaePatterson 1990
- GéneroJakobaPatterson 1990
- EspécieJakoba bahamiensisBurger & Lang (indeitum)
- EspécieJakoba libera(Ruinen 1938) Patterson 1990[Cryptobia liberaRuinen 1938]
- GéneroJakobaPatterson 1990
- FamíliaHistionidaeFlavin & Nerad 1993
- GéneroHistionaVoigt 1902[ZachariasiaVoigt 1901 non Lemmermann 1895]
- Espécie?H. planctonicaScourfield 1937
- EspécieH. aroidesPascher 1943
- EspécieH. velifera(Voigt 1901) Pascher 1943[Zachariasia veliferaVoigt 1901;Histiona zachariasiiVoigt 1901 nom. illeg.]
- GéneroReclinomonasFlavin & Nerad 1993
- EspécieR. americanaFlavin & Nerad 1993
- EspécieR. campanula(Penard 1921) Flavin & Nerad 1993[Histiona campanulaPenard 1921;Stenocodon campanula(Penard 1921) Pascher 1942]
- GéneroStenocodonPascher 1942
- EspécieStenocodon epiplanktonPascher 1942
- GéneroStomatochonePascher 1942
- EspécieS. infundibuliformisPascher 1942
- EspécieS. cochlearPascher 1942
- EspécieS. excavataPascher 1942
- EspécieS. epiplanktonPascher 1942
- GéneroHistionaVoigt 1902[ZachariasiaVoigt 1901 non Lemmermann 1895]
- SubordemOphirininaYabuki et al. 2018
- OrdemJakobidaCavalier-Smith 1993
Ver também[editar|editar código-fonte]
Referências[editar|editar código-fonte]
- ↑Cavalier-Smith T (2003). «The excavate protozoan phyla Metamonada Grassé emend. (Anaeromonadea, Parabasalia, Carpediemonas, Eopharyngia) and Loukozoa emend. (Jakobea, Malawimonas): their evolutionary affinities and new higher taxa».International Journal of Systematic and Evolutionary Microbiology.53:1741–1758.doi:10.1099/ijs.0.02548-0
- ↑Cavalier-Smith T (1997). «Amoeboflagellates and Mitochondrial Cristae in Eukaryote Evolution: Megasystematics of the New Protozoan Subkingdoms Eozoa and Neozoa».Archiv für Protistenkunde.147:237–258
- ↑abcO'Kelly, Charles J. (1993). «The Jakobid flagellates: structural features of Jakoba, Reclinomonas, and Histonia and implications for the early diversification of eukaryotes».Journal of Eukaryotic Microbiology.40(5): 627–636.doi:10.1111/j.1550-7408.1993.tb06120.x
- ↑abcdStrassert, Jürgen F. H.; Tikhonenov, Denis V.; Pombert, Jean-François; Kolisko, Martin; Tai, Vera; Mylnikov, Alexander P.; Keeling, Patrick J. (2016).«Moramonas marocensis gen. nov., sp. nov.: a jakobid flagellate isolated from desert soil with a bacteria-like, but bloated mitochondrial genome».Open Biology.6(2). 150239 páginas.PMC4772810
 .PMID26887409.doi:10.1098/rsob.150239
.PMID26887409.doi:10.1098/rsob.150239
- ↑abcdefBurger, Gertraud; Gray, Michael W.; Forget, Lise; Lang, B. Franz (2013).«Strikingly Bacteria-Like and Gene-Rich Mitochondrial Genomes throughout Jakobid Protists».Genome Biology and Evolution.5(2): 418–438.PMC3590771
 .PMID23335123.doi:10.1093/gbe/evt008
.PMID23335123.doi:10.1093/gbe/evt008
- ↑abcdefghijklmnopqrstuSimpson, Alastair G. B. (2017). «Jakobids». In: Archibald, John M.; Simpson, Alastair G. B.; Slamovits, Claudio H.Handbook of the Protists.[S.l.]: Springer, Cham. pp. 973–1003.ISBN978-3-319-28147-6.doi:10.1007/978-3-319-28149-0_6
- ↑abcLara, Enrique; Chatzinotas, Antonis; Simpson, Alastair G. B. (2006). «Andalucia (n. gen.)—the Deepest Branch Within Jakobids (Jakobida; Excavata), Based on Morphological and Molecular Study of a New Flagellate from Soil».Journal of Eukaryotic Microbiology.53(2): 112–120.PMID16579813.doi:10.1111/j.1550-7408.2005.00081.x
- ↑Hampl V, Hug L, Leigh JW, Dacks JB, Lang BF, Simpson AG, Roger AJ (fevereiro de 2009).«Phylogenomic analyses support the monophyly of Excavata and resolve relationships among eukaryotic "supergroups"».Proc. Natl. Acad. Sci. U.S.A.106(10): 3859–64.Bibcode:2009PNAS..106.3859H.PMC2656170
 .PMID19237557.doi:10.1073/pnas.0807880106
.PMID19237557.doi:10.1073/pnas.0807880106
- ↑abSimpson, Alastair G. B.; Patterson, David J. (2001). «On Core Jakobids and Excavate Taxa: The Ultrastructure of Jakoba incarcerata».Journal of Eukaryotic Microbiology.48(4): 480–492.PMID11456326.doi:10.1111/j.1550-7408.2001.tb00183.x
- ↑abRodriguez-Ezpeleta, Naiara; Brinkmann, Henner; Burger, Gertraud; Roger, Andrew J.; Gray, Michael W.; Philippe, Herve; Lang, B. Franz (2007). «Toward Resolving the Eukaryotic Tree: The Phylogenetic Positions of Jakobids and Cercozoans».Current Biology.17(16): 1420–1425.PMID17689961.doi:10.1016/j.cub.2007.07.036

- ↑Stechmann, Alexandra; Cavalier-Smith, Thomas (2002). «Rooting the Eukaryote Tree by Using a Derived Gene Fusion».Science.297(5578): 89–91.Bibcode:2002Sci...297...89S.PMID12098695.doi:10.1126/science.1071196
- ↑Lara, Enrique; Berney, Cedric; Ekelund, Flemming; Harms, Hauke; Chatzinotas, Antonis (2007).«Molecular comparison of cultivable protozoa from a pristine and a polycyclic aromatic hydrocarbon polluted site»(PDF).Soil Biology and Biochemistry.39(1): 139–148.doi:10.1016/j.soilbio.2006.06.017
- ↑abcPánek, Tomáš; Táborský, Petr; Pachiadaki, Maria G.; Hroudová, Miluše; Vlček, Čestmir; Edgcomb, Virginia P.; Čepička, Ivan (2015).«Combined Culture-Based and Culture-Independent Approaches Provide Insights into Diversity of Jakobids, an Extremely Plesiomorphic Eukaryotic Lineage».Frontiers in Microbiology.6:art. 1288.PMC4649034
 .PMID26635756.doi:10.3389/fmicb.2015.01288
.PMID26635756.doi:10.3389/fmicb.2015.01288
- ↑Christaki, Urania; Vázquez-Domínguez, Evaristo; Courties, Claude; Lebaron, Phillipe (2005). «Grazing impact of different heterotrophic nanoflagellates on eukaryotic (Ostreococcus tauri) and prokaryotic picoautotrophs (ProchlorococcusandSynechococcus)».Environmental Microbiology.7(8): 1200–1210.PMID16011757.doi:10.1111/j.1462-2920.2005.00800.x
