DNA


DNAhayADN (deoxyribonucleic)làphân tửmangthông tin di truyềnquy định mọi hoạt động sống (sinh trưởng,phát triểnvàsinh sản) của cácsinh vậtvà nhiều loàivirus.Đây là từ viết tắtthuật ngữtiếng Anhdeoxyribonucleic acid(/diːˈɒksɪˌraɪboʊnjuːˌkliːɪk,
Hai mạch DNA này được gọi là cácpolynucleotidevì thành phần của chúng bao gồm cácđơn phânnucleotide.[4][5]Mỗinucleotideđược cấu tạo từ một trong bốn loạinucleobasechứanitơ—hoặc làcytosine(C, ở Việt Nam còn viết làxitôzin,viết tắt X),guanine(G),adenine(A), haythymine(T)—liên kết vớiđườngdeoxyribosevà mộtnhóm phosphat.Các nucleotide liên kết với nhau thành một mạch DNA bằng liên kết phosphodieste (liên kết cộng hóa trị) giữa phân tử đường của nucleotide với nhóm phosphat của nucleotide tiếp theo, tạo thành "khung xương sống" đường-phosphat luân phiên vững chắc.
Những base nitơ giữa hai mạch đơn polynucleotide liên kết với nhau theonguyên tắc bổ sung(A liên kết với T, và C liên kết với G) thông qua các mối liên kết hydro để tạo nên chuỗi DNA mạch kép. Tổng số lượng cặp base liên quan tới DNA trênTrái Đấtước tính bằng 5,0 x 1037,và nặng khoảng 50 tỷtấn.[6]Để so sánh, tổngkhối lượngcủasinh quyểnxấp xỉ bằng 4 nghìn tỷ tấncarbon.[7]
DNA lưu trữthông tinsinh học, cácmã di truyềnđến các thế hệ tiếp theo và để chỉ dẫn cho quá trìnhsinh tổng hợp protein.Mạch đơn DNA có liên kết hóa học vững chắc chống lại sự phân cắt, và hai mạch đơn của chuỗi xoắn kép lưu trữ thông tin sinh học như nhau. Thông tin này được sao chép nhờ sự phân tách hai mạch đơn. Một tỷ lệ đáng kể DNA (hơn 98% ởngười) là các đoạnDNA không mã hóa(non-coding), nghĩa là những vùng này không giữ vai trò mạch khuôn để xác định trình tự protein thông qua các quá trìnhphiên mã,dịch mã.
Hai mạch DNA chạy song song theo hai hướng ngược nhau. Gắn với mỗi phân tử đường là một trong bốn loạinucleobase(hay cácbase). Thông tin di truyền được mã hóa bởitrình tựcủa bốn nucleobase gắn trên mỗi mạch đơn. Những mạch RNA được tổng hợp từ những khuôn mẫu là mạch gốc của DNA trong quá trìnhphiên mã.Và dưới sự chỉ dẫn củamã di truyền,phân tử RNA tiếp tục được diễn dịch để xác định trình tự cácamino acidở cấu trúc protein trong quá trình dịch mã.
DNA ởtế bào nhân thực(động vật,thực vật,nấmvànguyên sinh vật) được lưu trữ bên trongnhân tế bàovà một sốbào quan,nhưty thểhoặclục lạp.[8]Ngược lại, ởsinh vật nhân sơ(vi khuẩnvàvi khuẩn cổ), do không có nhân tế bào, DNA nằm trongtế bào chất.Bên trongtế bào,DNA tổ chức thành những cấu trúc dài gọi lànhiễm sắc thể(chromosome). Trong giai đoạnphân bàocác nhiễm sắc thể hình thành được nhân đôi bằng cơ chếnhân đôi DNA,mang lại cho mỗi tế bào có một bộ nhiễm sắc thể hoàn chỉnh như nhau. Ở nhiễm sắc thể sinh vật nhân thực, những proteinchất nhiễm sắc(chromatin) như histone giúp thắt chặt và tổ chức cấu trúc DNA. Chính cấu trúc thắt chặt này sẽ quản lý sự tương tác giữa DNA với các protein khác, quy định vùng nào của DNA sẽ được phiên mã.
Friedrich Miescherđã cô lập được DNA lần đầu tiên vào năm 1869.Francis CrickvàJames Watsonnhận ra cấu trúc phân tử chuỗi xoắn kép của nó vào năm 1953, dựa trên mô hình xây dựng từ dữ liệu thu thập qua ảnh chụpnhiễu xạ tia XdoRosalind Franklinthực hiện. DNA trở thành một công cụ phân tử giúp các nhà nghiên cứu khám phá các lý thuyết và định luậtvật lý sinh học,nhưđịnh lý ergodicvà lý thuyếtđàn hồi.Những tính chất vật liệu độc đáo của DNA biến nó trở thành phân tử hữu ích đối với các nhà khoa học vật liệu quan tâm trong lĩnh vực chế tạo vật liệu cỡ micro và nano, như trongcông nghệ nano DNA.Các tiến bộ trong lĩnh vực này bao gồm phương pháporigami DNAvà vật liệu lai dựa trên DNA.[9]
Cấu trúc
[sửa|sửa mã nguồn]DNA là mộtpolymerdài cấu tạo bởi các đơn phânnucleotidelặp lại.[10][11]Cấu trúc DNA của mọi loài là động không phải là tĩnh.[12]Hai mạchpolynucleotideliên kết với nhau bằng liên kết hydro, xoắn đều quanh một trục tưởng tượng theo chiều từ trái sang phải (xoắn phải), mỗi chu kỳ xoắn (gồm 10 cặpnucleotide) dài 34ångström(3,4nm) và có bán kính 10 ångström (1,0 nm).[13]Theo một nghiên cứu, khi đo đạc trong mộtdung dịch,chuỗi phân tử DNA rộng 22–26 Å (2,2–2,6 nm, và một đơn phân nucleotide dài 3,3 Å (0,33 nm).[14]Dù cho mỗi đơn vị lặp lại có kích thước rất nhỏ, polymer DNA vẫn là những phân tử rất lớn chứa hàng triệu nucleotide. Ví dụ, DNA trong nhiễm sắc thể lớn nhất ở người,nhiễm sắc thể số 1,chứa xấp xỉ 220 triệucặp base[15]và dài đến 85 mm nếu được duỗi thẳng.

DNA thường không là một chuỗi đơn lẻ, mà thay vào đó là cặp chuỗi liên kết chặt chẽ với nhau.[13][16]Hai mạch dài này quấn gắn kết với nhau. Mộtnucleobaseliên kết với một phân tử đường tạo thành cấu trúc gọi lànucleoside,và một base liên kết với một phân tử đường và một hoặc nhiều nhóm phosphat gọi lànucleotide(nucleotidetrong DNA vàRNAlà loại nucleotide chỉ mang một nhómphosphat). Mạchpolymerchứa nhiềunucleotideliên kết với nhau (như trong DNA) được gọi làpolynucleotide.[17]
Khung xương chính của mạch DNA tạo nên từ các nhómphosphatvàphân tử đườngxen kẽ nhau.[18]Phân tử đường trong DNA là2-deoxyribose,là đườngpentose(5carbon). Các phân tửđườngliên kết với các nhómphosphattạo thànhliên kết phosphodiestegiữanguyên tửcarbonthứ 3 với nguyên tửcarbonthứ 5 trên hai mạch vòng của hai phân tửđườngliền kề.Liên kếtbất đối xứng này cho phép xác định hướng chạy của mạch đơn DNA. Xem xét gần hơn trên một chuỗi xoắn kép, người ta nhận thấy cácnucleotidehướng theo một chiều trên một mạch và theo chiều ngược lại trên mạch kia, gọi là: hai mạchhướng ngược chiều nhauhayđối song song(antiparallel). Các đầu không đối xứng kết thúc của chuỗi DNA là đầu 5′ (năm phẩy) và đầu 3′ (ba phẩy), với đầu 5′ kết thúc bởi nhómphosphatvà đầu 3′ kết thúc bởi nhómhydroxyl(OH). Sự khác nhau chủ yếu giữa DNA vàRNAlà ở phân tử đường, vớiđường2-deoxyribosetrong DNA được thay thế bởi đườngribosetrongRNA.[16]
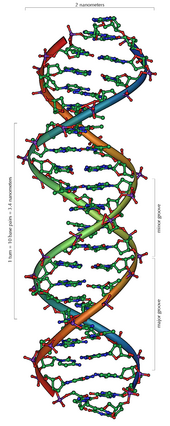
Chuỗi xoắn kép DNA được ổn định bởi hai lực liên kết chính:liên kết hydrogiữa cácnucleotidecủa hai mạch vàtương tác xếp chồng(base-stacking) giữa cácbase nitơ.[20]Bốn base trong DNA làadenine(A),cytosine(C, ở Việt Nam còn viết làxitôzin,viết tắt X),guanine(G) vàthymine(T). Bốn base này gắn với nhómđường/phosphatđể tạo thànhnucleotidehoàn chỉnh, nhưadenosine monophosphate.Adenineghép cặp vớithyminevàguanineghép cặp vớicytosine,ký hiệu bằng các cặpbaseA-T và G-C.[21][22]

Phân loại nucleobase
[sửa|sửa mã nguồn]Nucleobaseđược phân thành hai loại:purine,gồmadenine(A) vàguanine(G), làhợp chất dị vòng5 và 6 cạnh hợp nhất; vàpyrimidine,gồm cytosine (C) và thymine (T), có vòng 6 cạnh C và T.[16]Nucleobase pyrimidinethứ năm làuracil(U), thường thay thếthymine(T) trongRNAvà khác vớithyminelà thiếu đinhóm methyl(–CH3) trên vòng. NgoàiRNAvà DNA, một số lượng lớn chất tương tựacid nucleicnhân tạo được tạo ra để nghiên cứu các tính chất củaacid nucleic,hoặc sử dụng trongcông nghệ sinh học.[23]
Uracilthường không có ở DNA, nó chỉ xuất hcủacytosine.Tuy nhiên, ở một số thực khuẩn thể như: thực khuẩn thể ởBacillus subtilisPBS1 và PBS2 và thực khuẩnYersiniapiR1-37, thì thymine được thay bằng uracil.[24]Một thực khuẩn thể khác - thểStaphylococcalS6 - được phát hiện với bộ gene mà thymine thay bằng uracil.[25]
Base J(beta-d-glucopyranosyloxymethyluracil), một dạng tinh chỉnh của uracil, cũng xuất hiện ở một số sinh vật: trùng roiDiplonemavàEuglena,và mọi nhómKinetoplastida.[26]Sinh tổng hợp base J diễn ra theo hai bước: bước thứ nhất một thymidine xác định trong DNA được biến đổi thành hydroxymethyldeoxyuridine (HOMedU); bước thứ hai HOMedU đượcglycosyl hóathành base J.[27]Các nhà khoa học cũng khám phá ra những protein được tổng hợp từ base này.[28][29][30]Những protein này dường như có họ hàng xa vớigene gây ung thư(oncogene) Tet1 mà tham gia vào quá trình phát sinhbệnh bạch cầu myeloid cấp tính.[31]Base J cũng đóng vai trò làm tín hiệu kết thúc cho enzymeRNA polymerase II.[32][33]

Rãnh DNA
[sửa|sửa mã nguồn]Hai mạch đơn xoắn đôi vào nhau tạo thành bộ khung cho DNA. Ở chuỗi xoắn kép này có thể xuất hiện những khoảng trống nằm cách nhau giữa hai mạch gọi là cácrãnh(groove). Những rãnh này nằm liền kề với các cặp base và có thể hình thành mộtđiểm bám(binding site). Vì hai mạch đơn không đối xứng nhau nên dẫn đến các rãnh có kích thước không đều, trong đó rãnh lớn (major groove) rộng 22 Å và rãnh nhỏ (minor groove) rộng 12 Å.[34]Độ rộng của rãnh giúp cho các cạnh của base trở nên dễ tiếp cận hơn trong rãnh lớn so với rãnh nhỏ. Kết quả là, các protein của cácnhân tố phiên mãmà liên kết với những đoạn trình tự cụ thể trong chuỗi xoắn kép DNA thường thực hiện bằng việc tiếp xúc với các cạnh của các base ở rãnh lớn.[35]Tình huống này thay đổi đa dạng tùy theo hình dáng bất thường của DNA bên trong tế bào(xem ở dưới),nhưng các rãnh lớn và rãnh nhỏ luôn luôn được đặt tên để phản ánh sự khác nhau về kích thước đo được nếu DNA vặn xoắn trở về dạng B thường gặp.
Cặp base
[sửa|sửa mã nguồn]Trong chuỗi xoắn kép DNA, mỗi loại nucleobase trên một mạch chỉ liên kết với một loại nucleobase trên mạch kia. Đây được gọi là nguyên tắc bắt cặp bổ sung. Ở đây, purine tạoliên kết hydrovới pyrimidine, trong đó adenine chỉ liên kết với thymine bằng hai liên kết hydro, và cytosine chỉ liên kết với guanine bằng ba liên kết hydro. Sự sắp xếp giữa hai nucleotide liên kết với nhau qua chuỗi xoắn kép gọi là cặp base Watson-Crick. DNA có cặp nhiều G-C ổn định hơn DNA có ít cặp G-C.Cặp base Hoogsteenlà một dạng biến thể hiếm gặp của việc bắt cặp bổ sung. Vì liên kết hydro không phải làliên kết cộng hóa trị,nên có thể bị đứt gãy và liên kết lại tương đối dễ dàng. Hai mạch của DNA trong chuỗi xoắn kép do vậy có thể tách rời nhau bằng lực cơ học hoặc bằngnhiệt độcao, giống nhưkhóa kéo.[36]Hệ quả của nguyên tắc bắt cặp bổ sung này là mọi thông tin trong trình tự chuỗi xoắn kép DNA được lặp lại ở mỗi mạch, và có vai trò quan trọng trongquá trình nhân đôi DNA.Sự tương tác đặc hiệu và có thể hồi phục này giữa các cặp bazơ bổ sung là tối quan trọng đối với mọi chức năng của DNA trong sinh vật.[11]

|

|
Hình dưới, cặp baseA-Tcó hai liên kết hydro. Liên kết hydro không phải là liên kết cộng hóa trị được thể hiện bằng gạch ngang.
ssDNA và dsDNA
[sửa|sửa mã nguồn]Như đã nói ở trên, hầu hết phân tử DNA bao gồm hai mạch polymer, liên kết thành dạng xoắn kép bằng liên kết hydro không phải là liên kết cộng hóa trị; cấu trúc mạch kép (dsDNA- double stranded DNA) được duy trì chủ yếu bởi tương tác xếp chồng base, mạnh nhất là xếp chồng G,C. Hai mạch có thể tách rời—quá trình gọi là sự nóng chảy—tạo thành hai phân tử DNA mạch đơn (ssDNA- single-stranded DNA). Sự tách rời xảy ra ở nhiệt độ cao, độ mặn thấp vàđộ pHcao (độ pH thấp cũng làm tách DNA, nhưng vì DNA không ổn định doquá trình khử purine,do đó độ pH thấp ít khi được sử dụng).
Sự ổn định của dạng mạch kép dsDNA không chỉ phụ thuộc vào thành phầnG-C(tỷ lệ % cặp base G-C) mà còn phụ thuộc vào trình tự các base và độ dài (phân tử càng dài thì càng ổn định). Độ ổn định được đo bằng nhiều cách khác nhau; cách phổ biến là đưa phân tử đạt tớinhiệt độ nóng chảy(còn gọi là giá trịTm), đó là nhiệt độ mà tại đấy khoảng 50% số phân tử mạch kép biến đổi thành phân tử mạch đơn; nhiệt độ nóng chảy phụ thuộc vào cường độ ion và nồng độ DNA. Do vậy, cả tỷ lệ phần trăm số cặp baseG-Cvà chiều dài tổng thể của chuỗi xoắn kép DNA xác định nên cường độ liên kết giữa hai mạch DNA. Những chuỗi xoắn kép DNA dài có nhiều cặpG-Ccó tương tác giữa hai mạch mạnh hơn so với những chuỗi xoắn kép ngắn với thành phần nhiềuA-T.[37]Trong hoạt động sinh học, có những phần của chuỗi xoắn kép DNA dễ dàng tách rời, ví dụ nhưPribnow boxTATAATở một sốvùng khởi động(promoter), có xu hướng chứa nhiều thành phầnA-T,khiến cho các mạch có thể tách rời dễ dàng.[38]
Trong phòng thí nghiệm, cường độ của tương tác này có thể đo bằng cách tìm ra nhiệt độ nóng chảyTmcần thiết để phân tách một nửa liên kết hydro giữa hai mạch. Khi tất cả các cặp base nóng chảy, hai mạch của chuỗi DNA sẽ tách rời và tồn tại trong dung dịch như 2 phân tử hoàn toàn độc lập. Những phân tử mạch đơn DNA không có hình dạng chung, nhưng một số cấu trúc ổn định hơn cấu trúc khác.[39]
Có nghĩa và đối nghĩa
[sửa|sửa mã nguồn]Một trình tự DNA gọi là"có nghĩa"(sense) nếu trình tự của nó giống với trình tự của bản saoRNA thông tindùng để dịch mã thành protein.[40]Khi đó, trình tự trên mạch bổ sung còn lại được gọi là trình tự"đối nghĩa"(antisense). Cả trình tự có nghĩa và đối nghĩa có thể tồn tại trên các đoạn khác nhau của cùng một mạch đơn DNA (tức là cả hai mạch có thể chứa cả trình tự có nghĩa lẫn đối nghĩa). Ở tế bào nhân thực và nhân sơ, các trình tự RNA đối nghĩa đều được tạo ra, nhưng chức năng của những RNA này vẫn chưa được hiểu rõ hoàn toàn.[41]Có đề xuất cho rằng các RNA đối nghĩa có khả năng tham gia vào hoạt động điều hòabiểu hiện genethông qua sự bổ sung base RNA-RNA.[42]
Một vài trình tự DNA ở sinh vật nhân thực và nhân sơ, và hay gặp hơn ởplasmidvàvirus,xóa nhòa sự khác biệt giữa những mạch có nghĩa và đối nghĩa do có sự hiện diện của cácgene chồng lợp(overlapping gene).[43]Trong trường hợp này, một số trình tự DNA đảm nhận đến hai trách nhiệm, mã hóa cho một protein khi đọc dọc theo một mạch, và mã hóa protein thứ hai khi đọc theo hướng ngược lại dọc theo mạch kia. Trong vi khuẩn, sự chồng lợp này có thể tác động đến quá trình điều hòa phiên mã gene,[44]trong khi ở virus, các gene chồng lợp lại làm tăng lượng thông tin được mã hóa bên trong bộ gene nhỏ bé của virus.[45]
DNA siêu xoắn
[sửa|sửa mã nguồn]DNA có thể xoắn lại tựa như một sợi dây thừng theo một tiến trình gọi làDNA siêu xoắn(DNA supercoiling). Với DNA ở trạng thái "bình thường", một mạch thường xoắn đều quanh trục tưởng tượng của chuỗi xoắn kép theo từng đoạn ngắn mang khoảng 10,4 cặp base, nhưng nếu DNA bị vặn xoắn thì các mạch có thể trở nên siết chặt hơn hoặc lỏng lẻo hơn.[46]Nếu DNA bị xoắn theo hướng của chuỗi xoắn kép, haysiêu xoắn thuận(positive supercoiling), thì các base giữ chặt với nhau hơn. Còn nếu DNA bị xoắn ngược hướng với chuỗi xoắn kép, haysiêu xoắn nghịch(negative supercoiling), thì các base phân tách dễ dàng hơn. Trong tự nhiên, hầu hết DNA trong tế bào đều ở trạng thái gần siêu xoắn nghịch do chịu sự tác động của nhómenzymecó tên gọitopoisomerase.[47]Những enzyme này cũng cần thiết để tháo xoắn các mạch DNA trong những quá trình nhưphiên mãvànhân đôi DNA.[48]

Mô hình cấu trúc DNA khác
[sửa|sửa mã nguồn]DNA tồn tại ở nhiều cấu trúc, trong đó bao gồm dạngA,B, vàZ,thế nhưng chỉ có cấu trúc B và Z là trực tiếp quan sát thấy trong những sinh vật chức năng.[18]Cấu trúc DNA tương ứng phụ thuộc vào mức độhydrathóa, trình tự DNA, số lượng và chiều hướng siêu xoắn, các tu sửa hóa học trên base, loại và hàm lượngionkim loại, cũng như sự hiện hữu củapolyamintrong dung dịch.[49]
Báo cáo đầu tiên về các mẫu nhiễu xạ tia X của dạng A và B—họ sử dụng phương pháp phân tích dựa trênhàm Pattersonchỉ cung cấp một lượng thông tin giới hạn về cấu trúc của các sợi định hướng DNA (oriented fibers).[50][51]Một phân tích khác, do Wilkins cùng cộng sự (et al.) đề xướng vào năm 1953, cho biết các mẫu nhiễu xạ tia Xin vivoDNA dạng B của các sợi DNA hydrat hóa cao tuân theo bình phươnghàm Bessel.[52]Trong cùng tạp chí,James WatsonvàFrancis Cricktrình bày phân tíchmô hình phân tửDNA của họ các mẫu nhiễu xạ tia X và gợi ý rằng cấu trúc của DNA là chuỗi xoắn kép.[13]
Dạng B là cấu trúc phổ biến nhất tìm thấy dưới những điều kiện của tế bào sống,[53]tồn tại ở trạng thái gần giống tinh thể (paracrystalline state), đó là cấu trúc động mặc dù tính tương đối cứng của chuỗi xoắn kép DNA được giữ ổn định bởi liên kết hydro giữa các base. Để đơn giản, hầu hết những mô hình phân tử DNA đều bỏ qua liên kết động lực của nước và các ion đối với phân tử dạng B, và do đó ít hữu ích khi dùng các mô hình này để hiểu cách hoạt động của dạng B trong tế bào sống ở trạng thái bình thường (in vivo).[54]Phân tích vật lý và toán học của ảnh chụp tia X[55][56]cũng như dữ liệu quang phổ thu được cho dạng B tiền tinh thể (paracrystalline), do vậy phức tạp hơn so với dữ liệu nhiễu xạ tia X của ảnh chụp dạng A.
So với dạng B, dạng A xoắn ốc theo chiều tay phải rộng hơn, có rãnh nhỏ nông và rộng, trong khi rãnh lớn sâu hơn và hẹp hơn. Dạng A thường xuất hiện dưới các điều kiện phi sinh lý trong các mẫu DNA khử nước một phần, trong khi ở tế bào nó có thể ở dạng lai ghép 2 mạch đơn DNA với mạch đơn RNA, cũng như trong phức hệ enzym-DNA.[57][58]Ở đoạn DNA nơi các base đã bị tu sửa về mặt hóa học bằngquá trình methyl hóacó thể trải qua sự thay đổi lớn về hình dạng cấu trúc và trở thành dạngZ.Ở cấu trúc này hai mạch xoắn quanh trục theo chiều tay trái, ngược chiều với hướng của dạng B phổ biến.[59]Những cấu trúc bất thường này có thể nhận ra bằng loại protein đặc hiệu liên kết với DNA dạng Z và có thể tham gia vào quá trình điều hòa phiên mã.[60]
| Đặc tính hình học | A-DNA | B-DNA | Z-DNA |
|---|---|---|---|
| Chiều xoắn | phải | phải | trái |
| Đường kính | ≈ 2,3 nm | ≈ 2,0 nm | ≈ 1,8 nm |
| Đơn vị lặp lại | 1 bp | 1 bp | 2 bp |
| Góc quay/bp | 32,7° | 34,3° | 60°/2 |
| Số bp trung bình/vòng xoắn | 11 | 10,4 | 12 |
| Độ nghiêng của bp so với trục | +19° | -1,2° | -9° |
| Độ dài dốc/bp dọc theo trục | 0,23 nm | 0,332 nm | 0,38 nm |
| Bước/vòng xoắn | 2,82 nm | 3,32 nm | 4,56 nm |
| Góc xoắn trung bình giữa hai bp | +18° | +16° | 0° |
| Góc glycosyl | anti | anti | Pyrimidine: anti Purine: syn |
| Chế độ gấp phân tử đường (sugar puckering) |
C3'-endo | C2'-endo | Pyrimidine: C2'-endo Purine: C3'-endo |
| Rãnh lớn | hẹp và sâu | rộng và sâu, độ sâu: 0,85 nm | phẳng |
| Rãnh nhỏ | rộng và phẳng | hẹp và sâu, độ sâu: 0,75 nm | hẹp và sâu |
DNA có thành phần hóa học thay thế
[sửa|sửa mã nguồn]Trong một vài năm, cácnhà sinh học vũ trụđã đề xuất về một sinh quyển bóng tối (shadow biosphere), một sinh quyển vi sinh vật giả thuyết tồn tại trên Trái Đất mà sử dụng các quá trình phân tử và hóa học khác căn bản so với những gì đã biết về sự sống hiện tại. Một trong các đề xuất đó là sự tồn tại của dạng sinh vật sống mà nguyên tửasenthay chophosphotrong DNA. Một báo cáo năm 2010 cho thấy khả năng này có mặt trong vi khuẩnGFAJ-1,[64][64][65]mặc dù đã có những tranh cãi,[65][66]và cuối cùng năm 2012 một báo cáo khác nêu ra bằng chứng cho thấy các vi khuẩn này chủ động ngăn không cho asen kết hợp vào bộ khung DNA của nó và những phân tử sinh học khác.[67]
Cấu trúc 4 sợi
[sửa|sửa mã nguồn]Tại đầu mút của mỗi nhiễm sắc thể tuyến tính có những vùng chuyên biệt của DNA gọi làtelomere.Chức năng chính của các vùng này đó là cho phép tế bào tái bản các đầu mút nhiễm sắc thể bằng cách sử dụng enzymtelomerase,bởi vì bình thường các enzym tái bản DNA không thể nhân đôi đầu cực 3′ của nhiễm sắc thể.[68]Những đầu mũ đặc hiệu này của nhiễm sắc thể cũng giúp bảo vệ đầu cực của DNA bị rút ngắn, và ngăn hệ thốngsửa chữa DNAtrong tế bào coi DNA bị hỏng cần được sửa chữa.[69]Trong tế bào người, các telomere thường là những đoạn mạch đơn DNA chứa vài nghìn trình tự TTAGGG lặp đi lặp lại đơn giản.[70]
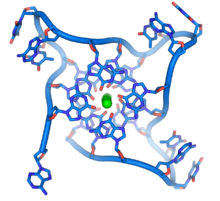
Các trình tự giàu guanine giữ ổn định đầu mút của nhiễm sắc thể bằng cách hình thành nên cấu trúc gồm những đơn vị bốn base xếp chồng, hơn là bắt cặp bổ sung tìm thấy ở các phân tử DNA khác. Ở đây, bốn base, được gọi là 4guanin tetrad,tạo thành một tấm phẳng. Sau đó, bộ 4 base phẳng này xếp chồng lẫn nhau hình thành nên cấu trúcG-quadruplex(cấu trúc 4 sợi) ổn định.[72]Cấu trúc này được ổn định bởi liên kết hydro vàchelat hóaion kim loại nằm ở trung tâm của mỗi đơn vị bốn base.[73]Những cấu trúc khác cũng có thể tồn tại, trung tâm của bộ bốn base đến từ một mạch đơn uốn quanh các base, hoặc là một vài mạch song song với nhau, trong đó mỗi mạch đều đóng góp một base vào cấu trúc trung tâm.
Bên cạnh cấu trúc xếp chồng, telomere cũng tạo thành cấu trúc dạng vòng lớn gọi là telomere loop, hay T-loop. Trong cấu trúc này, DNA mạch đơn cuộn quanh thành một vòng tròn dài được ổn định bởi các protein liên kết với telomere.[74]Tại đầu mút của T-loop, DNA telomere mạch đơn được cho vào một vùng bao bởi DNA mạch kép bằng mạch telomere phân tách DNA mạch kép và bắt cặp bổ sung với một trong hai mạch. Cấu trúcmạch banày (triple-stranded) được gọi là vòng 3 sợi (displacement loop) hayD-loop.[72]
DNA phân nhánh
[sửa|sửa mã nguồn]Ở chuỗi xoắn kép DNA, hiện tượngsờn tước đầu mútxuất hiện khi những đoạn không được bổ sung hiện diện tại đầu mút của DNA mạch kép. Qua đó, DNA phân nhánh có thể hình thành nếu có một mạch DNA thứ ba xuất hiện và mang những đoạn mới liên hợp với đoạn không được bổ sung của chuỗi xoắn kép đã bị sờn tước trước đó. Dạng đơn giản nhất của DNA phân nhánh chỉ bao gồm ba mạch DNA, tất nhiên là có thể tồn tại thêm nhiều nhánh phức tạp khác.[75]DNA phân nhánh được ứng dụng trongcông nghệ nanođể lắp ráp những cấu hình phân tử mong muốn.
Tu sửa base và trình tự của DNA
[sửa|sửa mã nguồn]
|

|

|
| Cytosine | 5-methylcytosine | Thymine |
Tu sửa base và bao gói DNA
[sửa|sửa mã nguồn]Biểu hiện của gene chịu ảnh hưởng bởi phương cách đóng gói DNA trong nhiễm sắc thể, thành một cấu trúc gọi làchất nhiễm sắc(chromatin). Những tác động chỉnh sửa base có thể xảy ra trong quá trình đóng gói, với các vùng không có hoặc có mức biểu hiện gene thấp thông thường chứa các base cytosine ở mứcmethyl hóacao. Sự đóng gói DNA và ảnh hưởng của nó lên biểu hiện gene cũng xảy ra bởi hiệu ứng thay đổi liên kết cộng hóa trị tại lõi proteinhistonebọc quanh DNA trong cấu trúc chất nhiễm sắc hoặc bởi phức hệ chất nhiễm sắc tái mô hình hóa (xemTái mô hình hóa chất nhiễm sắc(chromatin remodeling)). Do vậy, tác độngxen lẫngiữa methyl hóa DNA và thay đổi liên kết ở histone có ảnh hưởng phối hợp đến chất nhiễm sắc và biểu hiện gene.[76]
Ví dụ, sự methyl hóa cytosine, tạo ra5-methylcytosine,có vai trò quan trọng đối với sựbất hoạt Xcủa nhiễm sắc thể (X-inactivation).[77]Mức độ methyl hóa trung bình thay đổi theo mỗi sinh vật – giun trònCaenorhabditis eleganskhông có phản ứng methyl hóa cytosine, trong khi ởđộng vật có xương sốngcó mức độ cao hơn, lên tới 1% lượng DNA chứa 5-methylcytosine.[78]Tuy 5-methylcytosine có vai trò quan trọng, nhưng nó vẫn có thể bị khử amin hóa để chuyển thành base thymine, do đó cytosine methyl hóa có khuynh hướng gâyđột biến.[79]Những thay đổi base khác bao gồm sự methyl hóa adenine ở vi khuẩn, sự hiện diện của5-hydroxymethylcytosinetrongnão,[80]và sựglycosyl hóacủa uracil tạo thành "Base J" trong các loàiKinetoplastida.[81][82]
Hư hại
[sửa|sửa mã nguồn]
DNA có thể bị hư hại bởi nhiềutác nhân đột biến,làm thay đổitrình tự DNA.Những tác nhân đột biến bao gồmcác chất oxy hóa,ankyl hóavới cảbức xạ điện từnăng lượng cao nhưtia cực tímvàtia X.Loại hư hại DNA được tạo ra phụ thuộc vào loại tác nhân đột biến. Ví dụ, tia UV có thể phá hủy DNA bằng cách tạo radimer thymine,nghĩa là liên kết chéo giữa các base của pyrimidine với nhau.[84]Mặt khác, những tác nhân oxy hóa nhưgốc tự dohayhydro peroxidetạo ra nhiều dạng hư hại, bao gồm tu sửa base, đặc biệt làguanosine,và đứt gãy chuỗi xoắn kép.[85]Một tế bào điển hình ở người chứa khoảng 150.000 base chịu tổn thương do bị oxy hóa.[86]Trong số những tổn thương oxy hóa, nguy hiểm nhất đó là chuỗi xoắn kép bị đứt gãy, vì rất khó để gắn lại và có thể dẫn tớiđột biến mất,thêmvà thay thế trên trình tự DNA, cũng như làchuyển đoạn nhiễm sắc thể(chromosomal translocation).[87]Những đột biến này có thể gây raung thư.Bởi vì những giới hạn vốn có trong cơ chếsửa chữa DNA,nên nếu con người sống đủ lâu, thì những hư hại này cuối cùng sẽ dẫn tới sự phát triển của ung thư.[88][89]Những hư hại DNAxảy ra tự nhiên,là do các quá trình bình thường trong tế bào tạo ra cácgốc tự do oxy hóa(ROS), chẳng hạn các phản ứng thủy phân của nước trong tế bào, v.v, cũng xảy ra thường xuyên. Mặc dù hầu hết những hư hại này đều sẽ được sửa chữa, nhưng trong bất kỳ tế bào nào vẫn có một số hư hại DNA có thể còn tồn tại mặc cho các hoạt động của quá trình sửa chữa. Những hư hại DNA còn sót lại sẽ tích tụ dần theo độ tuổi bên trong các mô hậu nguyên phân của động vật có vú. Sự tích tụ này dường như là một nguyên nhân quan trọng dẫn tới sự già yếu.[90][91][92]
Nhiều tác nhân đột biến nằm gọn trong không gian giữa hai cặp base liền kề, hay gọi làsự xen kẹp(intercalation). Hầu hết các chất xen kẹp là những phân tửvòng thơmvà vòng phẳng; ví dụ như:ethidium bromide,acridine,daunomycinvàdoxorubicin.Để cho một chất xen kẹp có thể vừa vặn không gian giữa hai cặp base, các base phải tách ra, bóp méo mạch DNA bằng cách tháo xoắn chuỗi xoắn kép. Điều này khiến ức chế quá trình phiên mã và nhân đôi DNA, gây ra độc tính và đột biến.[93]Do đó, các phân tử xen kẹp vào DNA có thể làtác nhân gây ung thư,và trong trường hợp củathalidomidelàtác nhân gây quái thai(teratogen).[94]Những phân tử khác nhưbenzo[a]pyrene diol epoxidevàaflatoxintạo thành sản phẩm cộng vào DNA gây ra lỗi trong quá trình nhân đôi.[95]Tuy nhiên, do khả năng ức chế phiên mã và nhân đôi DNA, những độc tố tương tự khác cũng được sử dụng tronghóa trị liệuđể ức chế phát triển nhanh chóng của cáctế bào ung thư.[96]
Chức năng sinh học
[sửa|sửa mã nguồn]
DNA thông thường hiện diện trong nhiễm sắc thể dạng thẳng ở sinh vật nhân thực, và nhiễm sắc thể dạng vòng ở sinh vật nhân sơ.Nhiễm sắc thể(chromosome) thực chất làchất nhiễm sắc(chromatin) bị co xoắn từ kỳ đầu của quá trình phân bào. Còn chất nhiễm sắc chính là phức hợp giữa chuỗi xoắn kép DNA với các protein histone và phi histone gói gọn thành một cấu trúc cô đặc. Điều này cho phép các phân tử DNA rất dài nằm gọn trong nhân tế bào. Cấu trúc vật lý của nhiễm sắc thể và chất nhiễm sắc thay đổi luân phiên tùy thuộc vào từng giai đoạn của chu kỳ tế bào. Tập hợp các nhiễm sắc thể trong một tế bào tạo thànhbộ genecủa nó;bộ gene ngườicó xấp xỉ 3 tỷ cặp base DNA xếp thành 46 nhiễm sắc thể.[97]Thông tin chứa trong DNA tổ chức dưới dạngtrình tựcủa các đoạn DNA gọi làgene.Sựkế thừathông tin di truyền trong gene được thực hiện thông qua các cặp base bổ sung. Ví dụ, trong quá trình phiên mã, khi một tế bào sử dụng thông tin ở một gene, trình tự DNA sẽ được sao mã vào trình tự bổ sung RNA thông qua lực hút giữa DNA và các nucleotide chính xác của RNA. Thông thường, bản sao RNA này được dùng làm khuôn mẫu để xác địnhtrình tựcác amino acid trong quá trình dịch mã, thông qua sự tương tác giữa các nucleotide RNA. Trong quá trình khác, một tế bào có thể tự sao chép thông tin di truyền của nó bằng quá trình nhân đôi DNA. Chi tiết của những chức năng này được nêu trong những bài viết liên quan; bài này tập trung vào tương tác giữa DNA và các phân tử khác mà đảm trách các chức năng của bộ gene.
Gene và bộ gene
[sửa|sửa mã nguồn]
DNA chứa các đoạn gene được gói gọn và xếp chặt có thứ tự bởi quá trìnhcô đặc DNA(DNA condensation),để có thể vừa vặn trong một thể tích nhỏ của tế bào. Ở sinh vật nhân thực, DNA nằm trongnhân tế bào,cùng với một lượng nhỏ nằm trongty thểvàlục lạp.Ở sinh vật nhân sơ, DNA nằm trong một thể có hình dạng không đều giữa tế bào chất, gọi làthể nhân(hoặcvùng nhân,nucleoid).[98]Thông tin di duyền trong một bộ gene được lưu trữ bởi các gene, và tập hợp toàn bộ các gene trong tế bào của cơ thể thuộc một loài sinh vật được gọi làkiểu gene.Mỗi gene là một đơn vị củatính di truyềnvà là một đoạn của DNA có ảnh hưởng tới một đặc tính cụ thể trong cơ thể sinh vật. Các gene chứa mộtkhung đọc mở(open reading frame) có thể được phiên mã, cùng với các vùngtrình tự điều hòa(regulatory sequence) nhưvùng khởi động(promoter) vàvùng tăng cường(enhancer) có khả năng điều hòa quá trình phiên mã của khung đọc mở.
Ở nhiềuloài,chỉ một phần nhỏ trong tổng số trình tự của bộ gene là mã hóa cho protein. Ví dụ, chỉ khoảng 1,5% bộ gene người chứa các đoạnexonmã hóa cho protein, trong khi trên 50% DNA ở người chứa cáctrình tự lặp lạikhông mã hóa (non-coding repeated sequence).[99]Những lý do cho sự có mặt của rất nhiềuDNA không mã hóaở bộ gene của sinh vật nhân thực và sự cách biệt rất lớn trongkích cỡ bộ gene,haygiá trị C,giữa các loài đã đưa đến một vấn đề nan giải lâu năm gọi là "nghịch lý giá trị C".[100]Tuy nhiên, một số trình tự DNA không mã hóa protein vẫn có thể có chức năng mã hóa các phân tửRNA không mã hóatham gia vào quá trìnhđiều hòa biểu hiện gene.[101]

Một số trình tự DNA không mã hóa đóng vai trò cấu trúc bộ khung trong nhiễm sắc thể.Telomerevàtâm động(centromere) điển hình chỉ chứa vài gene, nhưng lại có vai trò quan trọng đối với chức năng và sự ổn định của nhiễm sắc thể.[69][103]Một dạng DNA không mã hóa xuất hiện ở người gọi làgene giả(pseudogene), là những bản sao của gene nhưng đã bị bất hoạt do tác động của đột biến.[104]Những trình tự này thường chỉ là cáchóa thạchphân tử, mặc dù chúng có thể phục vụ như là vật liệu di truyền dạng thô cho sự sản sinh gene mới thông qua quá trìnhnhân đôi(gene duplication) vàphân lygene.[105]
Phiên mã và dịch mã
[sửa|sửa mã nguồn]
Mỗi gene là một đoạn trình tự DNA chứa thông tin di truyền và có thể ảnh hưởng đếnkiểu hìnhcủa sinh vật. Bên trong một gene, trình tự các base dọc theo một mạch DNA xác định nên trình tự củaRNA thông tin,rồi từ đó xác lập nên trình tự của một hay nhiều protein. Mối liên hệ giữa trình tự nucleotide của các gene và trình tự cácamino acidcủa protein được xác định bởi những quy tắc trong quá trìnhdịch mã,được biết đến với cái tên bộmã di truyền.Mỗi mã di truyền chứa bộ ba 'chữ cái' gọi làtriplet(bộ ba mã gốc) trên DNA haycodon(bộ ba mã sao) trên mRNA hayanticodon(bộ ba đối mã) trên tRNA tạo thành một trình tự gồm ba nucleotide (v.d. ACT, CAG, TTT trên mạch gốc DNA).
Trong quá trình phiên mã, các triplet của một gene được sao chép sang RNA thông tin thành các codon tương ứng bằng enzymeRNA polymerase.Bản sao RNA này sau đó được giải mã bởiribosomethông qua hoạt động đọc trình tự RNA bằng cách bổ sung cặp base trong RNA thông tin vớiRNA vận chuyển,loại phân tử mang theo amino acid. Vì có bốn loại base khác nhau được tổ hợp thành các mã bộ ba, do vậy có tất cả 64 codon (tổ hợp 43). Tất cả chúng được phân bổ để mã hóa cho 20 loạiamino acid cơ bảncủa sự sống, do đó một amino acid có thể có nhiều hơn một codon mã hóa cho nó. Bên cạnh đó cũng có ba codon 'kết thúc' hoặc 'vô nghĩa' (nonsense) đánh dấu điểm kết thúc của một vùng mã hóa; chúng là các codon UAA, UAG và UGA (tương ứng với các triplet TAA, TAG và TGA).
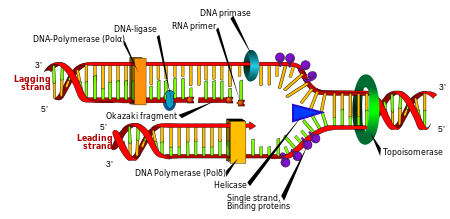
Nhân đôi DNA (sao chép, tái bản)
[sửa|sửa mã nguồn]Phân bàolà quá trình cơ bản của sinh vật để có thể sinh trưởng, nhưng khi một tế bào phân chia, nó phải nhân đôi DNA trong bộ gene của nó sao cho hai tế bào con có cùng thông tin di truyền như của tế bào mẹ. Cấu trúc mạch kép DNA giúp hình thành một cơ chế đơn giản cho quá trìnhnhân đôiDNA. Ở đây, hai mạch đơn tháo xoắn tách rời nhau và mỗi mạch mới bổ sung với mỗi mạch gốc được tổng hợp bằng một loạienzymegọi làDNA polymerase.Enzyme này tạo ra những mạch mới bằng cách tìm những nucleotide tự do từ môi trường nội bào và gắn kết chính xác với nucleotide trên mạch gốc ban đầu theo nguyên tắc bổ sung. Vì DNA polymerase chỉ tổng hợp mạch mới theo chiều 5′ → 3′, do vậy trên mạch khuôn có chiều 3' → 5' thì mạch bổ sung được tổng hợp liên tục do cùng chiều với chiều tháo xoắn.[106]Còn trên mạch khuôn có chiều 5' → 3' thì mạch bổ sung được tổng hợp ngắt quãng tạo nên các đoạn ngắn gọi làđoạn Okazakido ngược chiều với chiều tháo xoắn, sau đó các đoạn này được nối lại với nhau nhờenzymenốiDNA ligase.[107]
acid nucleic ngoại bào
[sửa|sửa mã nguồn]DNA ngoại bào trần (extracellular DNA -eDNA), hầu hết được giải phóng khi tế bào chết đi, xuất hiện khắp nơi trong môi trường. Mức độ tập trung của nó trong đất có thể lên tới 2 μg/lít, và trong môi trường nước tự nhiên lên tới 88 μg/lít.[108]Đã có một số chức năng khả thi của eDNA được đề xuất: nó có thể tham gia vàochuyển gene ngang;[109]cung cấp dinh dưỡng;[110]và có khả năng hoạt động như một chất đệm để khôi phục hoặc chuẩn độ ion hoặc tính kháng sinh.[111]DNA ngoại bào hoạt động như một thành phần chức năng của chất nền ngoại bào trong lớpmàng vi sinh vật(phim sinh học - biofilm) của một số loài vi khuẩn. Nó có thể hoạt động như một nhân tố nhận diện để điều phối sự bám dính và phân tán của một số loại tế bào đặc hiệu trong phim sinh học;[112]hoặc đóng góp vào sự hình thành phim sinh học;[113]cũng như đóng góp vào đặc tính vật lý chắc chắn của phim sinh học và sức đề kháng trước những căng thẳng sinh học (biological stress).[114]
Tương tác với protein
[sửa|sửa mã nguồn]Mọi chức năng của DNA phụ thuộc vào tương tác với protein. Nhữngtương tác proteinnày có thể không đặc hiệu hoặc đặc hiệu khi protein liên kết với một trình tự DNA cụ thể. Các enzyme cũng liên kết với DNA và trong số này, những enzyme polymerase sao chép trình tự base của DNA trong quá trình phiên mã và nhân đôi DNA có vai trò đặc biệt quan trọng.
Protein liên kết DNA
[sửa|sửa mã nguồn]
|
Các protein cấu trúc liên kết với DNA là những ví dụ đã được nghiên cứu khá kĩ về tương tác không đặc hiệu DNA-protein. Bên trong nhiễm sắc thể, DNA được giữ trong phức hợp với protein cấu trúc. Những protein này tổ chức DNA thành một cấu trúc thắt đặc gọi làchất nhiễm sắc(chromatin). Trong sinh vật nhân thực, cấu trúc này bao gồm DNA liên kết với phức hợp các đơn vị protein cơ sở nhỏ gọi làhistone,trong khi ở sinh vật nhân sơ lại có nhiều loại protein tham gia hơn.[115][116]Các histone tạo thành một phức hợp dạng đĩa gọi lànucleosome,với chuỗi xoắn kép DNA bao quanh bề mặt cấu trúc bằng hai vòng xoắn. Những tương tác không đặc hiệu được hình thành thông qua các phần dư cơ bản trong histone, tạo raliên kết ionvới bộ khung đường-phosphat có tính acid của DNA, và do vậy phần lớn tương tác là độc lập với trình tự các base.[117]Những phản ứng hóa học làm thay đổi các amino acid cơ bản này bao gồm phản ứngmethyl hóa,phosphoryl hóavàacethyl hóa.[118]Những thay đổi hóa học này làm ảnh hưởng tới cường độ tương tác giữa DNA và histone, khiến cho cácnhân tố phiên mãtrở nên dễ dàng hoặc khó tiếp cận được với DNA và do vậy thay đổi tốc độ quá trình phiên mã.[119]Những protein liên kết DNA không đặc hiệu khác trong chất nhiễm sắc bao gồm các nhóm protein có tính linh động cao mà khi liên kết có thể uốn hoặc làm vặn DNA.[120]Các protein này có vai trò quan trọng trong việc sắp uốn nucleosome và xếp đặt chúng thành những cấu trúc lớn hơn tạo thành nhiễm sắc thể.[121]
Có một nhóm protein liên kết DNA đặc biệt là các protein chỉ liên kết đặc hiệu với một mạch đơn DNA. Ở người,protein Aphục vụ quá trình nhân đôi DNA là protein được hiểu biết rõ ràng nhất trong nhóm này và tham gia vào những quá trình khi hai mạch xoắn kép đã tách rời nhau, bao gồm sao chép DNA, tái tổ hợp và sửa chữa DNA.[122]Những protein liên kết này giúp ổn định hóa mạch đơn DNA và bảo vệ nó khỏi hiện tượng hình thành cấu trúcvòng gấp kẹp tóc(stem-loop/hairpin loop) hoặc bị phân cắt bởi enzymenuclease.

Ngược lại, có những protein khác phải biến đổi cấu hình để liên kết với những trình tự DNA riêng biệt. Lĩnh vực nghiên cứu sâu rộng nhất về những protein này đó là nghiên cứu nhiều loạinhân tố phiên mã(transcription factor) khác nhau, đây chính là các protein điều hòa quá trình phiên mã. Mỗi nhân tố phiên mã liên kết với một tập hợp cụ thể các trình tự DNA và kích hoạt hoặc ức chế hoạt động phiên mã của gene tại những trình tự gần vớivùng khởi độngcủa chúng. Nhân tố phiên mã thực hiện vai trò này theo hai cách. Đầu tiên, chúng có thể gắn với RNA polymerase chịu trách nhiệm cho quá trình phiên mã, hoặc trực tiếp hoặc gián tiếp thông qua các protein trung gian; giúp định vị polymerase tại vùng gene khởi động và cho phép bắt đầu phiên mã.[124]Hoặc cách khác, nhân tố phiên mã có thể gắn vớienzymelàm biến đổi các histone ở vùng khởi động. Điều này làm thay đổi khả năng tiếp cận của polymerase với mạch khuôn DNA.[125]
Do những DNA đích này xuất hiện trong toàn thể bộ gene sinh vật, vì vậy những thay đổi trong hoạt động của một loại nhân tố phiên mã có thể ảnh hưởng tới hàng nghìn gene.[126]Hệ quả là, những protein này thường là mục tiêu của các quá trìnhtruyền tín hiệu tải nạp(signal transduction) mà điều khiển sự đáp ứng đối với những thay đổi của môi trường hoặcbiệt hóa tế bàovà điều khiển sự phát triển. Nét đặc trưng của những tương tác của các nhân tố phiên mã với DNA đến từ các protein tạo nhiều tiếp xúc với các cạnh của các base DNA, cho phép chúng "đọc" được trình tự DNA. Phần lớn những tương tác với base diễn ra ở rãnh lớn, nơi có thể tiếp xúc nhiều nhất với các base.[35]

Enzyme chỉnh sửa DNA
[sửa|sửa mã nguồn]Nuclease và ligase
[sửa|sửa mã nguồn]Nucleaselà cácenzymecó khả năng cắt mạch DNA bằng cách xúc tác cho phản ứngthủy phâncácliên kết phosphodieste.Loại nuclease thủy phân nucleotide từ những đầu mút của mạch DNA được gọi làexonuclease,trong khiendonucleaselại phân cắt từ những điểm trong mạch. Những nuclease được sử dụng thường xuyên nhất trongsinh học phân tửlà cácendonuclease giới hạn,do chúng cắt DNA tại những đoạn trình tự đặc hiệu. Ví dụ,enzyme EcoRVở hình ảnh bên trái nhận ra trình tự gồm 6 base 5′-GATATC-3′ và thực hiện việc cắt theo một đường nằm ngang. Trong tự nhiên, nhữngenzymenày bảo vệvi khuẩnchống lại sự tấn công củathể thực khuẩnbằng cách tiêu hóa DNA thể thực khuẩn khi chúng xâm nhập vào tế bào vi khuẩn, lúc này cácenzymehoạt động như một phần tronghệ thống hạn chế cải biến(restriction modification system).[128]Trong công nghệ sinh học, những nuclease hoạt động với các trình tự đặc hiệu được sử dụng trongtách dòng phân tử(molecular cloning) vàkỹ thuật nhận diện DNA(DNA profiling).

a: mạch khuôn, b: mạch dẫn đầu(leading strand),c: mạch theo sau(lagging strand),d: chạc tái bản, e: đoạn mồi RNA, f: các đoạn Okazaki
Những enzyme có chức năng nối lại những đoạn DNA bị cắt hoặc bị đứt gãy được gọi làDNA ligase.[129]Ligaseđặc biệt quan trọng trong việc nối lại cácmạch theo saungắt quãng của DNA, tức là các đoạn Okazaki tại chạc tái bản thành một bản sao hoàn chỉnh từ mạch khuôn DNA. Chúng cũng tham gia vào việcsửa chữa DNAvàtái tổ hợp di truyền.[129]
Topoisomerase và helicase
[sửa|sửa mã nguồn]- Topoisomeraselà nhữngenzymemang hoạt tính của cảnucleaselẫnligase.Nhữngproteinnày có khả năng thay đổi cấu trúc chuỗi xoắn kép DNA: chúng có thể thoái bỏ trạng thái siêu xoắn, hoặc ngược lại, chúng đóng xoắn. Một sốenzymetrong nhóm này thực hiện hoạt động cắt chuỗi xoắn kép DNA và cho phép một phần phân tử quay được, do vậy làm giảm mức siêu xoắn của nó; sau cuối enzyme sẽ gắn khít hoàn chỉnh lại đoạn DNA bị gãy.[47]Những loạienzymekhác có thể cắt một chuỗi xoắn kép DNA và rồi kéo một mạch DNA thứ hai vào vị trí cắt này, trước khi thực hiện việc nối lại chuỗi xoắn kép.[130]Topoisomerase cần thiết cho nhiều quá trình liên quan đến DNA, như nhân đôi và phiên mã.[48]
- Helicaselà những protein thuộc một trong những loạiđộng cơ phân tử.Chúng sử dụng năng lượng hóa học trongnucleoside triphosphat,thường sử dụng nhất làadenosine triphosphat(ATP), để phá vỡ liên kết hydro giữa các base và tháo xoắn chuỗi kép DNA thành hai mạch đơn.[131]Nhữngenzymenày có vai trò quan trọng thiết yếu đối với hầu hết quá trìnhenzymecần thiết có tương tác với các base nitơ.
Polymerase
[sửa|sửa mã nguồn]
Polymeraselà nhữngenzymethực hiện tổng hợp mạchpolynucleotidetừnucleoside triphosphat.Tính tuần tự của các sản phẩm của chúng được sinh ra dựa trên những mạchpolynucleotideđã có—gọi làmạch khuôn.Nhữngenzymenày hoạt động bằng lần lượt thêm vào một nucleotide tại nhóm 3′hydroxylở điểm cuối của mạchpolynucleotideđang phát triển. Kết quả là, mọipolymerasehoạt động luôn theo chiều từ đầu 5′ đến đầu 3′.[132]Tạitrung tâm hoạt độngcủa cácenzymenày, phân tửnucleoside triphosphatđi đến ghép cặp vớibasecủa mạch khuôn: điều này cho phép polymerase tổng hợp một cách chính xác mạch bổ sung đối với mạch khuôn của nó. Các polymerase được phân loại theo các nhóm mạch khuôn mà chúng sử dụng.
Trong quá trình sao chép DNA,DNA polymerasephụ thuộc DNA tạo nên những bản sao của những mạchpolynucleotide DNA.Để bảo toàn thông tin sinh học, điều cơ bản là trình tự của cácbasetrong mỗi bản sao là trình tự bổ sung chính xác cho trình tự base trong mạch khuôn mẫu. NhiềuDNA polymerasecó hoạt tínhđọc và sửa sai(proofreading). Ở đây, polymerase nhận ra các lỗi thường xuất hiện trong phản ứng tổng hợp do sự thiếu đi những base ghép cặp giữa các nucleotide không khớp với nhau. Nếu polymerase phát hiện một sự không ăn khớp, hoạt tínhexonuclease3'-5′ được kích hoạt và base không khớp nào được phát hiện sẽ bị cắt bỏ.[133]Trong hầu hết các sinh vật,DNA polymerasehoạt động trong một phức hệ lớn gọi làreplisomecó chứa nhiều tiểu đơn vị phụ, nhưprotein kẹp DNA(DNA clamp) hayhelicase.[134]
DNA polymerasephụ thuộcRNAlà những loạipolymerasechuyên biệt thực hiện sao chép trình tự của mạchRNAsang DNA. Chúng bao gồmenzyme phiên mã ngược(reverse transcriptase, RT), ví dụ như một enzyme của virutretrovirustham gia vào quá trình xâm nhập tế bào, vàtelomerase,cần cho quá trình sao chép telomere.[68][135]Telomerase là một polymerase khác thường bởi vì nó chứa chính mạch khuôn RNA của nó như là một phần trong cấu trúc của enzyme này.[69]
Sự phiên mã được thực hiện bởiRNA polymerasephụ thuộc DNA thông qua quá trình sao chép trình tự của mạch DNA sangRNA.Để bắt đầu giải mã mộtgene,RNA polymerasegắn với một trình tự của DNA gọi làvùng khởi động(promoter) và tách hai mạch DNA khỏi nhau. Sau đó nó sao chép trình tựgenevào mộtRNA thông tincho đến khi nó đi đếnvùng kết thúc(terminator) của DNA, nơiRNA polymerasedừng lại và tách khỏi DNA. VớiDNA polymerasephụ thuộc DNA ở người,RNA polymerase II,enzymethực hiện phiên mã hầu hết cácgenetrong bộgenengười, hoạt động như là một phần của mộtphức hệ proteinlớn với nhiều tiểu đơn vị phụ và vùng điều hòa khác nhau.[136]
Tái tổ hợp di truyền
[sửa|sửa mã nguồn]
|

|
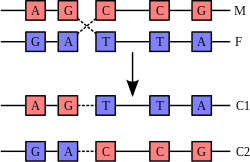
Chuỗi xoắn kép DNA thường không tương tác với những đoạn khác của DNA, và trong tế bào người các nhiễm sắc thể khác nhau thậm chí còn nằm ở những vùng tách biệt trong nhân tế bào gọi là "vùng nhiễm sắc thể"(chromosome territory).[138]Sự tách biệt về không gian giữa các nhiễm sắc thể khác nhau là quan trọng đối với khả năng hoạt động của DNA như là nơi lưu giữ ổn định thông tin di truyền, khi một vài lần nhiễm sắc thể tương tác trong sựtrao đổi chéo nhiễm sắc thểxảy ra trong quá trìnhsinh sản hữu tính,khi ấy tái tổ hợp di truyền mới diễn ra. Trao đổi chéo nhiễm sắc thể là khi hai chuỗi DNA tháo xoắn và tách rời từng mạch đơn ra, trao đổi các đoạn DNA cho nhau rồi tái gắn kết hai mạch đơn lại.
Tái tổ hợp cho phép nhiễm sắc thể trao đổi thông tin di truyền và tạo ra những tổ hợp gene mới, làm tăng hiệu quả của tínhchọn lọc tự nhiênvà có thể quan trọng đối với sự tiến hóa nhanh chóng cho những protein mới.[139]Tái tổ hợp di truyền cũng bao gồm trong quá trình sửa chữa DNA, đặc biệt trong sự đáp ứng của tế bào đối với sự kiện chuỗi xoắn kép bị đứt gãy.[140]
Dạng phổ biến nhất của trao đổi chéo nhiễm sắc thể làtái tổ hợp tương đồng,khi hai nhiễm sắc thể tham gia quá trình trên có trình tự DNA tương đồng. Tái tổ hợp không tương đồng có thể phá hủy tế bào, gây rachuyển đoạn nhiễm sắc thểvà biến dị di truyền. Phản ứng tái tổ hợp được xúc tác bởi các enzymerecombinase,nhưRAD51.[141]Bước đầu tiên trong quá trình tái tổ hợp là một chuỗi DNA bị đứt gãy do tác động bởi enzymeendonucleasehay những phá hủy đối với DNA.[142]Một loạt các bước tiếp theo có sự xúc tác một phần của recombinase, sau đó hai chuỗi xoắn kép nối lại tại ít nhất mộtđiểm giao Holliday(Holliday junction), trong đó một đoạn của mạch đơn của chuỗi xoắn kép này được ghép nối với đoạn mạch đối ứng của chuỗi xoắn kép kia. Điểm giao Holliday là một cấu trúc tiếp xúc bốn nhánh mà có thể di chuyển dọc theo cặp nhiễm sắc thể, tráo đổi một mạch sang cho mạch khác. Phản ứng tái tổ hợp dừng lại khi điểm giao Holliday bị đứt và xảy ra quá trình hàn gắn lại chuỗi DNA được giải phóng.[143]
Tiến hóa
[sửa|sửa mã nguồn]DNA chứa thông tin di truyền cho phép tất cả dạng sống hiện đại hoạt động chức năng, sinh trưởng và sinh sản. Tuy nhiên, không rõ bao lâu trong hành trìnhlịch sử4 tỷ năm của sự sống DNA đã bắt đầu đảm nhận chức năng này, vì có những đề xuất cho rằng các dạng sống xuất hiện sớm nhất có khả năng đã sử dụng phân tử RNA thay vì DNA làm vật liệu di truyền.[144][145]RNA có thể đã trở thành thành phần trung tâm của quá trìnhtrao đổi chấttrong những tế bào sơ khai vì phân tử này có thể vừa truyền đạt thông tin di truyền cũng như mang hoạt tínhxúc tácphản ứng dưới dạngribozyme.[146]Thế giới RNAcổ xưa này, một nơiacid nucleicđược sử dụng cho cả quá trình xúc tác và di truyền, có thể ảnh hưởng đến sựtiến hóacủa hệ thống mã di truyền hiện tại trên cơ sở bốn loại nucleobase. Điều này thực sự đã xảy ra, vì số lượng của những base khác nhau trong một cơ thể sống như là một sự thỏa hiệp giữa một số lượng nhỏbasetăng cường qua hoạt động nhân đôi chính xác và một số lượng lớn những base tăng cường qua hoạt động xúc tác hiệu quả củaribozyme.[147]Không may thay, thực tế lại không có bằng chứng trực tiếp nào của hệ thống di truyền cổ xưa, như việc phục hồi DNA từ phần lớn các hóa thạch là điều không thể vì phân tử DNA chỉ tồn tại trong môi trường ít hơn một triệu năm và dần dần phân hủy thành những mảnh ngắn tan vào dung dịch.[148]Những yêu cầu khảo sát đối với dạng DNA cổ xưa đã được thực hiện, trong đó báo cáo đáng chú ý nhất là về sự cô lập của một loại vi khuẩn tồn tại phát triển độc lập từ một tinh thể muối có niên đại cách đây 250 triệu năm,[149]tuy nhiên những tuyên bố này vẫn còn trong vòng tranh cãi.[150][151]
Những thành phần "vữa gạch" của DNA (adenine,guaninevà cả nhữngphân tử hữu cơliên quan) có thể đã hình thành từ vũ trụ trong nhữngkhoảng không liên thiên thể.[152][153][154]Nhữnghợp chất hữu cơcấu tạo nền tảng khác của DNA vàRNAtrongsự sống,bao gồmuracil,cytosinevàthymine,cũng đã được tổng hợp trong phòng thí nghiệm dưới các điều kiện mô phỏng tương ứng tìm thấy trongkhông gian ngoài thiên thể,bằng cách sử dụng những chất hóa học khởi đầu, ví dụpyrimidine,tìm thấy trong các mảnhvẫn thạch.Pyrimidine, như nhữnghydrocarbon đa vòng thơm(polycyclic aromatic hydrocarbons - PAHs), là hợp chất hóa học giàu carbon nhất tìm thấy trongvũ trụ,có thể được hình thành trong nhữngngôi sao khổng lồ đỏhay trong những đám mây khí vàbụigiữa các vì sao.[155]
Sử dụng trong công nghệ
[sửa|sửa mã nguồn]Kỹ thuật di truyền
[sửa|sửa mã nguồn]Nhiều phương pháp đã được phát triển để sàng lọc DNA từ sinh vật sống, nhưchiết lỏng-lỏng phenol-clorofom,và vận dụng trong phòng thí nghiệm, như phương phápphân hủy giới hạn(restriction digest) vàphản ứng chuỗi polymerase.Sinh họchiện đại và ngànhhóa sinhsử dụng thường xuyên những kỹ thuật này trong công nghệ tái tổ hợp DNA.Tái tổ hợp DNAlà một trình tự DNA nhân tạo được lắp ghép từ các trình tự DNA khác. Chúng có thể đượcbiến nạpvào tế bào sinh vật dưới dạngplasmidhoặc trong những dạng thích hợp khác, bằng cách sử dụngvector virut.[156]Những sinh vậtbiến đổi di truyềncó thể được ứng dụng để sinh ra các sản phẩm như protein tái tổ hợp, sử dụng trongnghiên cứu y học,[157]hoặc được nuôi trồng trongnông nghiệp.[158][159]
Kỹ thuật nhận diện DNA
[sửa|sửa mã nguồn]Các nhàkhoa học pháp ysử dụng DNA trongmáu,tinh dịch,da,nước bọthaytóctìm thấy tại hiện trường để nhận ra DNA khớp với của một cá nhân, như của thủ phạm chẳng hạn. Quá trình này được gọi làkỹ thuật nhận diện DNA(DNA profiling), hay còn gọi làkỹ thuật in dấu DNA(DNA fingerprintin)). Trong kỹ thuật nhận diện DNA, độ dài của nhiều đoạn DNA lặp lại, như các đoạn trình tựvi vệ tinh(microsatellite) vàvệ tinh nhỏ(minisatellite), được so sánh giữa các cá nhân có liên quan. Phương pháp này thường là một kỹ thuật cực kỳ tin cậy cho phép xác định những trình tự DNA ăn khớp với nhau.[160]Tuy vậy, việc nhận dạng có thể trở nên phức tạp nếu tại hiện trường gây án có nhiều DNA của nhiều người.[161]Kỹ thuật nhận diện DNA phát triển vào năm 1984 bởi nhà di truyền học người Anh SirAlec Jeffreys,[162]và lần đầu tiên được sử dụng trong ngành pháp y để cáo buộcColin Pitchforktrong vụ án Enderby năm 1988.[163]
Sự phát triển của khoa học pháp y, và khả năng hiện nay có thể nhận ra thông tin di truyền từ các mẫu máu, da, nước bọt hay tóc đã dẫn đến nhiều vụ án phải lật lại hồ sơ mặc dù tòa đã tuyên án. Chứng cứ mà hiện nay có thể được tiết lộ ra trong khi ở thời điểm thẩm vấn là bất khả thi về mặt khoa học. Kết hợp với đạo luật loại bỏ trường hợp bất trùng khả tố (double jeopardy-một người không bị xử hai lần về một tội) ở một số nơi, đã cho phép khởi tố lại một số vụ án khi bản án trước đó đã không nêu được chứng cứ thuyết phục để kết án. Những người mang tội danh nặng được phép yêu cầu lấy mẫu DNA nhằm mục đích so sánh. Trường hợp biện hộ rõ ràng nhất đó là mẫu DNA nhận được từ pháp y bị cho là đã bị ảnh hưởng từ những người ở xung quanh vụ án. Điều này làm cho các thủ tục điều tra trở nên chặt chẽ hơn trong những trường hợp phạm tội mới. Nhận diện DNA cũng được áp dụng thành công cho nhận dạng các nạn nhân trong những vụ tai nạn có thương vong lớn,[164]từ những phần cơ thể, và nhận biết từng nạn nhân trong những mồ chôn tập thể trong chiến tranh, thông qua so sánh với DNA của người nhà nạn nhân.
Kỹ thuật nhận diện DNA cũng được sử dụng đểxác thựcmối liên hệ sinh học với cha mẹ hoặc ông bà của một đứa trẻ với xác suất chính xác lên tới 99,99%. Những phương pháp kiểm trình tự DNA bình thường được thực hiện sau sinh, nhưng những phương pháp mới có thể kiểm tra quan hệ huyết thống ngay cả khi người mẹ đang mang thai.[165]
DNA enzyme hay xúc tác DNA
[sửa|sửa mã nguồn]Deoxyribozyme,cũng gọi làDNAzymehay xúc tác DNA phát hiện lần đầu tiên vào năm 1994.[166]Phần lớn chúng là những trình tự mạch đơn DNA được cô lập khỏi một vũng lớn gồm nhiều trình tự DNA ngẫu nhiên thông qua một hướng tiếp cận tổ hợp gọi là kỹ thuật lựa chọnin vitrohayphương pháp SELEX.Những DNAzyme tham gia xúc tác các phản ứng hóa học bao gồm phân cắt RNA-DNA, kết nối RNA-DNA, sựphosphoryl hóa- phản phosphoryl hóa cácamino acid,hình thành liên kết carbon-carbon, v.v... DNAzyme có thể tăng cường tốc độ phản ứng hóa học gấp 100.000.000.000 lần so với phản ứng không có sự tham gia xúc tác của nó.[167]Các DNAzyme được nghiên cứu nhiều nhất là những loại phân cắt RNA dùng để phát hiện các ion kim loại khác nhau và thiết kế các tác nhân trị liệu. Một vài DNAzyme đặc hiệu ion kim loại bao gồm GR-5 DNAzyme (đặc hiệu với chì),[166]CA1-3 DNAzymes (với đồng),[168]39E DNAzyme (với ion uranyl) và NaA43 DNAzyme (với natri).[169]NaA43 DNAzyme, nhạy với natri gấp 10.000 lần so với các ion kim loại khác, được dùng để theo dõi natri theo thời gian thực trong tế bào sống.
Tin sinh học
[sửa|sửa mã nguồn]
Tin sinh họcbao gồm các kỹ thuật lưu trữ,khai phá dữ liệu,tìm kiếm và thao tác với dữ liệu sinh học, bao gồm dữ liệu vềtrình tự acid nucleicDNA. Các kỹ thuật này mang đến những ứng dụng rộng rãi trongkhoa học máy tính,đặc biệt làthuật toán tìm kiếm chuỗi,học máyvàlý thuyết cơ sở dữ liệu.[170]Thuật toán tìm kiếm chuỗi hay so khớp, trong đó tìm kiếm sự xuất hiện của một trình tự các chữ cái trong một trình tự các chữ cái lớn hơn, được phát triển để tìm ra những trình tự nucleotide cụ thể.[171]Trình tự DNA có thểsắp gióngvới những trình tự DNA khác để nhận ra các trình tựtương đồngvà xác định vị tríđột biếnkhiến chúng khác biệt. Những kỹ thuật này, đặc biệt là kỹ thuật "sắp gióng đa trình tự"(multiple sequence alignment), được sử dụng để nghiên cứu các mối quan hệphát sinh chủng loài họcvà chức năng của protein.[172]Tập hợp dữ liệu của toàn bộ trình tự DNA, như được lập ra bởiDự án bản đồ gene người,là khó để sử dụng mà không có các chú giải cho phép nhận ra vị trí của các gene hay các yếu tố điều hòa ở mỗi nhiễm sắc thể. Vùng trình tự DNA với những phần đặc trưng gắn với gene mã hóa cho protein hoặc RNA có thể tìm ra bằng thuật toántìm kiếm gene(gene finding algorithm), cho phép các nhà nghiên cứu dự đoán sự có mặt của nhữngsinh phẩmđặc biệt mã hóa bởi gen và chức năng của chúng trong sinh vật trước khi chúng được phát hiện bằng thực nghiệm.[173]Toàn bộ hệ gene cũng có thể được đối sánh, để làm sáng tỏ lịch sử tiến hóa của từng sinh vật cụ thể và cho phép kiểm tra các sự kiện tiến hóa phức tạp.
Công nghệ nano DNA
[sửa|sửa mã nguồn]
Công nghệ nano DNA sử dụng nhữngtính chất tương táccủa phân tử DNA và những acid nucleic khác để tạo ra những phức hợp DNA phân nhánh tự lắp ráp có tính năng hữu ích.[174]Do vậy DNA được sử dụng như là vật liệu cấu trúc hơn là vật liệu mang thông tin sinh học. Các nhà khoa học đã tạo ra những dàn lưới hai chiều tuần hoàn (bằng phương pháp lát gạch vàorigami DNA) và cấu trúc ba chiềuđa diệnđều.[175]Thiết bị cơ nanovàthuật toán tự lắp rápcũng được chứng minh là khả dĩ,[176]và những cấu trúc DNA này dùng làm khuôn mẫu để sắp xếp các phân tử khác nhưkeo vàng(colloidal gold) và proteinstreptavidintrong vi khuẩnStreptomyces avidinii.[177]
Lịch sử và nhân chủng học
[sửa|sửa mã nguồn]Bởi vì theo thời gian DNA tích lũy các đột biến, do vậy chúng được di truyền lại, nên DNA chứa thông tin lịch sử, và bằng cách so sánh các trình tự DNA, những nhà di truyền học có thể suy luận ra lịch sử tiến hóa của mỗi loài sinh vật, hayphát sinh chủng loàicủa chúng.[178]Lĩnh vực phát sinh chủng loài học là một công cụ mạnh củasinh học tiến hóa.Nếu so sánh những trình tự DNA của một loài với nhau, các nhàdi truyền quần thểcó thể biết được lịch sử phát triển của một quần thể đang nghiên cứu. Kết quả nghiên cứu của ngành này được áp dụng sang chodi truyền sinh tháivànhân chủng học.Ví dụ, các nhà khoa học đã sử dụng bằng chứng DNA để nghiên cứu sự kiệnMười bộ tộc biến mất(Ten Lost Tribes) của Israel.[179][180]
Lưu trữ thông tin
[sửa|sửa mã nguồn]Trong một bài báo trên tạp chíNaturetháng 1 năm 2013, các nhà khoa học từHọc viện Tin sinh học Châu Âuvà công tyAgilent Technologiesđã đề xuất một cơ chế sử dụng khả năng mã hóa thông tin của DNA để phục vụ cho việc lưu trữ kỹ thuật số. Nhóm nghiên cứu mã hóa 739 kilobyte dữ liệu vào mã DNA, rồi tổng hợp nên DNA thực thụ, tiếp đó thực hiện giải trình tự DNA và giải mã thông tin ngược trở lại dạng ban đầu, mà họ thông báo là kết quả thu được với độ chính xác 100%. Thông tin được mã hóa chứa các tập tin định dạng văn bản và âm thanh. Một thí nghiệm khác thực hiện trước đó bởi nhóm nghiên cứu ởĐại học Harvardtháng 8 năm 2012, khi nhóm này mã hóa một quyển sách chứa 54.000 từ vào DNA.[181][182]
Trong tế bào sinh vật sống, thông tin lưu trữ ở DNA có thể được kích hoạt bởi các enzyme. Ví dụ như các kênh ion có proteincảm thụ ánh sángphối hợp với enzyme xử lý DNA là phù hợp cho nhiệm vụ trên trong ống nghiệm (in vitro).[183][184]Những phân tử exonuclease huỳnh quang có thể truyền tín hiệu ra bên ngoài tuân theo các trình tự nucleotide mà chúng đọc được.[185]
Lịch sử nghiên cứu DNA
[sửa|sửa mã nguồn]
DNA lần đầu tiên được cô lập bởi thầy thuốc ngườiThụy SĩFriedrich Miescher,người mà vào năm 1869, đã khám phá ra một chất vi mô trong mủ của băng gạc được tháo bỏ sau phẫu thuật. Vì nó nằm trong nhân của tế bào, ông đã gọi nó là "nuclein".[186][187]Năm 1878,Albrecht Kosselđã cô lập được thành phần không phải là protein của "nuclein", acid nucleic, và sau đó ông cô lập được nămnucleobasecơ bản của nó.[188][189]Năm 1919, Phoebus Levene nhận biết được các đơn vị của nucleotide là base, đường và phosphat.[190]Levene đề xuất rằng DNA chứa một chuỗi các đơn vị nucleotide được liên kết với nhau bằng các nhóm phosphat. Levene đã nghĩ rằng mạch này là ngắn và các base lặp lại theo một thứ tự cố định. Năm 1937,William Astburychụp được ảnh thành phần nhiễu xạ tia X đầu tiên cho thấy DNA có một cấu trúc đều đặn.[191]
Năm 1927,Nikolai Koltsovđề xuất rằng các tính trạng di truyền có thể được thừa hưởng thông qua một "phân tử di truyền khổng lồ" cấu thành từ "hai mạch đối xứng mà có thể sao chép theo cách bán bảo tồn sử dụng từng mạch như là một khuôn mẫu".[192][193]Năm 1928,Frederick Griffithtrongthí nghiệmcủa ông đã khám phá ra cáctính trạngdạng "trơn" củaphế cầu khuẩn(Pneumococcus) có thể truyền sang dạng "thô" của cùng một loài vi khuẩn bằng cách trộn các vi khuẩn dạng "trơn" đã bị giết với các vi khuẩn dạng "thô" còn sống bằng thí nghiệm nổi tiếng gọi làthí nghiệm Griffith.[194][195]Hệ thống thí nghiệm này cung cấp gợi ý rõ ràng đầu tiên về DNA mang thông tin di truyền—theothí nghiệm Avery–MacLeod–McCarty—khiOswald Avery,cùng với các đồng nghiệpColin MacLeodvàMaclyn McCarty,nhận ra DNA tuân theonguyên lý biến nạptrong thí nghiệm Griffith vào năm 1943.[196]Vai trò của DNA trongdi truyềnđược xác nhận vào năm 1952, khiAlfred HersheyvàMartha Chasetrongthí nghiệm Hershey–Chasechỉ ra rằng DNA làvật liệu di truyềncủathực khuẩn thể T2.[197]
Năm 1953,James WatsonvàFrancis Cricklần đầu tiênđề xuất mô hìnhmà được chấp nhận ngày nay với cấu trúc DNA chuỗi xoắn kép đăng trên tạp chíNature.[13]Mô hình phân tử chuỗi xoắn kép DNA của họ khi ấy dựa trên ảnh chụpnhiễu xạ tia X(còn gọi là "Ảnh chụp 51")[198]doRosalind FranklinvàRaymond Goslingthực hiện vào tháng 5 năm 1952, và dựa trên thông tin rằng các base DNA ghép cặp với nhau.
Chứng cứ thực nghiệm ủng hộ mô hình Watson và Crick được công bố trong một loạt 5 bài báo đăng trên cùng một số của tờNature.[199]Trong các bài báo này, bài viết của Franklin và Gosling là công trình đầu tiên của chính họ công bố dữ liệu về nhiễu xạ tia X và phương pháp phân tích gốc giúp ủng hộ một phần mô hình của Watson và Crick;[51][200]trong số báo này cũng bao gồm bài viết về cấu trúc DNA củaMaurice Wilkinsvới hai đồng nghiệp của ông, khi họ thực hiện phân tích ảnh chụp tia X của dạng B-DNAtrong cơ thể sống(in vivo) mà cũng ủng hộ cho sự có mặttrong cơ thể sốngcủa cấu trúc chuỗi xoắn kép DNA như đề xuất của Crick và Watson về mô hình phân tử DNA của họ trong bài báo dài 2 trang đăng ở số trước của tạp chíNature.[52]Năm 1962, khi ấy Franklin đã qua đời, Watson, Crick, vàWilkinscùng nhậnGiải Nobel Sinh lý và Y học.[201]Do điều lệ củaQuỹ Nobelchỉ trao giải cho những nhà khoa học còn sống. Vẫn có những tranh luận về sau liên quan đến những ai xứng đáng được công nhận liên quan đến khám phá này.[202]
Trong một buổi nói chuyện có tầm ảnh hưởng vào năm 1957, Crick đưa raluận thuyết trung tâm của sinh học phân tử,báo hiệu trước về mối quan hệ giữa các phân tử DNA,RNA,vàprotein,và khớp nối với "giả thuyết về dòng thông tin".[203]Chứng cứ thực nghiệm cuối cùng xác nhận cơ chế sao chép mà hàm ý cấu trúc chuỗi xoắn kép được công bố vào năm 1958 thông qua thí nghiệm Meselson–Stahl.[204]Những công trình về sau của Crick và các đồng nghiệp cũng như của nhiều nhà khoa học khác chứng tỏ mã di truyền có cơ sở là tổ hợp của bộ ba base không chồng lợp nhau, hay còn gọi là các codon, cho phépHar Gobind Khorana,Robert W. HolleyvàMarshall Warren Nirenberglàm sáng tỏ mã di truyền.[205]Những phát hiện này đã khai sinh ra ngànhsinh học phân tử.
Xem thêm
[sửa|sửa mã nguồn]- Nhiễm sắc thể thường
- Tinh thể học
- Thư viện hóa học DNA mã hóa
- Giải trình tự DNA
- Đại phân tử
- Rối loạn di truyền
- Haplotype
- DNA microarray
- Bệnh di truyền
- Bản so sánh những phần mềm mô phỏng acid nucleic
- Giảm phân
- Chuỗi xoắn kép acid nucleic
- Ký hiệu acid nucleic
- Trình tự acid nucleic
- Thuyết pangen
- Phosphoramidit
- Thẩm tách Southern
- Kỹ thuật tán xạ tia X
- Acid nucleic xeno
- RNA
- Deoxyribozyme
Chú thích
[sửa|sửa mã nguồn]- ^“deoxyribonucleic acid”.Merriam-Webster Dictionary.
- ^Harvey Lodish, Arnold Berk, Chris A. Kaiser, Monty Krieger, Anthony Bretscher (Bản dịch: Nhiều tác giả) (2012). “4”.Molecular Cell Biology (Sinh học phân tử của tế bào).Tập 2. Di truyền học và sinh học phân tử (ấn bản 7). Hoa Kỳ (Bản dịch: Việt Nam): W. H. Freeman (Bản dịch: Nhà xuất bản Trẻ). tr. 2.ISBN9781429234139.Bản gốclưu trữ ngày 7 tháng 4 năm 2017.Truy cập ngày 7 tháng 4 năm 2017.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- ^Neil A. Campbell, Jane B. Reece, Lisa A. Urry, Michael L. Cain, Steven A. Wasserman, Peter V. Minorsky, Robert B. Jackson (Bản dịch: Nhiều tác giả) (2008). “5”.Biology 8th Edition (Sinh học)(ấn bản 8). Hoa Kỳ (Bản dịch: Việt Nam): Pearson Benjamin Cummings (Bản dịch: Nhà xuất bản Giáo dục). tr. 86.ISBN978-0805368444.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- ^Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2014).Molecular Biology of the Cell(ấn bản 6). Garland. tr. Chapter 4: DNA, Chromosomes and Genomes.ISBN9780815344322.Bản gốclưu trữ ngày 14 tháng 7 năm 2014.Truy cập ngày 8 tháng 11 năm 2016.
- ^Purcell, Adam.“DNA”.Basic Biology.Lưu trữbản gốc ngày 5 tháng 1 năm 2017.Truy cập ngày 8 tháng 12 năm 2016.
- ^Nuwer, Rachel (ngày 18 tháng 7 năm 2015).“Counting All the DNA on Earth”.The New York Times.New York: The New York Times Company.ISSN0362-4331.Bản gốclưu trữ ngày 26 tháng 7 năm 2016.Truy cập ngày 18 tháng 7 năm 2015.
- ^“The Biosphere: Diversity of Life”.Aspen Global Change Institute.Basalt, CO.Lưu trữbản gốc ngày 20 tháng 12 năm 2016.Truy cập ngày 19 tháng 7 năm 2015.
- ^Russell, Peter (2001).iGenetics.New York: Benjamin Cummings.ISBN0-8053-4553-1.
- ^Mashaghi A, Katan A (2013). “A physicist's view of DNA”.De Physicus.24e(3): 59–61.arXiv:1311.2545v1.Bibcode:2013arXiv1311.2545M.
- ^Saenger, Wolfram (1984).Principles of Nucleic Acid Structure.New York: Springer-Verlag.ISBN0-387-90762-9.
- ^abAlberts B, Johnson A, Lewis J, Raff M, Roberts K, Peter W (2002).Molecular Biology of the Cell.New York and London: Garland Science.ISBN0-8153-3218-1.OCLC145080076.Lưu trữbản gốc ngày 1 tháng 11 năm 2016.
- ^Irobalieva, Rossitza N.; Fogg, Jonathan M.; Catanese Jr, Daniel J.; Sutthibutpong, Thana; Chen, Muyuan; Barker, Anna K.; Ludtke, Steven J.; Harris, Sarah A.; Schmid, Michael F. (ngày 12 tháng 10 năm 2015).“Structural diversity of supercoiled DNA”.Nature Communications.6:8440.doi:10.1038/ncomms9440.PMC4608029.PMID26455586.Bản gốclưu trữ ngày 20 tháng 12 năm 2016.Truy cập ngày 8 tháng 12 năm 2016.
- ^abcdWatson JD, Crick FH (1953).“A Structure for Deoxyribose Nucleic Acid”(PDF).Nature.171(4356): 737–738.Bibcode:1953Natur.171..737W.doi:10.1038/171737a0.PMID13054692.Bản gốc(PDF)lưu trữ ngày 24 tháng 10 năm 2017.Truy cập ngày 3 tháng 11 năm 2014.
- ^Mandelkern M, Elias JG, Eden D, Crothers DM (1981). “The dimensions of DNA in solution”.J Mol Biol.152(1): 153–61.doi:10.1016/0022-2836(81)90099-1.PMID7338906.
- ^Gregory SG, Barlow KF, McLay KE, Kaul R, Swarbreck D, Dunham A, và đồng nghiệp (2006). “The DNA sequence and biological annotation of human chromosome 1”.Nature.441(7091): 315–21.Bibcode:2006Natur.441..315G.doi:10.1038/nature04727.PMID16710414.
- ^abcBerg J., Tymoczko J. and Stryer L. (2002)Biochemistry.W. H. Freeman and CompanyISBN0-7167-4955-6
- ^Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their ConstituentsLưu trữ2007-02-05 tạiWayback MachineIUPAC-IUB Commission on Biochemical Nomenclature (CBN). Truy cập 17 tháng 7 năm 2016.
- ^abGhosh A, Bansal M (2003). “A glossary of DNA structures from A to Z”.Acta Crystallogr D.59(4): 620–6.doi:10.1107/S0907444903003251.PMID12657780.
- ^Lấy từPDB 1D65Lưu trữ2016-11-30 tạiWayback Machinedoi:10.2210/pdb1d65/pdb
- ^Yakovchuk P, Protozanova E, Frank-Kamenetskii MD (2006).“Base-stacking and base-pairing contributions into thermal stability of the DNA double helix”.Nucleic Acids Res.34(2): 564–74.doi:10.1093/nar/gkj454.PMC1360284.PMID16449200.
- ^Burton E. Tropp -"Molecular Biology"- Jones and Barlett Learning,ISBN 978-0-7637-8663-2
- ^“Watson-Crick Structure of DNA - 1953”.Steven Carr.Memorial University of Newfoundland.Lưu trữbản gốc ngày 19 tháng 7 năm 2016.Truy cập ngày 13 tháng 7 năm 2016.
- ^Verma S, Eckstein F (1998).“Modified oligonucleotides: synthesis and strategy for users”.Annu. Rev. Biochem.67:99–134.doi:10.1146/annurev.biochem.67.1.99.PMID9759484.
- ^Kiljunen S, Hakala K, Pinta E, Huttunen S, Pluta P, Gador A, Lönnberg H, Skurnik M (2005). “Yersiniophage phiR1-37 is a tailed bacteriophage having a 270 kb DNA genome with thymidine replaced by deoxyuridine”.Microbiology.151(12): 4093–4102.doi:10.1099/mic.0.28265-0.PMID16339954.
- ^Uchiyama J, Takemura-Uchiyama I, Sakaguchi Y, Gamoh K, Kato SI, Daibata M, Ujihara T, Misawa N, Matsuzaki S (tháng 3 năm 2014). “Intragenus generalized transduction inStaphylococcusspp. by a novel giant phage”.ISME J.8:1949–1952.doi:10.1038/ismej.2014.29.
- ^Simpson L (1998).“A base called J”.Proc Natl Acad Sci USA.95(5): 2037–2038.Bibcode:1998PNAS...95.2037S.doi:10.1073/pnas.95.5.2037.PMC33841.PMID9482833.
- ^Borst P, Sabatini R (2008).“Base J: discovery, biosynthesis, and possible functions”.Annual Review of Microbiology.62:235–51.doi:10.1146/annurev.micro.62.081307.162750.PMID18729733.
- ^Cross M, Kieft R, Sabatini R, Wilm M, de Kort M, van der Marel GA, van Boom JH, van Leeuwen F, Borst P (1999).“The modified base J is the target for a novel DNA-binding protein in kinetoplastid protozoans”.The EMBO Journal.18(22): 6573–6581.doi:10.1093/emboj/18.22.6573.PMC1171720.PMID10562569.
- ^DiPaolo C, Kieft R, Cross M, Sabatini R (2005). “Regulation of trypanosome DNA glycosylation by a SWI2/SNF2-like protein”.Mol Cell.17(3): 441–451.doi:10.1016/j.molcel.2004.12.022.PMID15694344.
- ^Vainio S, Genest PA, ter Riet B, van Luenen H, Borst P (2009). “Evidence that J-binding protein 2 is a thymidine hydroxylase catalyzing the first step in the biosynthesis of DNA base J”.Molecular and biochemical parasitology.164(2): 157–61.doi:10.1016/j.molbiopara.2008.12.001.PMID19114062.
- ^Iyer LM, Tahiliani M, Rao A, Aravind L (2009).“Prediction of novel families of enzymes involved in oxidative and other complex modifications of bases in nucleic acids”.Cell Cycle.8(11): 1698–1710.doi:10.4161/cc.8.11.8580.PMC2995806.PMID19411852.
- ^van Luenen HG, Farris C, Jan S, Genest PA, Tripathi P, Velds A, Kerkhoven RM, Nieuwland M, Haydock A, Ramasamy G, Vainio S, Heidebrecht T, Perrakis A, Pagie L, van Steensel B, Myler PJ, Borst P (2012).“Leishmania”.Cell.150(5): 909–921.doi:10.1016/j.cell.2012.07.030.PMC3684241.PMID22939620.
- ^Hazelbaker DZ, Buratowski S (2012).“Transcription: base J blocks the way”.Curr Biol.22(22): R960–2.doi:10.1016/j.cub.2012.10.010.PMC3648658.PMID23174300.
- ^Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson RE (1980). “Crystal structure analysis of a complete turn of B-DNA”.Nature.287(5784): 755–8.Bibcode:1980Natur.287..755W.doi:10.1038/287755a0.PMID7432492.
- ^abPabo CO, Sauer RT (1984).“Protein-DNA recognition”.Annu Rev Biochem.53:293–321.doi:10.1146/annurev.bi.53.070184.001453.PMID6236744.
- ^Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub HE (2000).“Mechanical stability of single DNA molecules”.Biophys J.78(4): 1997–2007.Bibcode:2000BpJ....78.1997C.doi:10.1016/S0006-3495(00)76747-6.PMC1300792.PMID10733978.
- ^Chalikian TV, Völker J, Plum GE, Breslauer KJ (1999).“A more unified picture for the thermodynamics of nucleic acid duplex melting: A characterization by calorimetric and volumetric techniques”.Proc Natl Acad Sci USA.96(14): 7853–8.Bibcode:1999PNAS...96.7853C.doi:10.1073/pnas.96.14.7853.PMC22151.PMID10393911.
- ^deHaseth PL, Helmann JD (1995).“Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA”.Mol Microbiol.16(5): 817–24.doi:10.1111/j.1365-2958.1995.tb02309.x.PMID7476180.
- ^Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J (2004). “Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern”.Biochemistry.43(51): 15996–6010.doi:10.1021/bi048221v.PMID15609994.
- ^Designation of the two strands of DNALưu trữ2008-04-24 tạiWayback MachineJCBN/NC-IUB Newsletter 1989. Truy cập ngày 7 tháng 5 năm 2008
- ^Hüttenhofer A, Schattner P, Polacek N (2005).“Non-coding RNAs: hope or hype?”.Trends Genet.21(5): 289–97.doi:10.1016/j.tig.2005.03.007.PMID15851066.
- ^Munroe SH (2004). “Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns”.J Cell Biochem.93(4): 664–71.doi:10.1002/jcb.20252.PMID15389973.
- ^Makalowska I, Lin CF, Makalowski W (2005).“Overlapping genes in vertebrate genomes”.Comput Biol Chem.29(1): 1–12.doi:10.1016/j.compbiolchem.2004.12.006.PMID15680581.
- ^Johnson ZI, Chisholm SW (2004).“Properties of overlapping genes are conserved across microbial genomes”.Genome Res.14(11): 2268–72.doi:10.1101/gr.2433104.PMC525685.PMID15520290.
- ^Lamb RA, Horvath CM (1991). “Diversity of coding strategies in influenza viruses”.Trends Genet.7(8): 261–6.doi:10.1016/0168-9525(91)90326-L.PMID1771674.
- ^Benham CJ, Mielke SP (2005). “DNA mechanics”.Annu Rev Biomed Eng.7:21–53.doi:10.1146/annurev.bioeng.6.062403.132016.PMID16004565.
- ^abChampoux JJ (2001).“DNA topoisomerases: structure, function, and mechanism”.Annual Review of Biochemistry.70:369–413.doi:10.1146/annurev.biochem.70.1.369.PMID11395412.
- ^abWang JC (tháng 6 năm 2002). “Cellular roles of DNA topoisomerases: a molecular perspective”.Nature Reviews. Molecular Cell Biology.3(6): 430–40.doi:10.1038/nrm831.PMID12042765.
- ^Basu HS, Feuerstein BG, Zarling DA, Shafer RH, Marton LJ (1988). “Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies”.J Biomol Struct Dyn.6(2): 299–309.doi:10.1080/07391102.1988.10507714.PMID2482766.
- ^Franklin RE, Gosling RG (ngày 6 tháng 3 năm 1953).“The Structure of Sodium Thymonucleate Fibres I. The Influence of Water Content”(PDF).Acta Crystallogr.6(8–9): 673–7.doi:10.1107/S0365110X53001939.Bản gốc(PDF)lưu trữ ngày 9 tháng 1 năm 2016.Truy cập ngày 8 tháng 11 năm 2016.
Franklin RE, Gosling RG (1953). “The structure of sodium thymonucleate fibres. II. The cylindrically symmetrical Patterson function”.Acta Crystallogr.6(8–9): 678–85.doi:10.1107/S0365110X53001940. - ^abFranklin RE, Gosling RG (1953).“Molecular Configuration in Sodium Thymonucleate. Franklin R. and Gosling R.G”(PDF).Nature.171(4356): 740–1.Bibcode:1953Natur.171..740F.doi:10.1038/171740a0.PMID13054694.Bản gốc(PDF)lưu trữ ngày 3 tháng 1 năm 2011.Truy cập ngày 8 tháng 11 năm 2016.
- ^abWilkins MH, Stokes AR, Wilson HR (1953).“Molecular Structure of Deoxypentose Nucleic Acids”(PDF).Nature.171(4356): 738–740.Bibcode:1953Natur.171..738W.doi:10.1038/171738a0.PMID13054693.Bản gốc(PDF)lưu trữ ngày 13 tháng 5 năm 2011.Truy cập ngày 8 tháng 11 năm 2016.
- ^Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (1980). “Polymorphism of DNA double helices”.J. Mol. Biol.143(1): 49–72.doi:10.1016/0022-2836(80)90124-2.PMID7441761.
- ^Baianu, I.C. (1980). “Structural Order and Partial Disorder in Biological systems”.Bull. Math. Biol.42(4): 137–141.doi:10.1016/s0092-8240(80)80083-8.http://cogprints.org/3822/Lưu trữ2009-07-25 tạiWayback Machine
- ^Hosemann R., Bagchi R.N.,Direct analysis of diffraction by matter,North-Holland Publs., Amsterdam – New York, 1962.
- ^Baianu, I.C. (1978). “X-ray scattering by partially disordered membrane systems”.Acta Crystallogr. A.34(5): 751–3.Bibcode:1978AcCrA..34..751B.doi:10.1107/S0567739478001540.
- ^Wahl MC, Sundaralingam M (1997).“Crystal structures of A-DNA duplexes”.Biopolymers.44(1): 45–63.doi:10.1002/(SICI)1097-0282(1997)44:1<45::AID-BIP4>3.0.CO;2-#.PMID9097733.
- ^Lu XJ, Shakked Z, Olson WK (2000). “A-form conformational motifs in ligand-bound DNA structures”.J. Mol. Biol.300(4): 819–40.doi:10.1006/jmbi.2000.3690.PMID10891271.
- ^Rothenburg S, Koch-Nolte F, Haag F (2001). “DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression ofalleles”.Immunol Rev.184:286–98.doi:10.1034/j.1600-065x.2001.1840125.x.PMID12086319.
- ^Oh DB, Kim YG, Rich A (2002).“Z-DNA-binding proteins can act as potent effectors of gene expression in vivo”.Proc. Natl. Acad. Sci. U.S.A.99(26): 16666–71.Bibcode:2002PNAS...9916666O.doi:10.1073/pnas.262672699.PMC139201.PMID12486233.
- ^Rich A, Norheim A, Wang AH (1984).“The chemistry and biology of left-handed Z-DNA”.Annual Review of Biochemistry.53:791–846.doi:10.1146/annurev.bi.53.070184.004043.PMID6383204.
- ^Sinden, Richard R (ngày 15 tháng 1 năm 1994).DNA structure and function(ấn bản 1). Academic Press. tr.398.ISBN0-12-645750-6.
- ^Ho PS (ngày 27 tháng 9 năm 1994).“The non-B-DNA structure of d(CA/TG)n does not differ from that of Z-DNA”.Proc Natl Acad Sci USA.91(20): 9549–9553.Bibcode:1994PNAS...91.9549H.doi:10.1073/pnas.91.20.9549.PMC44850.PMID7937803.
- ^abPalmer, Jason (ngày 2 tháng 12 năm 2010).“Arsenic-loving bacteria may help in hunt for alien life”.BBC News.Bản gốclưu trữ ngày 3 tháng 12 năm 2010.Truy cập ngày 2 tháng 12 năm 2010.
- ^abBortman, Henry (ngày 2 tháng 12 năm 2010).“Arsenic-Eating Bacteria Opens New Possibilities for Alien Life”.Space.com.Bản gốclưu trữ ngày 17 tháng 11 năm 2016.Truy cập ngày 2 tháng 12 năm 2010.
- ^Katsnelson, Alla (ngày 2 tháng 12 năm 2010).“Arsenic-eating microbe may redefine chemistry of life”.Nature News.doi:10.1038/news.2010.645.Bản gốclưu trữ ngày 24 tháng 2 năm 2012.Truy cập ngày 8 tháng 11 năm 2016.
- ^Cressey, Daniel (ngày 3 tháng 10 năm 2012). “'Arsenic-life' Bacterium Prefers Phosphorus after all”.Nature News.doi:10.1038/nature.2012.11520.
- ^abGreider CW, Blackburn EH (1985). “Identification of a specific telomere terminal transferase activity in Tetrahymena extracts”.Cell.43(2 Pt 1): 405–13.doi:10.1016/0092-8674(85)90170-9.PMID3907856.
- ^abcNugent CI, Lundblad V (1998). “The telomerase reverse transcriptase: components and regulation”.Genes Dev.12(8): 1073–85.doi:10.1101/gad.12.8.1073.PMID9553037.
- ^Wright WE, Tesmer VM, Huffman KE, Levene SD, Shay JW (1997).“Normal human chromosomes have long G-rich telomeric overhangs at one end”.Genes Dev.11(21): 2801–9.doi:10.1101/gad.11.21.2801.PMC316649.PMID9353250.
- ^Created fromNDB UD0017
- ^abBurge S, Parkinson GN, Hazel P, Todd AK, Neidle S (2006).“Quadruplex DNA: sequence, topology and structure”.Nucleic Acids Res.34(19): 5402–15.doi:10.1093/nar/gkl655.PMC1636468.PMID17012276.
- ^Parkinson GN, Lee MP, Neidle S (2002). “Crystal structure of parallel quadruplexes from human telomeric DNA”.Nature.417(6891): 876–80.Bibcode:2002Natur.417..876P.doi:10.1038/nature755.PMID12050675.
- ^Griffith JD, Comeau L, Rosenfield S, Stansel RM, Bianchi A, Moss H, de Lange T (1999). “Mammalian telomeres end in a large duplex loop”.Cell.97(4): 503–14.doi:10.1016/S0092-8674(00)80760-6.PMID10338214.
- ^Seeman NC (2005).“DNA enables nanoscale control of the structure of matter”.Q. Rev. Biophys.38(4): 363–71.doi:10.1017/S0033583505004087.PMC3478329.PMID16515737.
- ^Hu Q, Rosenfeld MG (2012).“Epigenetic regulation of human embryonic stem cells”.Frontiers in Genetics.3:238.doi:10.3389/fgene.2012.00238.PMC3488762.PMID23133442.
- ^Klose RJ, Bird AP (2006). “Genomic DNA methylation: the mark and its mediators”.Trends Biochem Sci.31(2): 89–97.doi:10.1016/j.tibs.2005.12.008.PMID16403636.
- ^Bird A (2002). “DNA methylation patterns and epigenetic memory”.Genes Dev.16(1): 6–21.doi:10.1101/gad.947102.PMID11782440.
- ^Walsh CP, Xu GL (2006). “Cytosine methylation and DNA repair”.Curr Top Microbiol Immunol.Current Topics in Microbiology and Immunology.301:283–315.doi:10.1007/3-540-31390-7_11.ISBN3-540-29114-8.PMID16570853.
- ^Kriaucionis S, Heintz N (2009).“The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain”.Science.324(5929): 929–30.Bibcode:2009Sci...324..929K.doi:10.1126/science.1169786.PMC3263819.PMID19372393.
- ^Ratel D, Ravanat JL, Berger F, Wion D (2006).“N6-methyladenine: the other methylated base of DNA”.BioEssays.28(3): 309–15.doi:10.1002/bies.20342.PMC2754416.PMID16479578.
- ^Gommers-Ampt JH, Van Leeuwen F, de Beer AL, Vliegenthart JF, Dizdaroglu M, Kowalak JA, Crain PF, Borst P (1993). “beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei”.Cell.75(6): 1129–36.doi:10.1016/0092-8674(93)90322-H.PMID8261512.
- ^Tạo ra từPDB 1JDGLưu trữ2015-12-31 tạiWayback Machinedoi:10.2210/pdb1jdg/pdb
- ^Douki T, Reynaud-Angelin A, Cadet J, Sage E (2003). “Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation”.Biochemistry.42(30): 9221–6.doi:10.1021/bi034593c.PMID12885257.
- ^Cadet J, Delatour T, Douki T, Gasparutto D, Pouget JP, Ravanat JL, Sauvaigo S (1999). “Hydroxyl radicals and DNA base damage”.Mutat Res.424(1–2): 9–21.doi:10.1016/S0027-5107(99)00004-4.PMID10064846.
- ^Beckman KB, Ames BN (1997). “Oxidative decay of DNA”.J. Biol. Chem.272(32): 19633–6.doi:10.1074/jbc.272.32.19633.PMID9289489.
- ^Valerie K, Povirk LF (2003). “Regulation and mechanisms of mammalian double-strand break repair”.Oncogene.22(37): 5792–812.doi:10.1038/sj.onc.1206679.PMID12947387.
- ^Johnson, George (ngày 28 tháng 12 năm 2010).“Unearthing Prehistoric Tumors, and Debate”.The New York Times.Bản gốclưu trữ ngày 22 tháng 6 năm 2016.Truy cập ngày 8 tháng 11 năm 2016.
If we lived long enough, sooner or later we all would get cancer.
- ^Alberts, B; Johnson A, Lewis J; và đồng nghiệp (2002). “The Preventable Causes of Cancer”.Molecular biology of the cell(ấn bản 4). New York: Garland Science.ISBN0-8153-4072-9.Bản gốclưu trữ ngày 2 tháng 1 năm 2016.Truy cập ngày 11 tháng 12 năm 2016.
A certain irreducible background incidence of cancer is to be expected regardless of circumstances: mutations can never be absolutely avoided, because they are an inescapable consequence of fundamental limitations on the accuracy of DNA replication, as discussed in Chapter 5. If a human could live long enough, it is inevitable that at least one of his or her cells would eventually accumulate a set of mutations sufficient for cancer to develop.
- ^Bernstein H, Payne CM, Bernstein C, Garewal H, Dvorak K (2008). Cancer and aging as consequences of un-repaired DNA damage. In: New Research on DNA Damages (Editors: Honoka Kimura and Aoi Suzuki) Nova Science Publishers, Inc., New York, Chapter 1, pp. 1–47. open access, but read only Cancer and Aging as Consequences of Un-Repaired DNA Damage (pp. 1-47)ISBN 978-1604565812.Cancer and Aging as Consequences of Un-Repaired DNA Damage (pp. 1-47)Lưu trữ2014-10-25 tạiWayback Machine
- ^Hoeijmakers JH (tháng 10 năm 2009). “DNA damage, aging, and cancer”.N. Engl. J. Med.361(15): 1475–85.doi:10.1056/NEJMra0804615.PMID19812404.
- ^Freitas AA, de Magalhães JP (2011). “A review and appraisal of the DNA damage theory of ageing”.Mutat. Res.728(1–2): 12–22.doi:10.1016/j.mrrev.2011.05.001.PMID21600302.
- ^Ferguson LR, Denny WA (1991). “The genetic toxicology of acridines”.Mutat Res.258(2): 123–60.doi:10.1016/0165-1110(91)90006-H.PMID1881402.
- ^Stephens TD, Bunde CJ, Fillmore BJ (2000). “Mechanism of action in thalidomide teratogenesis”.Biochem Pharmacol.59(12): 1489–99.doi:10.1016/S0006-2952(99)00388-3.PMID10799645.
- ^Jeffrey AM (1985).“DNA modification by chemical carcinogens”.Pharmacol Ther.28(2): 237–72.doi:10.1016/0163-7258(85)90013-0.PMID3936066.
- ^Braña MF, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (2001). “Intercalators as anticancer drugs”.Curr Pharm Des.7(17): 1745–80.doi:10.2174/1381612013397113.PMID11562309.
- ^Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, và đồng nghiệp (2001). “The sequence of the human genome”.Science.291(5507): 1304–51.Bibcode:2001Sci...291.1304V.doi:10.1126/science.1058040.PMID11181995.
- ^Thanbichler M, Wang SC, Shapiro L (2005). “The bacterial nucleoid: a highly organized and dynamic structure”.J Cell Biochem.96(3): 506–21.doi:10.1002/jcb.20519.PMID15988757.
- ^Wolfsberg TG, McEntyre J, Schuler GD (2001). “Guide to the draft human genome”.Nature.409(6822): 824–6.Bibcode:2001Natur.409..824W.doi:10.1038/35057000.PMID11236998.
- ^Gregory TR (2005). “The C-value enigma in plants and animals: a review of parallels and an appeal for partnership”.Annals of Botany.95(1): 133–46.doi:10.1093/aob/mci009.PMID15596463.
- ^Birney E, Stamatoyannopoulos JA, Dutta A, Guigó R, Gingeras TR, Margulies EH, và đồng nghiệp (2007).“Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project”.Nature.447(7146): 799–816.Bibcode:2007Natur.447..799B.doi:10.1038/nature05874.PMC2212820.PMID17571346.
- ^Hình tạo từPDB 1MSWLưu trữ2008-01-06 tạiWayback Machine
- ^Pidoux AL, Allshire RC (2005).“The role of heterochromatin in centromere function”.Philosophical Transactions of the Royal Society B.360(1455): 569–79.doi:10.1098/rstb.2004.1611.PMC1569473.PMID15905142.
- ^Harrison PM, Hegyi H, Balasubramanian S, Luscombe NM, Bertone P, Echols N, Johnson T, Gerstein M (2002).“Molecular Fossils in the Human Genome: Identification and Analysis of the Pseudogenes in Chromosomes 21 and 22”.Genome Res.12(2): 272–80.doi:10.1101/gr.207102.PMC155275.PMID11827946.
- ^Harrison PM, Gerstein M (2002). “Studying genomes through the aeons: protein families, pseudogenes and proteome evolution”.J Mol Biol.318(5): 1155–74.doi:10.1016/S0022-2836(02)00109-2.PMID12083509.
- ^Albà M (2001).“Replicative DNA polymerases”.Genome Biol.2(1): reviews3002.1–reviews3002.4.doi:10.1186/gb-2001-2-1-reviews3002.PMC150442.PMID11178285.
- ^“Replication fork”.Andrew Staroscik.scienceprimer.com.Lưu trữbản gốc ngày 12 tháng 11 năm 2016.Truy cập 6 tháng 11 năm 2016.
- ^Tani, Katsuji; Nasu, Masao (2010). “Roles of Extracellular DNA in Bacterial Ecosystems”. Trong Kikuchi, Yo; Rykova, Elena Y. (biên tập).Extracellular Nucleic Acids.Springer. tr.25–38.ISBN978-3-642-12616-1.
- ^Vlassov, V. V.; Laktionov, P. P.; Rykova, E. Y. (2007). “Extracellular nucleic acids”.BioEssays.29:654–667.doi:10.1002/bies.20604.
- ^Finkel, S. E.; Kolter, R. (2001).“DNA as a nutrient: novel role for bacterial competence gene homologs”.J. Bacteriol.183:6288–6293.doi:10.1128/JB.183.21.6288-6293.2001.Bản gốclưu trữ ngày 23 tháng 6 năm 2017.Truy cập ngày 8 tháng 11 năm 2016.
- ^Mulcahy, H.; Charron-Mazenod, L.; Lewenza, S. (2008).“Extracellular DNA chelates cations and induces antibiotic resistance inPseudomonas aeruginosabiofilms”.PLoS Pathog.4:e1000213.doi:10.1371/journal.ppat.1000213.Bản gốclưu trữ ngày 13 tháng 8 năm 2020.Truy cập ngày 4 tháng 7 năm 2021.
- ^Berne, C.; Kysela, D. T.; Brun, Y. V. (2010).“A bacterial extracellular DNA inhibits settling of motile progeny cells within a biofilm”.Mol. Microbiol.77:815–829.doi:10.1111/j.1365-2958.2010.07267.x.Bản gốclưu trữ ngày 8 tháng 3 năm 2016.Truy cập ngày 8 tháng 11 năm 2016.
- ^Whitchurch, C. B.; Tolker-Nielsen, T.; Ragas, P. C.; Mattick, J. S. (2002).“Extracellular DNA required for bacterial biofilm formation”.Science.295:1487.doi:10.1126/science.295.5559.1487.Bản gốclưu trữ ngày 30 tháng 11 năm 2016.Truy cập ngày 29 tháng 11 năm 2016.
- ^Hu, W.; Li, L.; Sharma, S.; Wang, J.; McHardy, I.; Lux, R.; Yang, Z.; He, X.; Gimzewski, J. K.; Li, Y.; Shi, W. (2012). “DNA Builds and Strengthens the Extracellular Matrix in Myxococcus xanthus Biofilms by Interacting with Exopolysaccharides”.PLoS ONE.7(12): e51905.Bibcode:2012PLoSO...751905H.doi:10.1371/journal.pone.0051905.
- ^Sandman K, Pereira SL, Reeve JN (1998).“Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome”.Cell Mol Life Sci.54(12): 1350–64.doi:10.1007/s000180050259.PMID9893710.
- ^Dame RT (2005). “The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin”.Mol. Microbiol.56(4): 858–70.doi:10.1111/j.1365-2958.2005.04598.x.PMID15853876.
- ^Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (1997). “Crystal structure of the nucleosome core particle at 2.8 A resolution”.Nature.389(6648): 251–60.Bibcode:1997Natur.389..251L.doi:10.1038/38444.PMID9305837.
- ^Jenuwein T, Allis CD (2001). “Translating the histone code”.Science.293(5532): 1074–80.doi:10.1126/science.1063127.PMID11498575.
- ^Ito T (2003). “Nucleosome assembly and remodelling”.Curr Top Microbiol Immunol.Current Topics in Microbiology and Immunology.274:1–22.doi:10.1007/978-3-642-55747-7_1.ISBN978-3-540-44208-0.PMID12596902.
- ^Thomas JO (2001). “HMG1 and 2: architectural DNA-binding proteins”.Biochem Soc Trans.29(Pt 4): 395–401.doi:10.1042/BST0290395.PMID11497996.
- ^Grosschedl R, Giese K, Pagel J (1994).“HMG domain proteins: architectural elements in the assembly of nucleoprotein structures”.Trends Genet.10(3): 94–100.doi:10.1016/0168-9525(94)90232-1.PMID8178371.
- ^Iftode C, Daniely Y, Borowiec JA (1999). “Replication protein A (RPA): the eukaryotic SSB”.Crit Rev Biochem Mol Biol.34(3): 141–80.doi:10.1080/10409239991209255.PMID10473346.
- ^Created fromPDB 1LMBLưu trữ2008-01-06 tạiWayback Machine
- ^Myers LC, Kornberg RD (2000).“Mediator of transcriptional regulation”.Annu Rev Biochem.69:729–49.doi:10.1146/annurev.biochem.69.1.729.PMID10966474.
- ^Spiegelman BM, Heinrich R (2004). “Biological control through regulated transcriptional coactivators”.Cell.119(2): 157–67.doi:10.1016/j.cell.2004.09.037.PMID15479634.
- ^Li Z, Van Calcar S, Qu C, Cavenee WK, Zhang MQ, Ren B (2003).“A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells”.Proc Natl Acad Sci USA.100(14): 8164–9.Bibcode:2003PNAS..100.8164L.doi:10.1073/pnas.1332764100.PMC166200.PMID12808131.
- ^Created fromPDB 1RVALưu trữ2008-01-06 tạiWayback Machine
- ^Bickle TA, Krüger DH (1993).“Biology of DNA restriction”.Microbiol Rev.57(2): 434–50.PMC372918.PMID8336674.
- ^abDoherty AJ, Suh SW (2000).“Structural and mechanistic conservation in DNA ligases”.Nucleic Acids Res.28(21): 4051–8.doi:10.1093/nar/28.21.4051.PMC113121.PMID11058099.
- ^Schoeffler AJ, Berger JM (2005). “Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism”.Biochem Soc Trans.33(Pt 6): 1465–70.doi:10.1042/BST20051465.PMID16246147.
- ^Tuteja N, Tuteja R (2004). “Unraveling DNA helicases. Motif, structure, mechanism and function”.Eur J Biochem.271(10): 1849–63.doi:10.1111/j.1432-1033.2004.04094.x.PMID15128295.
- ^Joyce CM, Steitz TA (1995).“Polymerase structures and function: variations on a theme?”.J Bacteriol.177(22): 6321–9.PMC177480.PMID7592405.
- ^Hubscher U, Maga G, Spadari S (2002).“Eukaryotic DNA polymerases”.Annu Rev Biochem.71:133–63.doi:10.1146/annurev.biochem.71.090501.150041.PMID12045093.
- ^Johnson A, O'Donnell M (2005).“Cellular DNA replicases: components and dynamics at the replication fork”.Annu Rev Biochem.74:283–315.doi:10.1146/annurev.biochem.73.011303.073859.PMID15952889.
- ^Tarrago-Litvak L, Andréola ML, Nevinsky GA, Sarih-Cottin L, Litvak S (ngày 1 tháng 5 năm 1994). “The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention”.FASEB J.8(8): 497–503.PMID7514143.
- ^Martinez E (2002).“Multi-protein complexes in eukaryotic gene transcription”.Plant Mol Biol.50(6): 925–47.doi:10.1023/A:1021258713850.PMID12516863.
- ^Created fromPDB 1M6GLưu trữ2010-01-10 tạiWayback Machine
- ^Cremer T, Cremer C (2001). “Chromosome territories, nuclear architecture and gene regulation in mammalian cells”.Nature Reviews Genetics.2(4): 292–301.doi:10.1038/35066075.PMID11283701.
- ^Pál C, Papp B, Lercher MJ (2006). “An integrated view of protein evolution”.Nature Reviews Genetics.7(5): 337–48.doi:10.1038/nrg1838.PMID16619049.
- ^O'Driscoll M, Jeggo PA (2006). “The role of double-strand break repair – insights from human genetics”.Nature Reviews Genetics.7(1): 45–54.doi:10.1038/nrg1746.PMID16369571.
- ^Vispé S, Defais M (1997). “Mammalian Rad51 protein: a RecA homologue with pleiotropic functions”.Biochimie.79(9–10): 587–92.doi:10.1016/S0300-9084(97)82007-X.PMID9466696.
- ^Neale MJ, Keeney S (2006). “Clarifying the mechanics of DNA strand exchange in meiotic recombination”.Nature.442(7099): 153–8.Bibcode:2006Natur.442..153N.doi:10.1038/nature04885.PMID16838012.
- ^Dickman MJ, Ingleston SM, Sedelnikova SE, Rafferty JB, Lloyd RG, Grasby JA, Hornby DP (2002). “The RuvABC resolvasome”.Eur J Biochem.269(22): 5492–501.doi:10.1046/j.1432-1033.2002.03250.x.PMID12423347.
- ^Joyce GF (2002). “The antiquity of RNA-based evolution”.Nature.418(6894): 214–21.Bibcode:2002Natur.418..214J.doi:10.1038/418214a.PMID12110897.
- ^Orgel LE (2004). “Prebiotic chemistry and the origin of the RNA world”.Crit Rev Biochem Mol Biol.39(2): 99–123.doi:10.1080/10409230490460765.PMID15217990.
- ^Davenport RJ (2001). “Ribozymes. Making copies in the RNA world”.Science.292(5520): 1278.doi:10.1126/science.292.5520.1278a.PMID11360970.
- ^Szathmáry E (1992).“What is the optimum size for the genetic alphabet?”.Proc Natl Acad Sci USA.89(7): 2614–8.Bibcode:1992PNAS...89.2614S.doi:10.1073/pnas.89.7.2614.PMC48712.PMID1372984.
- ^Lindahl T (1993). “Instability and decay of the primary structure of DNA”.Nature.362(6422): 709–15.Bibcode:1993Natur.362..709L.doi:10.1038/362709a0.PMID8469282.
- ^Vreeland RH, Rosenzweig WD, Powers DW (2000). “Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal”.Nature.407(6806): 897–900.doi:10.1038/35038060.PMID11057666.
- ^Hebsgaard MB, Phillips MJ, Willerslev E (2005).“Geologically ancient DNA: fact or artefact?”.Trends Microbiol.13(5): 212–20.doi:10.1016/j.tim.2005.03.010.PMID15866038.
- ^Nickle DC, Learn GH, Rain MW, Mullins JI, Mittler JE (2002).“Curiously modern DNA for a" 250 million-year-old "bacterium”.J Mol Evol.54(1): 134–7.doi:10.1007/s00239-001-0025-x.PMID11734907.
- ^Callahan MP, Smith KE, Cleaves HJ, Ruzicka J, Stern JC, Glavin DP, House CH, Dworkin JP (tháng 8 năm 2011).“Carbonaceous meteorites contain a wide range of extraterrestrial nucleobases”.Proc. Natl. Acad. Sci. U.S.A.108(34): 13995–8.Bibcode:2011PNAS..10813995C.doi:10.1073/pnas.1106493108.PMC3161613.PMID21836052.
- ^Steigerwald, John (ngày 8 tháng 8 năm 2011).“NASA Researchers: DNA Building Blocks Can Be Made in Space”.NASA.Lưu trữbản gốc ngày 7 tháng 7 năm 2018.Truy cập ngày 10 tháng 8 năm 2011.
- ^ScienceDaily Staff (ngày 9 tháng 8 năm 2011).“DNA Building Blocks Can Be Made in Space, NASA Evidence Suggests”.ScienceDaily.Lưu trữbản gốc ngày 5 tháng 9 năm 2011.Truy cập ngày 9 tháng 8 năm 2011.
- ^Marlaire, Ruth (ngày 3 tháng 3 năm 2015).“NASA Ames Reproduces the Building Blocks of Life in Laboratory”.NASA.Lưu trữbản gốc ngày 18 tháng 1 năm 2017.Truy cập ngày 5 tháng 3 năm 2015.
- ^Goff SP, Berg P (1976). “Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells”.Cell.9(4 PT 2): 695–705.doi:10.1016/0092-8674(76)90133-1.PMID189942.
- ^Houdebine LM (2007). “Transgenic animal models in biomedical research”.Methods Mol Biol.360:163–202.doi:10.1385/1-59745-165-7:163.ISBN1-59745-165-7.PMID17172731.
- ^Daniell H, Dhingra A (2002).“Multigene engineering: dawn of an exciting new era in biotechnology”.Current Opinion in Biotechnology.13(2): 136–41.doi:10.1016/S0958-1669(02)00297-5.PMC3481857.PMID11950565.
- ^Job D (2002). “Plant biotechnology in agriculture”.Biochimie.84(11): 1105–10.doi:10.1016/S0300-9084(02)00013-5.PMID12595138.
- ^Collins A, Morton NE (1994).“Likelihood ratios for DNA identification”.Proc Natl Acad Sci USA.91(13): 6007–11.Bibcode:1994PNAS...91.6007C.doi:10.1073/pnas.91.13.6007.PMC44126.PMID8016106.
- ^Weir BS, Triggs CM, Starling L, Stowell LI, Walsh KA, Buckleton J (1997).“Interpreting DNA mixtures”.J Forensic Sci.42(2): 213–22.PMID9068179.
- ^Jeffreys AJ, Wilson V, Thein SL (1985). “Individual-specific 'fingerprints' of human DNA”.Nature.316(6023): 76–9.Bibcode:1985Natur.316...76J.doi:10.1038/316076a0.PMID2989708.
- ^Colin Pitchfork — first murder conviction on DNA evidence also clears the prime suspectForensic Science Service Accessed ngày 23 tháng 12 năm 2006
- ^“DNA Identification in Mass Fatality Incidents”(PDF).U.S. Department of Justice, Office of Justice Programs. tháng 9 năm 2006.Lưu trữ(PDF)bản gốc ngày 2 tháng 1 năm 2017.Truy cập ngày 4 tháng 12 năm 2016.
- ^“"Paternity Blood Tests That Work Early in a Pregnancy "New York Times ngày 20 tháng 6 năm 2012”.Lưu trữbản gốc ngày 22 tháng 7 năm 2016.Truy cập ngày 8 tháng 11 năm 2016.
- ^abBreaker, Ronald R.; Joyce, Gerald F. (ngày 12 tháng 1 năm 1994).“A DNA enzyme that cleaves RNA”(PDF).Chemistry & Biology.1(4): 223–229.doi:10.1016/1074-5521(94)90014-0.ISSN1074-5521.PMID9383394.Bản gốc(PDF)lưu trữ ngày 16 tháng 1 năm 2017.Truy cập ngày 5 tháng 12 năm 2016.
- ^Chandra, Madhavaiah; Sachdeva, Amit; Silverman, Scott K.“DNA-catalyzed sequence-specific hydrolysis of DNA”.Nature Chemical Biology.5(10): 718–720.doi:10.1038/nchembio.201.PMC2746877.PMID19684594.Bản gốclưu trữ ngày 5 tháng 1 năm 2021.Truy cập ngày 5 tháng 12 năm 2016.
- ^Carmi, Nir; Shultz, Lisa A.; Breaker, Ronald R. (ngày 12 tháng 1 năm 1996).“In vitro selection of self-cleaving DNAs”.Chemistry & Biology.3(12): 1039–1046.doi:10.1016/S1074-5521(96)90170-2.ISSN1074-5521.PMID9000012.Bản gốclưu trữ ngày 14 tháng 1 năm 2021.Truy cập ngày 5 tháng 12 năm 2016.
- ^Torabi, Seyed-Fakhreddin; Wu, Peiwen; McGhee, Claire E.; Chen, Lu; Hwang, Kevin; Zheng, Nan; Cheng, Jianjun; Lu, Yi (ngày 12 tháng 5 năm 2015).“In vitro selection of a sodium-specific DNAzyme and its application in intracellular sensing”.Proceedings of the National Academy of Sciences.112(19): 5903–5908.doi:10.1073/pnas.1420361112.ISSN0027-8424.PMC4434688.PMID25918425.Bản gốclưu trữ ngày 30 tháng 10 năm 2018.Truy cập ngày 8 tháng 11 năm 2016.
- ^Baldi, Pierre;Brunak, Soren (2001).Bioinformatics: The Machine Learning Approach.MIT Press.ISBN978-0-262-02506-5.OCLC45951728.
- ^Gusfield, Dan.Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology.Cambridge University Press,ngày 15 tháng 1 năm 1997.ISBN 978-0-521-58519-4.
- ^Sjölander K (2004). “Phylogenomic inference of protein molecular function: advances and challenges”.Bioinformatics.20(2): 170–9.doi:10.1093/bioinformatics/bth021.PMID14734307.
- ^Mount DM (2004).Bioinformatics: Sequence and Genome Analysis(ấn bản 2). Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press.ISBN0-87969-712-1.OCLC55106399.
- ^Rothemund PW (2006). “Folding DNA to create nanoscale shapes and patterns”.Nature.440(7082): 297–302.Bibcode:2006Natur.440..297R.doi:10.1038/nature04586.PMID16541064.
- ^Andersen ES, Dong M, Nielsen MM, Jahn K, Subramani R, Mamdouh W, Golas MM, Sander B, Stark H, Oliveira CL, Pedersen JS, Birkedal V, Besenbacher F, Gothelf KV, Kjems J (2009). “Self-assembly of a nanoscale DNA box with a controllable lid”.Nature.459(7243): 73–6.Bibcode:2009Natur.459...73A.doi:10.1038/nature07971.PMID19424153.
- ^Ishitsuka Y, Ha T (2009). “DNA nanotechnology: a nanomachine goes live”.Nat Nanotechnol.4(5): 281–2.Bibcode:2009NatNa...4..281I.doi:10.1038/nnano.2009.101.PMID19421208.
- ^Aldaye FA, Palmer AL, Sleiman HF (2008). “Assembling materials with DNA as the guide”.Science.321(5897): 1795–9.Bibcode:2008Sci...321.1795A.doi:10.1126/science.1154533.PMID18818351.
- ^Wray GA (2002).“Dating branches on the Tree of Life using DNA”.Genome Biol.3(1): reviews0001.1–reviews0001.7.doi:10.1046/j.1525-142X.1999.99010.x.PMC150454.PMID11806830.
- ^Lost Tribes of Israel,Nova, PBS airdate: ngày 22 tháng 2 năm 2000. Transcript available fromPBS.orgLưu trữ2008-09-16 tạiWayback Machine.Truy cập ngày 4 tháng 3 năm 2006.
- ^Kleiman, Yaakov."The Cohanim/DNA Connection: The fascinating story of how DNA studies confirm an ancient biblical tradition".Lưu trữ2016-11-22 tạiWayback Machineaish.com(ngày 13 tháng 1 năm 2000). Truy cập ngày 4 tháng 3 năm 2006.
- ^Goldman N, Bertone P, Chen S, Dessimoz C, LeProust EM, Sipos B, Birney E (ngày 23 tháng 1 năm 2013).“Towards practical, high-capacity, low-maintenance information storage in synthesized DNA”.Nature.494(7435): 77–80.Bibcode:2013Natur.494...77G.doi:10.1038/nature11875.PMC3672958.PMID23354052.
- ^Naik, Gautam (ngày 24 tháng 1 năm 2013).“Storing Digital Data in DNA”.The Wall Street Journal.Bản gốclưu trữ ngày 27 tháng 10 năm 2016.Truy cập ngày 24 tháng 1 năm 2013.
- ^“Towards practical, high-capacity, low-maintenance information storage in synthesized DNA”.Nature.Lưu trữbản gốc ngày 5 tháng 10 năm 2016.Truy cập 8 tháng 2 năm 2018.
- ^Emerging Technology Final, Dandekar T., Lopez, D., Programmable bacterial membranes with active DNA storage; presentation for the University of Würzburg for the Royal Society for Chemistry, Luân Đôn, 2016-06-29
- ^“Patent DE102013004584A1 - Molekulare hoch integrierte Datenspeicherung über aktiv gesteuerte DNA Molecular highly integrated data storage via active control DNA”.Google Books.Lưu trữbản gốc ngày 30 tháng 10 năm 2016.Truy cập 8 tháng 2 năm 2018.
- ^Miescher, Friedrich (1871)"Ueber die chemische Zusammensetzung der Eiterzellen"Lưu trữ2016-05-11 tạiWayback Machine(On the chemical composition of pus cells),Medicinisch-chemische Untersuchungen,4:441–460.From p. 456:Lưu trữ2016-04-24 tạiWayback Machine"Ich habe mich daher später mit meinen Versuchen an die ganzen Kerne gehalten, die Trennung der Körper, die ich einstweilen ohne weiteres Präjudiz als lösliches und unlösliches Nuclein bezeichnen will, einem günstigeren Material überlassend."(Therefore, in my experiments I subsequently limited myself to the whole nucleus, leaving to a more favorable material the separation of the substances, that for the present, without further prejudice, I will designate as soluble and insoluble nuclear material ( "Nuclein" ).)
- ^Dahm R (2008). “Discovering DNA: Friedrich Miescher and the early years of nucleic acid research”.Hum. Genet.122(6): 565–81.doi:10.1007/s00439-007-0433-0.PMID17901982.
- ^Xem:
- Albrect Kossel (1879)"Ueber Nucleïn der Hefe"Lưu trữ2016-12-21 tạiWayback Machine(On nuclein in yeast)Zeitschrift für physiologische Chemie,3:284-291.
- Albrect Kossel (1880)"Ueber Nucleïn der Hefe II"Lưu trữ2016-12-21 tạiWayback Machine(On nuclein in yeast, Part 2)Zeitschrift für physiologische Chemie,4:290-295.
- Albrect Kossel (1881)"Ueber die Verbreitung des Hypoxanthins im Thier- und Pflanzenreich"Lưu trữ2016-12-21 tạiWayback Machine(On the distribution of hypoxanthins in the animal and plant kingdoms)Zeitschrift für physiologische Chemie,5:267-271.
- Albrect Kossel,Untersuchungen über die Nucleine und ihre Spaltungsprodukte[Investigations into nuclein and its cleavage products] (Strassburg, Germany: K.J. Trübne, 1881), 19 pages.
- Albrect Kossel (1882)Hoppe-Seyler's Zeitschrift Für Physiologische Chemie "Ueber Xanthin und Hypoxanthin"Lưu trữ2016-12-20 tạiWayback Machine(On xanthin and hypoxanthin),Zeitschrift für physiologische Chemie,6:422-431.
- Albrect Kossel (1883)"Hoppe-Seyler's Zeitschrift Für Physiologische ChemieLưu trữ2016-12-21 tạiWayback Machine(On the chemistry of the cell nucleus),Zeitschrift für physiologische Chemie,7:7-22.
- Albrect Kossel (1886) "Weitere Beiträge zur Chemie des Zellkerns" (Further contributions to the chemistry of the cell nucleus),Zeitschrift für Physiologische Chemie,10:248-264. Có thể xem trực tuyến tại:Viện Lịch sử Khoa học Max Planck, Berlin, ĐứcLưu trữ2014-07-14 tạiWayback Machine.Tại trang 264, Kossel remarked presciently:"Der Erforschung der quantitativen Verhältnisse der vier stickstoffreichen Basen, der Abhängigkeit ihrer Menge von den physiologischen Zuständen der Zelle, verspricht wichtige Aufschlüsse über die elementaren physiologisch-chemischen Vorgänge."(The study of the quantitative relations of the four nitrogenous bases — [and] of the dependence of their quantity on the physiological states of the cell — promises important insights into the fundamental physiological-chemical processes.)
- ^Jones ME (tháng 9 năm 1953).“Albrecht Kossel, A Biographical Sketch”.Yale Journal of Biology and Medicine.National Center for Biotechnology Information.26(1): 80–97.PMC2599350.PMID13103145.
- ^Levene P (ngày 1 tháng 12 năm 1919). “The structure of yeast nucleic acid”.J Biol Chem.40(2): 415–24.
- ^Xem:
- W. T. Astbury and Florence O. Bell (1938) "Some recent developments in the X-ray study of proteins and related structures,"Cold Spring Harbor Symposia on Quantitative Biology,6:109-121. Có thể xem trực tuyến tại:Trang web của Đại học Leeds.Lưu trữ2014-07-14 tạiWayback Machine
- Astbury, W. T., (1947) "X-ray studies of nucleic acids,"Symposia of the Society for Experimental Biology,1:66-76. Có thể xem trực tuyến tại:Trang web của Đại học Bang Oregon.Lưu trữ2014-07-05 tạiWayback Machine
- ^Koltsov đề xuất rằng các thông tin di truyền của một tế bào được mã hoá trong một chuỗi dài các acid amino. Xem:
- Н. К. Кольцов, "Физико-химические основы морфологии" (The physical-chemical basis of morphology) -- speech given at the 3rd All-Union Meeting of Zoologist, Anatomists, and Histologists at Leningrad, U.S.S.R., 12 tháng 12 năm 1927.
- In lại trong:Успехи экспериментальной биологии(Advances in Experimental Biology), series B, 7 (1):?-? (1928).
- In lại bằng tiếng Đức trong: Nikolaj K. Koltzoff (1928) "Physikalisch-chemische Grundlagen der Morphologie" (The physical-chemical basis of morphology),Biologisches Zentralblatt,48(6): 345-369.
- In 1934, Koltsov contended that the proteins that contain a cell's genetic information replicate. Xem: N. K. Koltzoff (5 tháng 10 năm 1934) "The structure of the chromosomes in the salivary glands of Drosophila,"Science,80(2075): 312-313. Từ trang 313: "I think that the size of the chromosomes in the salivary glands [of Drosophila] is determined through the multiplication ofgenonemes.By this term I designate the axial thread of the chromosome, in which the geneticists locate the linear combination of genes;… In the normal chromosome there is usually only one genoneme; before cell-division this genoneme has become divided into two strands. "
- ^Soyfer VN (2001). “The consequences of political dictatorship for Russian science”.Nature Reviews Genetics.2(9): 723–729.doi:10.1038/35088598.PMID11533721.
- ^Griffith F (tháng 1 năm 1928).“The significance of pneumococcal types”.The Journal of Hygiene (Luân Đôn).27(2): 113–59.doi:10.1017/S0022172400031879.PMC2167760.PMID20474956.
- ^Lorenz MG, Wackernagel W (1994).“Bacterial gene transfer by natural genetic transformation in the environment”.Microbiol. Rev.58(3): 563–602.PMC372978.PMID7968924.
- ^Avery OT, Macleod CM, McCarty M (1944).“Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types: Induction of Transformation by a Desoxyribonucleic Acid Fraction Isolated from Pneumococcus Type Iii”.J Exp Med.79(2): 137–158.doi:10.1084/jem.79.2.137.PMC2135445.PMID19871359.
- ^Hershey AD, Chase M (1952).“Independent Functions of Viral Protein and Nucleic Acid in Growth of Bacteriophage”.J Gen Physiol.36(1): 39–56.doi:10.1085/jgp.36.1.39.PMC2147348.PMID12981234.
- ^The B-DNA X-ray patternon the right of this linked imagewas obtained byRosalind FranklinvàRaymond Goslingin May 1952 at high hydration levels of DNA and it has been labeled as "Photo 51"
- ^Nature ArchivesDouble Helix of DNA: 50 YearsLưu trữ2015-04-05 tạiWayback Machine
- ^“Rosalind Franklin's x-ray diffraction photo of structure B”.Osulibrary.oregonstate.edu. Lưu trữ bản gốc ngày 30 tháng 1 năm 2009.Truy cập ngày 6 tháng 2 năm 2011.Quản lý CS1: bot: trạng thái URL ban đầu không rõ (liên kết)
- ^The Nobel Prize in Physiology or Medicine 1962Lưu trữ2013-05-30 tạiWayback MachineNobelprize.org Accessed 22 December 06
- ^Maddox B (ngày 23 tháng 1 năm 2003).“The double helix and the 'wronged heroine'”(PDF).Nature.421(6921): 407–408.Bibcode:2003Natur.421..407M.doi:10.1038/nature01399.PMID12540909.Bản gốc(PDF)lưu trữ ngày 17 tháng 10 năm 2016.Truy cập ngày 8 tháng 11 năm 2016.
- ^Crick, F.H.C.On degenerate templates and the adaptor hypothesis (PDF).genome.wellcome.ac.uk (Lecture, 1955). Truy cập ngày 22 tháng 12 năm 2006.
- ^Meselson M, Stahl FW (1958).“The replication of DNA in Escherichia coli”.Proc Natl Acad Sci USA.44(7): 671–82.Bibcode:1958PNAS...44..671M.doi:10.1073/pnas.44.7.671.PMC528642.PMID16590258.
- ^The Nobel Prize in Physiology or Medicine 1968Lưu trữ2016-12-10 tạiWayback MachineNobelprize.org Accessed 22 December 06
Tham khảo
[sửa|sửa mã nguồn]- Berry, Andrew;Watson, James.(2003).DNA: the secret of life.New York: Alfred A. Knopf.ISBN0-375-41546-7.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- Calladine, Chris R.; Drew, Horace R.; Luisi, Ben F.; Travers, Andrew A. (2003).Understanding DNA: the molecule & how it works.Amsterdam: Elsevier Academic Press.doi:10.1016/0307-4412(94)90110-4.ISBN0-12-155089-3.
- Dennis, Carina; Julie Clayton (2003).50 years of DNA.Basingstoke: Palgrave Macmillan.doi:10.1007/978-1-137-11781-6.ISBN1-4039-1479-6.Bản gốclưu trữ ngày 4 tháng 7 năm 2021.Truy cập ngày 11 tháng 12 năm 2016.
- Judson, Horace F.1979.The Eighth Day of Creation: Makers of the Revolution in Biology.Touchstone Books,ISBN 0-671-22540-5.2nd edition: Cold Spring Harbor Laboratory Press, 1996 paperback:ISBN 0-87969-478-5.doi:10.1086/383894
- Olby, Robert C.(1994).The path to the double helix: the discovery of DNA(PDF).New York: Dover Publications.ISBN0-486-68117-3.Bản gốc(PDF)lưu trữ ngày 20 tháng 12 năm 2016.Truy cập ngày 11 tháng 12 năm 2016.,first published in October 1974 by MacMillan, with foreword by Francis Crick; the definitive DNA textbook, revised in 1994 with a 9-page postscript
- Micklas, David. 2003.DNA Science: A First Course.Cold Spring Harbor Press:ISBN 978-0-87969-636-8
- Ridley, Matt(2006).Francis Crick: discoverer of the genetic code.Ashland, OH: Eminent Lives, Atlas Books.doi:10.1056/NEJMbkrev56783.ISBN0-06-082333-X.Bản gốclưu trữ ngày 4 tháng 7 năm 2021.Truy cập ngày 11 tháng 12 năm 2016.
- Olby, Robert C. (2009).Francis Crick: A Biography.Plainview, N.Y: Cold Spring Harbor Laboratory Press.doi:10.1002/bmb.20462.ISBN0-87969-798-9.
- Rosenfeld, Israel. 2010.DNA: A Graphic Guide to the Molecule that Shook the World.Columbia University Press:ISBN 978-0-231-14271-7
- Schultz, Mark and Zander Cannon. 2009.The Stuff of Life: A Graphic Guide to Genetics and DNA.Hill and Wang:ISBN 0-8090-8947-5
- Stent, Gunther Siegmund;Watson, James. (1980).The Double Helix: A Personal Account of the Discovery of the Structure of DNA.New York: Norton.doi:10.1021/ed045p623.2.ISBN0-393-95075-1.Bản gốclưu trữ ngày 11 tháng 5 năm 2019.Truy cập ngày 11 tháng 12 năm 2016.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- Watson, James. 2004.DNA: The Secret of Life.Random House:ISBN 978-0-09-945184-6
- Wilkins, Maurice(2003).The third man of the double helix the autobiography of Maurice Wilkins.Cambridge, Eng: University Press.ISBN0-19-860665-6.
Liên kết ngoài
[sửa|sửa mã nguồn]| WikiquoteAnh ngữ sưu tập danh ngôn về: |
| Wikimedia Commons có thêm hình ảnh và phương tiện truyền tải vềDNA. |
- DNAtạiTừ điển bách khoa Việt Nam
- DNAtạiEncyclopædia Britannica(tiếng Anh)
- DNAtrênDMOZ
- DNA binding site prediction on proteinLưu trữ2007-03-06 tạiWayback Machine
- DNA the Double Helix GameLưu trữ2016-11-21 tạiWayback MachineFrom the official Nobel Prize web site
- DNA under electron microscopeLưu trữ2007-06-23 tạiWayback Machine
- Dolan DNA Learning CenterLưu trữ2016-10-30 tạiWayback Machine
- Double Helix: 50 years of DNALưu trữ2015-04-05 tạiWayback Machine,Nature
- ProteopediaDNALưu trữ2016-11-09 tạiWayback Machine
- ProteopediaForms of DNALưu trữ2016-11-09 tạiWayback Machine
- ENCODE threads explorerLưu trữ2019-01-06 tạiWayback MachineENCODE home page.Nature (journal)
- Double Helix 1953–2003National Centre for Biotechnology Education
- DNA from the Beginning Study GuideLưu trữ2016-04-06 tạiWayback MachineGenetic Education Modules for Teachers
- PDB Molecule of the MonthDNALưu trữ2016-12-03 tạiWayback Machine
- Olby R(2003).“Quiet debut for the double helix”(PDF).Nature.421(6921): 402–5.doi:10.1038/nature01397.PMID12540907.Bản gốc(PDF)lưu trữ ngày 16 tháng 6 năm 2010.Truy cập ngày 13 tháng 11 năm 2016.
- Ellen Moody's TeachingLưu trữ2010-06-16 tạiWayback MachineRosalind Franklin's contributions to the study of DNA
- The Register of Francis Crick Personal Papers 1938 - 2007Lưu trữ2007-08-25 tạiWayback Machinetại Mandeville Special Collections Library, Geisel Library,Đại học California, San Diego
- U.S. National DNA DayLưu trữ2016-12-01 tạiWayback Machine—watch videos and participate in real-time chat with top scientists
- “Clue to chemistry of heredity found”(PDF).The New York Times.ngày 13 tháng 6 năm 1953.Bản gốc(PDF)lưu trữ ngày 26 tháng 8 năm 2012.Truy cập ngày 9 tháng 4 năm 2010.Tờ báo Mỹ đầu tiên đưa tin về việc phát hiện ra cấu trúc DNA.
- Seven-page, handwritten letter that Crick sent to his 12-year-old son Michael in 1953 describing the structure of DNA.Lưu trữ2015-07-18 tạiWayback MachineXemCrick’s medal goes under the hammerLưu trữ2016-04-02 tạiWayback Machine,Nature, 5 tháng 4 năm 2013.
- 3D map of DNA reveals hidden loops that allow genes to work togetherLưu trữ2016-11-09 tạiWayback Machine(11 tháng 12 năm 2014),Science(Daily News)