Họ Nguyệt quế
| Họ Nguyệt quế | |
|---|---|
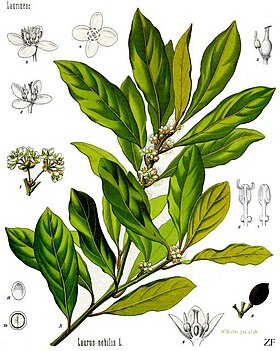 Nguyệt quế (Laurus nobilis) | |
| Phân loại khoa học | |
| Giới(regnum) | Plantae |
| (không phân hạng) | Angiospermae |
| (không phân hạng) | Magnoliidae |
| Bộ(ordo) | Laurales |
| Họ(familia) | Lauraceae Juss.,1789[1] |
| Chi điển hình | |
| Laurus L.,1753 | |
| Các chi | |
Nhiều, xem văn bản. | |
Lauraceaehayhọ Nguyệt quế,trong một số sách vở về thực vật tại Việt Nam gọi làhọ Long nãohayhọ Quế,nhưng tại Wikipedia gọi theo tên thứ nhất do tên khoa học của họ này lấy theo tên gọi của chinguyệt quếlàLaurusmà không lấy theo tên gọi của chi chứalong nãovàquếlàCinnamomum.Họ này là một nhómthực vật có hoanằm trongbộ Nguyệt quế(Laurales). Họ này chứa khoảng 55 chi và trên 2.000 (có thể nhiều tới 4.000) loài,[2][3]phân bố rộng khắp thế giới, chủ yếu trong các khu vực nhiệt đới và ôn đới ấm, đặc biệt là ở khu vựcĐông Nam ÁvàNam Mỹ(Brasil). Chúng chủ yếu là các loạicây thường xanhthân gỗ hay cây bụi có hương thơm, nhưng chiSassafrasvà một hoặc hai chi khác là các loạicây sớm rụng,cònCassytha(tơ xanh) là chi chứa các loàidây leosốngký sinh.
Các loại cây thân gỗ trong họ Nguyệt quế chiếm ưu thế trong các cánhrừng nguyệt quếtrên thế giới, có tại một số khu vực ẩm ướt của vùng cận nhiệt đới và ôn đới thuộc Bắc và Nam bán cầu, bao gồm các đảo thuộcMacaronesia,miền namNhật Bản,Madagascar,và miền trungChile.
Có ba mục đích sử dụng chính của các loài cây trong họ này. Hàm lượng cao của tinh dầu tìm thấy trong nhiều loại thuộc họ Lauraceae. Các tinh dầu này là nguyên liệu quan trọng cho nhiều gia vị và sản xuất nước hoa.Lê dầucũng cho quả chứa nhiều tinh dầu hiện nay được trồng nhiều tại khu vực nhiệt đới trên thế giới. Một vài loài còn cung cấp gỗ.
Trong số được biết nhiều nhất là các chi sau, là các chi có các loài được sử dụng ở phạm vi thương mại:
- Cinnamomum:Các loàiquế,long não.
- Laurus:Nguyệt quế.
- Lindera:
- Persea:Lê dầu.
- Sassafras:Sát mộc.
Các tông và chi
[sửa|sửa mã nguồn]Các dòng dõi chính trong Lauraceae liệt kê dưới đây về cơ bản dựa theo Rohwer (2000), Chanderbaliet al.(2001), Rohwer & Rudolph (2005) và Songet al.(2017).[4][5][6][7]
- HypodaphnideaeReveal:1 chi, 1 loài ở vùng nhiệt đới Tây Phi.
- CryptocaryeaeNees:13 chi, khoảng 775 loài. Liên nhiệt đới, một số loài cận nhiệt đới tớiNew Zealand.
- Aspidostemon
- Beilschmiedia(bao gồm cảBernieria,Hufelandia,Lauromerrillia,Nesodaphne,Thouvenotia,Tylostemon): Chắp, két, mong, sang gia, son, chập, bạch mi, quỳnh nam.
- Cryptocarya(bao gồm cảCaryodaphne,Dahlgrenodendron,Icosandra,Massoia,Pseudocryptocarya,Ravensara): Cà đuối, hậu xác quế.
- Endiandra(bao gồm cảBrassiodendron): Khuyết hùng, khuyết nhị, vừ, thổ nam.
- Eusideroxylon
- Hexapora(bao gồm cảMicropora).
- Potameia(có thể bao gồm cảSyndiclis)
- Potoxylon
- Sinopora
- Syndiclis:Dẹ, rạch, du quả chương.
- Triadodaphne
- Yasunia
- CassytheaeDumortier= CassythaceaeLindley:1 chi, 24 loài. Nhiệt đới, đặc biệt là tạiAustralia,bao gồm cả vùng ôn đới ấm tại đây.
- Cassytha:Tơ xanh, vô căn đằng.
- NeocinnamomeaeYu Song, W. B. Yu & Y. H. Tan:1 chi, 6 loài. Đông Nam Á, tây Malesia (Sumatra).
- Neocinnamomum:Rè, tân chương.
- CaryodaphnopsideaeYu Song, W. B. Yu & Y. H. Tan:1 chi, 15 loài. Trung và Nam Mỹ, Đông Nam Á tớiPhilippinesvàBorneo.
- Caryodaphnopsis:Giả sụ, long não quả chanh.
- NhómMelizaurusvà v.v.: 4-5 chi, 23 loài. Từ Trung Mỹ (Costa Rica) tới Nam Mỹ vàTrinidad.
- Anaueria
- Chlorocardium
- Mezilaurus(bao gồm cảClinostemon,Silvia).
- Sextonia
- Williamodendron
- PerseeaeNees:7 chi, 430 loài.
- Alseodaphne(bao gồm cảStemmatodaphne): Sụ, vàng trắng.
- Alseodaphnopsis.Tách ra từAlseodaphne.[8]
- Apollonias
- Dehaasia(bao gồm cảCyanodaphne): Cà đuối, liên quế.
- Machilus
- Nothaphoebe:Giả sụ, bời lời.
- Persea(có thể bao gồm cảMachilus,Apollonias): Bơ, lê dầu, kháo, sụ, vàng giền, ngạc lê.
- Phoebe:Sụ, nam.
- CinnamomeaeNees:Khoảng 12-20 chi, 1.165 loài. Liên nhiệt đới. ChiSassafrascó ở vùng ôn đới.
- Aiouea(bao gồm cảMocinnodaphne)
- Aniba(bao gồm cảAydendron).
- Cinnamomum:Quế, long não, rè, chương.
- Damburneya:Tách ra từNectandra.
- Dicypellium
- Endlicheria(bao gồm cảAmpelodaphne,Goeppertia,Huberodaphne).
- Gamanthera
- Kubitzkia
- Licaria(bao gồm cảAcrodiclidium,Chanekia,Misanteca,Nobeliodendron).
- Mespilodaphne(bao gồm cảDendrodaphne,Sassafridium).
- Nectandra
- Ocotea(bao gồm cảBellota,Camphoromoea,Evonymodaphne,Gymnobalanus,Leptodaphne,Oreodaphne,Petelanthera,Teleiandra).
- Paraia
- Phyllostemonodaphne
- Pleurothyrium
- Povedadaphne
- Rhodostemonodaphne
- Sassafras(bao gồm cảPseudosassafras): Sát mộc, sát thụ: Chuyển từ Laureae sang.
- Umbellularia(bao gồm cảSciadiodaphne).
- Urbanodendron
- LaureaeLe Maout & Decaisne:Khoảng 9 chi, 545 loài. Nhiệt đới và cận nhiệt đới, đặc biệt tại Đông Nam Á và Malesia, hiếm ở vùng ôn đới.
- Actinodaphne:Bộp, hoàng nhục nam.
- Dodecadenia
- Laurus:Nguyệt quế.
- Lindera(bao gồm cảDaphnidium,Iteadaphne,Parabenzoin): Liên đàn, sơn hồ tiêu.
- Litsea(bao gồm cảTetranthera): Bời lời, mộc khương tử.
- Neolitsea(bao gồm cảBryantea): Tân bời, tân mộc khương tử.
- Parasassafras
- Sinosassafras
- Incertae sedis:
- Cinnadenia:Dự. 2 loài ở Đông Nam Á, có thể gần vớiLitsea.
- Temmodaphne
Phát sinh chủng loài
[sửa|sửa mã nguồn]Rohwer (2000:matK) gợi ý rằngHypodaphnis,với bầu nhụy hạ, là chị-em với phần còn lại của họ Lauraceae, kế tiếp làCassytha(nhưng nhánh dài), kế tiếp là [Beilschmeidia+Cryptocarya+Endiandra], kế tiếp làCaryodaphnopsis,kế tiếp là [Chlorocardium+Mezilaurus+Williamodendron], kế tiếp là phần còn lại của Lauraceae;[4]chi tiết hơn, xem Chanderbaliet al.(2001).[5]Như thế ở đây có một lượng các đơn vị phân loại với các nhánh dài, và các phân tích tổ hợp của Rohwer & Rudolph (2005) gợi ý mạnh về sự thay đổi một chút các mối quan hệ này: [Hypodaphnis[[nhómCryptocarya] [Cassytha[[Caryodaphnopsis+Neocinnamomum] [[nhómMezilaurus] [Phần còn lại]]]]]] – phần lớn các nhánh này có xác suất hậu nghiệm 100%.[6]
Mặc dù Hanet al.(2014) thu được một cấu trúc liên kết trong đóHypodaphnislồng trong nhánh chủ yếu bao gồm các thành viên của nhánh 2 và 4 trên đây,[9]nhưng điều này có thể là vấn đề của việc tạo gốc cây phát sinh chủng loài. Massoniet al.(2014) tìm thấy nhánh bao gồmHypodaphnis,CassythavàEusideroxylonlà chị-em với phần còn lại của họ,[10]nhưng các mối quan hệ này được hỗ trợ yếu, trong khi L. Liet al.(2016) tìm thấy rằngCaryodaphnopsisvàNeocinnamomumlà các chi có mối quan hệ chị-em gần nhất củaCassytha,và mặc dù độ hỗ trợ là khá mạnh, nhưng việc lấy mẫu lẽ ra có thể làm tốt hơn: các tác giả đã không đưaHyphodaphnisvào phân tích.[11]Cassythaliên kết yếu với nhómCryptocarya,[Caryodaphnopsis+Neocinnamomum] là nhánh kế tiếp tính từ gốc, nhưng độ hỗ trợ cho vị trí củaCassythalà yếu (Z.-D. Chenet al.2016).[12]Y. Songet al.(2017),[7]sử dụng các trình tự plastome trọn bộ, đã phục hồi một cấu trúc liên kết được hỗ trợ tốt tương tự như cấu trúc liên kết trong Rohwer & Rudolph (2005),[6]mặc dùHyphodaphniskhông được đưa vào, vàCaryodaphnopsisvàNeocinnamomumtạo thành một bậc (grade), chứ không phải một nhánh, trong khi Y. Songet al.(2019),[13]với sự lấy mẫu lớn hơn, đã phục hồi cấu trúc liên kết tương tự và với độ hỗ trợ tốt. Joet al.(2019) phục hồi các mối quan hệ [Cryptocaryeae [Neocinnamomum[Caryodaphnopsis[Perseeae [Cinnamomeae + Laureae]]]]], nhưng với độ hỗ trợ yếu đối với vị trí của 2 chi và độ hỗ trợ mạnh cho vị trí của các tông.[14]
Y. Songet al.(2019) thảo luận về các mối quan hệ trong các tông khác nhau.[13]
- Cryptocaryeae.
Các mối quan hệ bắt đầu được dung giải trong phạm vi nhánhCryptocarya(Rohweret al.2014).[15]Beilschmiedialàcận ngànhtrong hình 3 của Songet al.(2019),[13]nhưng không cận ngành trong hình 2 và cũng không là cận ngành trong B. Liuet al.(2013).[16]
- Caryodaphnopsidae.
Xem L. Liet al.(2016) về các mối quan hệ.[11]
- Laureae.
Litsealàđa ngành,mặc dù tổLitsealà đơn ngành, vàLinderacũng đa ngành (Fijridiyanto & Murakami 2009; Joet al.2019).[14][17]Sự công nhận chiNeolitseadường như làm choActinodaphnetrở thành cận ngành (L. Liet al.2007, đặc biệt xem J. Liet al.2004, 2008).[18][19][20]Songet al.(2019) cũng tìm thấy các chi nhưLitseavàLinderalà đa ngành,[13]xem thêm Joet al.(2019).[14]
- Perseeae.
Về các mối quan hệ trong nhómPersea,xem Rohweret al.(2009) và L. Liet al.(2011);[21][22]PhoebevàPersealà cận/đa ngành.
- Cinnamomeae.
Trong các phân tích bộ gen lạp lục thìCinnamomumlà cận/đa ngành (xem thêm Songet al.2019; Joet al.2019:[13][14]Sassafrasslồng trongCinnamomum,nhưng với độ hỗ trợ thấp). J.-F. Huanget al.(2015) tìm thấy 3 nhánh chính, một chủ yếu là các loài thuộc tổCamphora,là chị em với 2 nhánh còn lại chủ yếu là các loài của tổCinnamomum.[23]Rohdeet al.(2017) tìm thấy rằng các tổ Cựu Thế giớiCinnamomumvàCamphora,cả hai đều là đơn ngành, tạo thành một bậc cận ngành yếu tại đáy của nhánh bao gồm các loàiCinnamomumTân Thế giới, chủ yếu gộp nhóm vớiAiouea,tổ hợpOcotea,bao gồm các đơn vị phân loại từ vài chi khác, là nhóm chị em của nó trong các phân tích ITS, và hai nhóm này cũng được tìm thấy trong các phân tích cpDNA.[24]CinnamomumCựu Thế giới rõ ràng không phải là một phần của một trong hai nhánh này, nhưng các chi tiết về mối quan hệ giữa các tổCinnamomumvàCamphorabất đồng là do và phụ thuộc vào các dấu hiệu được sử dụng (Rohdeet al.2017).[24]Về các mối quan hệ trong khu vựcNectandra(cận ngành)/Ocotea(đa ngành), xem Trofimovet al.(2016, đặc biệt 2019 với 123 trên khoảng 400 loài được kiểm tra), trong nghiên cứu sau thì các mối quan hệ dọc theo trụ chính của cây phát sinh vẫn chỉ được hỗ trợ yếu.[25][26]
Tham khảo
[sửa|sửa mã nguồn] Dữ liệu liên quan tớiLauraceaetạiWikispecies
Dữ liệu liên quan tớiLauraceaetạiWikispecies Tư liệu liên quan tớiLauraceaetạiWikimedia Commons
Tư liệu liên quan tớiLauraceaetạiWikimedia Commons
- ^Angiosperm Phylogeny Group (2009). “An update of the Angiosperm Phylogeny Group classification for the orders and families of flowering plants: APG III”.Botanical Journal of the Linnean Society.161(2): 105–121.doi:10.1111/j.1095-8339.2009.00996.x.
- ^Christenhusz M. J. M.; Byng J. W. (2016).“The number of known plants species in the world and its annual increase”.Phytotaxa.261(3): 201–217.doi:10.11646/phytotaxa.261.3.1.
- ^“Lauraceæ”.
- ^abJens G. Rohwer, 2000.Toward a Phylogenetic Classification of the Lauraceae: Evidence from matK Sequences.Syst. Bot.25(1): 60-71.doi:10.2307/2666673
- ^abAndre S. Chanderbali, Henk van der Werff & Susanne S. Renner, 2001.Phylogeny and Historical Biogeography of Lauraceae: Evidence from the Chloroplast and Nuclear Genomes.Ann. Missouri Bot. Gard.88(1): 104-134doi:10.2307/2666133
- ^abcRohwer J. G. & Rudolph B., 2005.Jumping genera: The phylogenetic positions ofCassytha,Hypodaphnis,andNeocinnamomum(Lauraceae) based on different analyses oftrnK intron sequences.Ann. Missouri Bot. Gard.92: 153-178.
- ^abSong Y., Yu W. B., Tan Y., Liu B., Yao X., Jin J., Padmanaba M., Yang J. B. & Corlett R. T., 2017.Evolutionary comparisons of the chloroplast genome in Lauraceae and insights into loss events in the magnoliids.Genome Biol. Evol.9(9): 2354-2364.doi:10.1093/gbe/evx180
- ^Mo Y. Q., Li L., Li J. W., Rohwer J. G., Li H. W. & Li J., 2017.Alseodaphnopsis:A new genus of Lauraceae based on molecular and morphological evidence.PLoS ONE12(10): e0186545.doi:10.1371/journal.pone.0186545
- ^Han F., Xu L., Peng Y., Ma P., Wang W., Zhang X. & Xiao P., 2014.The pattern of genetic diversity withinLitsea coreana(Lauraceae) in China: An implication for conservation.Plant Syst. Evol.300: 2229-2238.
- ^Massoni J., Forest F. & Sauquet H., 2014.Increased sampling of both genes and taxa improves resolution of phylogenetic relationships within Magnoliidae, a large and early-diverging clade of angiosperms.Molec. Phyl. Evol.70: 84-93.doi:10.1016/j.ympev.2013.09.010
- ^abLi L., Madriñán S. & Li J., 2016.Phylogeny and biogeography ofCaryodaphnopsis(Lauraceae) inferred from low-copy nuclear gene and ITS sequences.Taxon65: 433-443.doi:10.12705/653.1
- ^Chen Z. D., Yan T., Lin L., Lu L. M., Li H. L., Sun M., Liu B., Chen M., Niu Y. T., Ye J. F., Cao Z. Y., Liu H. M., Wang X. M., Wang W., Zhang J. B., Meng Z., Cao W., Li J. H., Wu S. D., Zhao H. L., Liu Z. J., Du Z. Y., Wang Q. F., Guo J., Tan X. X., Su J. X., Zhang L. J., Yang L. L., Liao Y. Y., Li M. H., Zhang G. Q., Chung S. W., Zhang J., Xiang K. L., Li R. Q., Soltis D. E., Soltis P. S., Zhou S. L., Ran J. H., Wang X. Q., Jin X. H., Chen Y. S., Gao T. G., Li J. H., Zhang S. Z., Lu A. M. & China Phylogeny Consortium. 2016.Tree of life for the genera of Chinese vascular plants.J. Syst. Evol.54: 277-306.doi:10.1111/jse.12219
- ^abcdeSong Y., Yu W. B., Tan Y., Jin J., Wang B., Yang J. B., Liu B. & Corlett R. T., 2019.Plastid phylogenomics improve phylogenetic resolution in the Lauraceae.J. Syst. Evol.doi:10.1111/jse.12536
- ^abcdJo S., Kim Y. K., Cheon S. H., Fan Q. & Kim K. J., 2019.Characterization of 20 complete plastomes from the tribe Laureae (Lauraceae) and distribution of small inversions.PLoS ONE14(11):e0224622.doi:10.1371/journal.pone.0224622
- ^Rohwer J. G., de Moraes P. L. R., Rudolph B. & van der Werff H., 2014.A phylogenetic analysis of theCryptocaryagroup (Lauraceae), and relationships ofDahlgrenodendron,Sinopora,Triadodaphne,andYasunia.Phytotaxa158: 111-132.doi:10.11646/phytotaxa.158.2.1
- ^Liu B., Yang Y., Xie L., Zeng G. & Ma K., 2013.Beilschmiedia turbinata:A newly recognized but dying species of Lauraceae from Tropical Asia based on morphological and molecular data.PLoS ONE8(6):e67636.doi:10.1371/journal.pone.0067636
- ^Fijridiyanto, I. A., & Murakami, N. 2009.Phylogeny ofLitseaand related genera (Laureae-Lauraceae) based on analysis ofrpb2 gene sequences.J. Plant Res.122(3): 283-298.
- ^Li L., Li J., Conran J. G. & Li X. W. (Li H. W.), 2007.Phylogeny ofNeolitsea(Lauraceae) inferred from Bayesian analysis of nr DNA ITS and ETS sequences.Plant Syst. Evol.269: 203-221.
- ^Li J., Ledger J., Ward T. & del Tredici P., 2004.Phylogenetics of Calycanthaceae based on molecular and morphological data with a special reference to divergent paralogues of the nrDNA ITS region.Harvard Papers Bot.9(1): 69-82.
- ^Li J., Conran J. G., Christophel D. C., Li Z. M., Li L. & Li H. W., 2008.Phylogenetic relationships in theLitseacomplex and core Laureae (Lauraceae) using ITS and ETS sequences and morphology.Ann. Missouri Bot. Gard.95(4): 580-599.doi:10.3417/2006125.9504
- ^Rohwer J. G., 2009.The timing of nectar secretion in staminal and staminodial glands in Lauraceae.Plant Biol.11: 490-492.doi:10.1111/j.1438-8677.2008.00184.x
- ^Li L., Rohwer J. G., van der Werff H., Wang Z. H. & Li H. W., 2011.Molecular phylogenetic analysis of the Persea group (Lauraceae) and its biogeographic implications on the evolution of tropical and subtropical amphi-Pacific disjunctions.American J. Bot.98(9): 1520-1536.doi:10.3732/ajb.1100006
- ^Huang J. F., Li L., van der Werff H., Li H. W., Rohwer J. G., Crayn D. M., Meng H. H., van der Merwe M., Conran J. G. & Li J., 2016.Origins and evolution of cinnamon and camphor: A phylogenetic and historical biogeographical analysis of theCinnamomumgroup (Lauraceae).Molec. Phyl. Evol.96: 33-44.doi:10.1016/j.ympev.2015.12.007
- ^abRohde R., Rudolph B., Ruthe K., Lorea-Hernández F. G., de Moreas P. L. R., Li J. & Rohwer G., 2017.NeitherPhoebenorCinnamomum- the terasporangiate species ofAiouea(Lauraceae).Taxon66: 1085-1111.doi:10.12705/665.6
- ^Trofimov D., Rudolph B. & Rohwer J. G., 2016.Phylogenetic study of the genusNectandra(Lauraceae), and reinstatement ofDamburneya.Taxon65(5): 980-996.doi:10.12705/655.3
- ^Trofimov D., Moraes P. L. R. D. & Rohwer J. G., 2019.Towards a phylogenetic classification of theOcoteacomplex (Lauraceae): Classification principles and reinstatement ofMespilodaphne.Bot. J. Linn. Soc.190(1): 25-50.doi:10.1093/botlinnean/boz010
Đọc thêm
[sửa|sửa mã nguồn]- Meissner (né Meisner), Carl Daniel Friedrich 1864. Lauraceae (Ordo 162) trong A. L. P. P. de Candolle (ed.),Prodromus Systematis Universalis Regni Vegetabilis15(1): 1-260, Parisiis [Paris], Victoris Masson et Filii.
- Mez, Carl Christian 1889. Lauraceae Americanae Monographice Descripsit.Jahrbuch des Königlichen Botanischen Gartens und des Botanischen Museums zu Berlin5: 1-556.
- Nees von Esenbeck, Christian Gottfried Daniel(1836): Systema Laurinarum, Berlin, Veitii et Sociorum. Cho đến trang 352, có sẵn, tự do, tệp pdf tạiGallica
- Kostermans, André Joseph Guillaume Henri 1957. Lauraceae.Reinwardtia4(2): 193-256
- LauraceaeLưu trữ2007-01-03 tạiWayback MachinetrongL. Watson và M.J. Dallwitz (1992 trở đi). Các họ thực vật có hoa.Lưu trữ2007-01-03 tạiWayback Machine
- Rohwer, Jens G. trong Kubitzki, K.(Chủ biên) 1993. The Families and Genera of Vascular Plants, Quyển 2: K. Kubitzki, J. G. Rohwer & V. Bittrich, 366-390.ISBN 3-540-55509-9
- Wagner, W. L., D. R. Herbst và S. H. Sohmer. 1990.Manual of the Flowering Plants of Hawai'i.Spec. Publ. 83. University of Hawaii Press and Bishop Museum Press. Bishop Museum. 1854 pp.
