Protein

Protein(phát âm theo tiếng Anh:/ˈproʊˌtiːn/,phát âm tiếng Việt:prồ-tê-in haycòn gọi làchất đạm) làphân tử sinh học,hayđại phân tử,gồm nhiềuamino acidliên kết lại với nhau. Protein thực hiện rất nhiều chức năng bên trongtế bào,bao gồm cácphản ứng trao đổi chất có xúc tác,sao chép DNA,đáp ứng lại kích thích,và vận chuyển phân tử từ một vị trí đến vị trí khác. Các protein khác nhau chủ yếu ở trình tự của các amino acid cấu thành (trình tự này lại được quy định bởi trình tựnucleotidecủa cácgenequy định tương ứng) và ở kết quả của giai đoạncuộn gập protein(protein folding) thành nhữngcấu trúc 3 chiềuxác định lên chức năng của nó.
Một mạch thẳng các nhóm amino acid liên kết với nhau gọi là chuỗipolypeptide.Protein chứa ít nhất một chuỗi dài polypeptide. Các chuỗi polypeptide ngắn, chứa ít hơn 20-30 nhóm amin, hiếm khi được coi như là protein và thường được gọi làpeptide,hoặc thỉnh thoảng làoligopeptide.Từng nhóm amino acid được liên kết với nhau bởiliên kết peptide.Trình tự của amino acid trong một protein được xác định bằngtrình tựcủa mộtgenetheobảng mã di truyền.Trong tự nhiên, nhìn chung là có 20 amino acid tham gia tạo nên protein; tuy nhiên, ở một số sinh vật nhất định, mã di truyền của chúng có thể bao gồmselenocysteinevà trong một sốCổ khuẩnlàpyrrolysine.Ngay sau khi tổng hợp hoặc thậm chí trong quá trình tổng hợp, các nhóm amin trong một protein thường bị thay đổi tính chất hóa học bởi quá trìnhsửa đổi sau dịch mã(post-translational modification), làm biến đổi tính chất hóa học và vật lý, sự gập xoắn, tính ổn định, hoạt tính và cuối cùng là chức năng của protein. Một số protein còn có nhóm phi-peptide gắn thêm vào, gọi là nhóm ngoại lai (prosthetic group) hayđồng yếu tố(cofactor). Protein cũng làm việc với nhau để có được một chức năng chuyên biệt, và chúng thường phối hợp để tạo thành dạngphức hệ proteinổn định.
Sau khi được sinh ra, các protein chỉ tồn tại trong một khoảng thời gian nhất định trước khi bịphân giảivà được tái sinh bởi bộ máy của tế bào thông qua quá trìnhquay vòng protein(protein turnover). Vòng đời của một protein được đo bằngchu kỳ bán rãvà nằm trong một khoảng giá trị rất lớn. Thời gian tồn tại của protein có thể có giá trị từ vài phút cho đến hàng năm với thời gian sống trung bình khoảng 1–2 ngày trong tế bàođộng vật.Các protein không bình thường hoặc gập xoắn bị lỗi thường được phân giải nhanh hơn, có thể là do chúng bị đánh dấu để phá hủy hoặc trở nên không ổn định.
Giống như những đại phân tử sinh học khác nhưpolysaccharidevàacid nucleic,protein là thành phần thiết yếu của cơ thể sinh vật và tham gia vào mọi quá trình bên trongtế bào.Nhiều protein là nhữngenzymelàm chấtxúc táccho các phản ứng hóa sinh và cần thiết chotrao đổi chất.Protein cũng có chức năng làm cấu trúc hoặc vận động, nhưactinvàmyosinở cơ và protein trongbộ xương tế bào,tạo nên hệ thống các khung đỡ giúp duy trì hình dáng nhất định của tế bào. Các protein khác tham gia vàotín hiệu tế bào,đáp ứng miễn dịch,kết dính tế bào,vàchu kỳ tế bào.Ở động vật, protein cần thiết phải có trong bữa ăn để cung cấp cácamino acid thiết yếumà không thểtổng hợp.Quá trìnhtiêu hóa"bẻ gãy" các protein để sử dụng trong trao đổi chất.
Protein có thể đượctinh sạchtừ các thành phần khác nhau của tế bào sử dụng nhiều kỹ thuật khác nhau như kỹ thuật siêu ly tâm (ultracentrifugation),kết tủa,điện di,vàsắc ký;sự phát triển củakỹ thuật di truyềnđã đem lại một số phương pháp để tinh sạch protein. Các phương pháp thường gặp để nghiên cứu cấu trúc và chức năng của protein bao gồm kỹ thuậthóa mô miễn dịch(immunohistochemistry), gây đột biến định hướng điểm (site-directed mutagenesis),tinh thể học tia X,cộng hưởng từ hạt nhânvàkhối phổ kế.
| BMP/ĐIỆN GIẢI: | |||
| Na+= 140 | Cl−= 100 | BUN= 20 | / |
| Glu= 150 | |||
| K+= 4 | CO2= 22 | PCr= 1.0 | \ |
| KHÍ MÁU ĐỘNG MẠCH: | |||
| HCO3-= 24 | paCO2= 40 | paO2= 95 | pH= 7.40 |
| THÔNG KHÍ PHẾ NANG: | |||
| pACO2= 36 | pAO2= 105 | A-a g= 10 | |
| KHÁC: | |||
| Ca= 9.5 | Mg2+= 2.0 | PO4= 1 | |
| CK= 55 | BE= −0.36 | AG= 16 | |
| ĐỘ THẨM THẤU HUYẾT TƯƠNG/THẬN: | |||
| PMO= 300 | PCO= 295 | POG= 5 | BUN:Cr= 20 |
| XÉT NGHIỆM NƯỚC TIỂU: | |||
| UNa+= 80 | UCl−= 100 | UAG= 5 | FENa= 0.95 |
| UK+= 25 | USG= 1.01 | UCr= 60 | UO= 800 |
| PROTEIN/ĐƯỜNG RUỘT/XÉT NGHIỆM CHỨC NĂNG GAN: | |||
| LDH= 100 | TP= 7.6 | AST= 25 | TBIL= 0.7 |
| ALP= 71 | Alb= 4.0 | ALT= 40 | BC= 0.5 |
| AST/ALT= 0.6 | BU= 0.2 | ||
| AF alb= 3.0 | SAAG= 1.0 | SOG= 60 | |
| DỊCH NÃO TỦY: | |||
| CSF alb= 30 | CSF glu= 60 | CSF/S alb= 7.5 | CSF/S glu= 0.4 |
Hóa sinh[sửa|sửa mã nguồn]

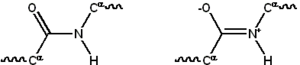
Hầu hết các protein đều chứa một hoặc nhiều chuỗipolymemạch thẳng cấu thành từ tập hợp 20L-α-amino acidkhác nhau. Các amino acid cấu tạo nên protein (amino acid sinh protein) có những đặc điểm cấu trúc giống nhau: đều có một α-carbon mà tại đó một nhómamin,một nhómcarboxyl,và nhiều loạinhóm bên(side chain) khác nhau có thể liên kết vào. Chỉ cóprolinelà khác với cấu trúc cơ bản này khi nó chứa một vòng tại điểm N-kết thúc của nhóm amin, khiến cho nửa nhóm CO–NH có hình dáng cố định là một mặt phẳng.[1]Nhóm bên của cácamino acid cơ sởcó tính chất và cấu trúc hóa học rất đa dạng; chính sự kết hợp và tương tác giữa các nhóm bên amino acid trong protein đã xác định cấu trúc 3 chiều và đặc tính phản ứng hóa học của protein.[2]
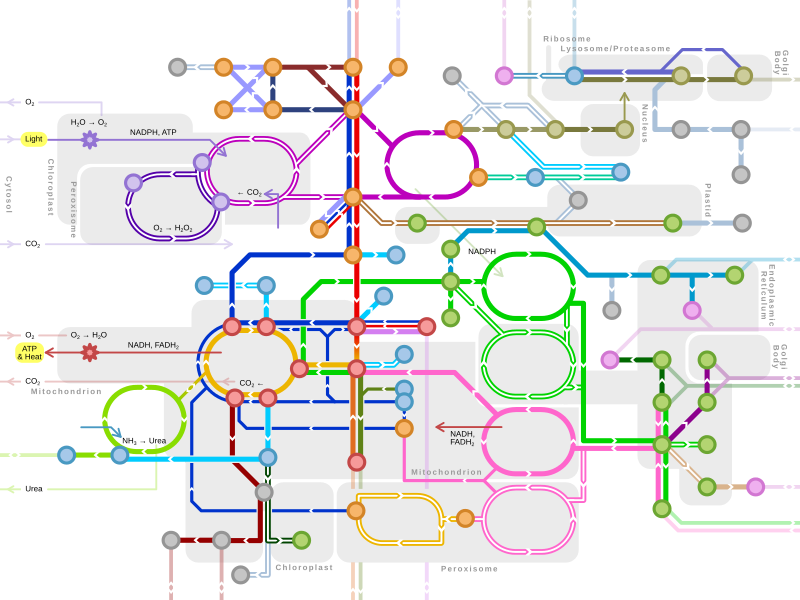
Amino acid trong một chuỗi polypeptide được liên kết với nhau bằng liên kết peptide. Khi được liên kết trong chuỗi protein, từng amino acid được gọi là phần thừa (hay phần dư,residue), và cấu trúc liên kết một loạt các nguyên tử carbon, nitro, và oxy được gọi làmạch chínhhaybộ khung protein.[3]
Liên kết peptide có hai dạng cộng hưởng (resonance,hay cấu trúcmesome) góp phần tạo nên một số đặc trưngliên kết đôivà làm cản trở sự quay xung quanh trục của nó, vì vậy mà các nguyên tử carbon Alpha hầu như là đồng phẳng với nhau. Haigóc nhị diệnkhác trong liên kết peptide xác định hình dạng cục bộ đảm nhiệm bởi khung xương protein.[4]Điểm kết thúc của protein với một nhóm carboxyl tự do được gọi làđiểm kết thúc-Choặc đầu mút cacboxy, trong khi điểm kết thúc với một nhóm amin tự do được gọi làđiểm kết thúc-Nhoặc đầu mút amin. Các thuật ngữprotein,polypeptide,vàpeptidecó một chút khó hiểu và có thể mang ý nghĩa chồng lặp.Proteinnói chung được sử dụng để nhắc đến những phân tử sinh học hoàn thiện trongcấu hìnhổn định, trong khipeptidethường chỉ một oligome amino acid ngắn mà không có cấu trúc ba chiều ổn định. Tuy vậy, ranh giới giữa hai định nghĩa này thường không xác định rõ ràng và thường là peptide dài khoảng 20–30 amino acid.[5]Polypeptidethường muốn đề cập tới bất kỳ một mạch thẳng nào tạo nên từ amino acid, bất kểchiều dài,và thường hàm ý sự vắng mặt của một cấu hình xác định.
Sự xuất hiện trong tế bào[sửa|sửa mã nguồn]
Các nhà sinh học ước tính mộtvi khuẩnkích thước trung bình chứa khoảng 2 triệu protein trong tế bào của nó (ví dụ nhưE. colivàStaphylococcus aureus). Các vi khuẩn nhỏ hơn, nhưMycoplasmahayspirochetessẽ chứa ít phân tử hơn, vào cỡ 50.000 đến 1 triệu phân tử protein. Ngược lại, các tế bàonhân thựccó kích thước lớn hơn và do vậy chứa nhiều protein hơn. Ví dụ, tế bàonấm menước tính có khoảng 50 triệu protein và tế bàongườicó từ 1 đến 3 tỷ protein.Bộ genecủa vi khuẩn mã hóa cho protein thấp hơn 10 lần so với của người (ví dụ vi khuẩn nhỏ ~1.000, E. coli: ~4.000, nấm men: ~6.000, loài người: ~20.000).[6]
Nồng độ của các protein trong một tế bào có một phổ giá trị rất rộng, từ chỉ một vài phân tử cho đến hàng trăm nghìn phân tử trong một tế bào. Khoảng một phần ba tổng số protein không được sản sinh ra trong tế bào hay chỉ sinh ra trong những điều kiện nhất định. Ví dụ, trong số 20.000 protein được mã hóa bởi bộ gene ở loài người chỉ có 6.000 được phát hiện trongnguyên bào lympho.[7]Hơn nữa, số lượng protein màbộ genemã hóa có mối tương quan với cấu trúc phức tạp của cơ thể vật chủ. Sinh vật nhân thật, vi khuẩn,vi khuẩn cổvàvi rúttương ứng có trung bình 15145, 3200, 2358 và 42 protein được mã hóa trong bộ gene của chúng.[8]
Tổng hợp[sửa|sửa mã nguồn]
Sinh tổng hợp[sửa|sửa mã nguồn]


Protein cấu tạo từ những amino acid lắp ghép lại sử dụng thông tin được mã hóa tronggene.Mỗi protein có trình tự amino acid duy nhất xác định bởi trình tự cácnucleotidetrong gene mã hóa cho protein này.Mã di truyềnlà một tập hợp chứa các tập hợp con của các bộ ba-nucleotide gọi là bộ ba mã hóa (codon) và mỗi tổ hợp ba-nucleotide tương ứng cho một amino acid, ví dụ AUG (adenine-uracil-guanine) mã hóa chomethionine.Bởi vìDNAchứa bốn nucleotide, tổng số codon khả dĩ là 64; tuy nhiên chỉ có 20 amino acid nên một số amino acid được mã hóa bởi nhiều hơn một codon.[9]Gen mã hóa trong DNA trước tiên đượcphiên mãthành phân tử tiền-mRNA (pre-mRNA) bởi các protein nhưRNA polymerase.Hầu hết các sinh vật sau đó xử lý tiền-mRNA (haysản phẩm phiên mã sơ cấp-primary transcript) với nhiều dạng của sửa đổi sau phiên mã (post-transcriptional modification) để tạo nên phân tử mRNA hoàn chỉnh (mature mRNA), làm khuôn mẫu cho sinh tổng hợp protein nhờribosome.Ởsinh vật nhân sơ,mRNA hoặc là được sử dụng ngay sau khi nó hình thành, hoăc được gắn với một ribosome sau khi rời khỏivùng nhân.Ngược lại, ởsinh vật nhân thực,mRNA được sinh ra trongnhân tế bàorồi sau đó chuyển dịch quamàng nhânđếnbào tương,nơi quá trìnhsinh tổng hợp proteindiễn ra. Tốc độ tổng hợp protein ở sinh vật nhân sơ nhanh hơn so với sinh vật nhân thực và có thể đạt tới 20 amino acid trong một giây.[10]
Giai đoạn tổng hợp protein từ khuôn mRNA gọi làdịch mã.mRNA được đưa vào ribosome và ribosome một lần đọc ba nucleotide bằng cách khớp theo nguyên tắc bổ sung mỗi bộ ba mã hóa (codon) với một bộ ba đối mã (anticodon) nằm trên phân tửRNA vận chuyển,nó mang theo amino acid tương ứng với codon mà nó nhận ra. Trước đó, enzymeaminoacyl tRNA synthetase"nạp" một amino acid đúng vào phân tử tRNA. Chuỗi polypeptide đang hình thành thường được gọi làchuỗi mới sinh(nascent chain). Protein luôn luôn sinh tổng hợp theo chiều từ đầu N (N-terminus,đầu có nhóm NH2) đến đầu C (C-terminus,đầu có nhóm COOH).[9]
Kích thước của một protein sinh tổng hợp có thể đo bằng số lượng amino acid chứa trong nó hoặc bằng tổngkhối lượng phân tử,mà thông thường tính bằng đơn vịdalton(đồng nghĩa vớiđơn vị khối lượng nguyên tử), hoặc đơn vị phái sinh kilodalton (kDa). Protein sinh tổng hợp từnấm mentrung bình dài 466 amino acid và có khối lượng 53 kDa.[5]Protein lớn nhất từng được biết đến làtitin,một thành phần của đơn vị cơ bản sợicơ vân(muscle sarcomere), với khối lượng phân tử 3.000 kDa và chứa tới 27.000 amino acid.[11]
Hóa tổng hợp[sửa|sửa mã nguồn]
Các protein ngắn có thể được tổng hợp hóa học bằng một số phương pháp gọi làtổng hợp peptide,mà dựa trên những kỹ thuậttổng hợp hữu cơnhư kỹ thuật nối hóa học (chemical ligation) để tạo ra các peptide vớichiều dàilớn.[12]Tổng hợp hóa học cho phép đưa những amino acid nhân tạo vào trong chuỗi polypeptide, như gắn các phân tử thăm dòhuỳnh quangvào mạch bên của chuỗi amino acid.[13]Những phương pháp này hữu hiệu trong phòng thí nghiệmhóa sinhvàsinh học tế bào,mặc dù nói chung không thương mại hóa được. Tổng hợp hóa học sẽ trở nên không hiệu quả đối với chuỗi polypeptide dài hơn khoảng 300 amino acid, và các protein được tổng hợp có thể không gập về dạng cấu trúc bậc bốn như mong đợi. Hầu hết các phương pháp tổng hợp hóa học bắt đầu từ đầu C đến đầu N, trái ngược so với các phản ứng sinh học.[14]
Protein[sửa|sửa mã nguồn]


Hầu hết các proteincuộn gấpthành một cấu trúc ba chiều duy nhất. Hình dạng mà một protein khi để uốn gấp một cách tự nhiên được gọi là hình dạng nguyên sinh (native conformation).[15]Mặc dù nhiều protein có thể uốn gấp mà không cần hỗ trợ, chỉ đơn giản nhờ vào các đặc tính hóa học của các amino acid thành phần, những protein khác đòi hỏi sự hỗ trợ của phân tửchaperoneđể uốn gấp thành hình dạng nguyên sinh của chúng.[16]Các nhà hóa sinh phân ra bốn cấp đối với cấu trúc của protein:[17]
- Cấu trúc sơ cấphaycấu trúc bậc 1:Là trình tự sắp xếp các gốc amino acid trong chuỗi polypeptide. Cấu trúc này được giữ vững nhờliên kết peptide(liên kết cộng hóa trị). Một protein làpolyamide(poliamit).
- Cấu trúc bậc 2:Là tương tác không gian giữa các gốc amino acid ở gần nhau trong chuỗi polypeptide. Cấu trúc được bền vững chủ yếu nhờliên kết hiđrôhình thành giữa các liên kết peptide ở kề gần nhau, cách nhau những khoảng xác định. Các ví dụ cho cấu trúc bậc 2 của phân tử protein làxoắn α(α-helix),phiến gấp nếp β(β-sheet) và cácvùng chuyển hướng.Bởi vì cấu trúc bậc 2 mang tính cục bộ, nhiều vùng với các cấu trúc bậc 2 khác nhau có thể tồn tại trong cùng một phân tử protein.
- Cấu trúc bậc 3:hình dạng tổng thể của một phân tử protein đơn nhất; hay mối quan hệ không gian giữa các cấu trúc bậc 2 với nhau. Nói chung cấu trúc bậc 3 được giữ ổn định bởi các tương tác phi cục bộ, phần lớn bởi sự hình thành mộtlõi kị nước(hydrophobic core), và ngoài ra giữ bởi các cầu muối (salt bridge), liên kết hiđrô,liên kết disulfide,và thậm chí là các chỉnh sửa sau dịch mã (post-translational modification). Thuật ngữ "cấu trúc bậc 3" thường được sử dụng mang nội dung đồng nghĩa với thuật ngữuốn gấp.Cấu trúc bậc 3 kiểm soát chức năng cơ bản của protein.
- Cấu trúc bậc 4:cấu trúc hình thành bởi một số phân tử protein liên kết với nhau (chuỗi polypeptide), mà hay gặp thuật ngữtiểu đơn vị proteintrong trường hợp này, mà chức năng của cấu trúc bậc 4 hoạt động như mộtphức hợp protein.
Protein không phải là một phân tử "cứng chắc" hoàn toàn. Không chỉ cố định ở một bậc cấu trúc nhất định, protein có thể chuyển sang một vài cấu trúc liên quan khi chúng thực hiện các chức năng sinh học. Trong trường hợp của những sự sắp xếp các chức năng này, các cấu trúc bậc 3 và bậc 4 thường được gọi là "cấu dạng",và sự chuyển tiếp giữa chúng gọi làsự thay đổi cấu dạng.Những thay đổi này thường do sự liên kết của một phân tử cơ chất (substrate molecule) với mộtvị trí hoạt độngcủa mộtenzyme,những vùng của protein tham gia vào xúc tác hóa học. Các protein trong dung dịch hòa tan cũng trải qua những biến đổi về cấu trúc tác động bởi các rung động nhiệt và sự va chạm với các phân tử khác.[18]

Toàn bộ protein hoặc những đoạn protein được phân loại thành bốn lớp chính, mà tương quan với cấu trúc bậc 4 của nó: protein dạng cầu (globular protein), protein dạng sợi (fibrous protein), protein màng tích hợp (integral membrane protein) và protein mất trật tự nội tại (intrinsically disordered protein).[19]Phần lớn toàn bộ protein dạng cầucó thể tan đượcvà đa phần là các enzyme. Protein dạng sợi thường có vai trò cấu trúc, nhưcollagene,thành phần chính của các mô liên kết, haykeratin,thành phần protein của tóc và móng chân tay. Protein dạng màng thường phục vụ như là nhữngthụ thểhoặc làm kênh dẫn cho các phân tử mang điện tích hay phân cực vượt quamàng tế bào.[20]Protein mất trật tự nội tại khác biệt cơ bản về tính trật tự về cấu hình hình dạng với ba loại trên. Nhiều protein có hình dạng xác định rõ ràng khi ở dạng nguyên thể (native), nhưng protein mất trật tự nội tại thì không, chuỗi polypeptide của chúng rất linh động và không có một hình dáng nhất định. Tính chất này cho phép protein mất trật tự nội tại có thể tương tác với nhiều protein đối tác hoặc gập thành những hình dáng nhất định chỉ khi nó liên kết với những đối tác này. Protein mất trật tư nội tại thường là các phân tử truyền tín hiệu, điều hòa hoạt động cho những phân tử khác, hoặc làm bộ khung cho các protein khác bám vào.[19]
Một trường hợp đặc biệt của liên kết hiđrô liên phân tử bên trong protein, chỉ che chắn yếu ớt từ ảnh hưởng của nước và do vậy tự chúng dễ bịkhử nước,được gọi làdehydron.[21]
Xác định cấu trúc[sửa|sửa mã nguồn]
Khám phá ra cấu trúc bậc ba của protein, hay cấu trúc bậc bốn của phức hợp protein, sẽ mang lại những chứng cứ quan trọng về chức năng của protein. Các phương pháp thực nghiệm phổ biến xác định cấu bao gồmtinh thể học tia Xvàkỹ thuật phổ NMR,cả hai đều cho thông tin ở mức phân giải cấpnguyên tử.Tuy nhiên, kỹ thuật NMR có thể cung cấp thông tin ước lượng về khoảng cách giữa từng cặp nguyên tử, và hình dạng khả dĩ cuối cùng đối với protein được xác định thông qua giảibài toán hình học khoảng cách.Kỹ thuật giao thoa phân cực hai sóng dẫn (Dual polarisation interferometry) là một phương pháp giải tích định lượng cho phép đo hình dạng tổng thể của protein và những thay đổi hình dạng do tương tác giữa các nguyên tử hoặc bởi các tác động khác. Lưỡng hướng sắc phân cực tròn (circular dichroism) là một kỹ thuật trong phòng thí nghiệm giúp xác định các thành phần cấu trúc bậc hai gấp nếp β / xoắn α của protein.Kính hiển vi điện tử truyền quaquan sát mẫu lạnh (cryoelectron microscopy) được sử dụng để thu được thông tin về cấu trúc có độ phân giải thấp hơn của các phức hợp protein rất lớn, bao gồm tổ hợp cácvirus;[22]một kỹ thuật biến thể khác gọi là "xác định cấu trúc tinh thể bằng kính hiển vi điện tử truyền qua" (electron crystallography) có thể thu được độ phân giải lớn ở một số trường hợp, đặc biệt đối với những tinh thể hai chiều ở các protein dạng màng.[23]Thông tin về những cấu trúc quan sát được thường lưu trữ ởNgân hàng Dữ liệu Protein(Protein Data Bank,PDB), một hệ thống nguồn truy cập tự do mà dữ liệu cấu trúc của hàng nghìn protein có thể nhận được dưới dạngtọa độ Descartescho từng nguyên tử trong protein.[24]
Có thêm nhiều trình tự gene được giải mã hơn so với số lượng protein có cấu trúc được làm rõ. Ngoài ra, tập hợp những cấu trúc được quan sát bị chệch khỏi hình dạng nguyên sinh của protein do tác động từ những điều kiện quan sát đòi hỏi trong kỹ thuật tinh thể học tia X, một trong những phương pháp chính nhằm xác định cấu trúc protein. Đặc biệt, những protein dạng cầu thường dễ dàng chuẩn bị làm mẫu tinh thể hóa trước khi tiến hành kỹ thuật chụp tinh thể học tia X. Ngược lại, các protein dạng màng, lại rất khó để đưa về dạng tinh thể và chưa có dữ liệu về chúng trong PDB.[25]Hướng nghiên cứu "bộ gene mã hóa cấu trúc protein" (structural geneomics) được khởi xướng gần đây đã cố gắng giảm bớt những lỗ hổng hiểu biết này bằng cách giải quyết một cách hệ thống cho cấu trúc của một số lớp protein thông qua cách chúng uốn gập. Phương pháp dự đoán cấu trúc protein có mục đích mang lại một bức tranh sơ bộ về cấu trúc của những protein mà chúng chưa được xác định cấu trúc bằng thực nghiệm.[26]
Chức năng tế bào[sửa|sửa mã nguồn]
Protein là diễn viên chính bên trong tế bào, thực hiện các nhiệm vụ xác định bởi thông tin mã hóa trong gene.[5]Ngoại trừ đối với một số loạiRNAnhất định, hầu hết các phân tử sinh học khác là những phân tử tương đối trơ với tác dụng của protein. Protein chiếm một nửa trọng lượng khô của tế bào vi khuẩnEscherichia coli,trong khi những đại phân tử khác như DNA và RNA chỉ chiếm tương ứng 3% và 20%.[27]Tập hợp các protein biểu hiện trong một tế bào cụ thể hoặc một loại tế bào được gọi là hệ protein (proteome) hay bộ protein hoàn chỉnh.

Đặc trưng chính của protein mà cũng làm lên các chức năng đa dạng đó là khả năng của chúng liên kết một cách đặc hiệu và chặt với các phân tử khác. Vùng protein có tính năng liên kết với các phân tử khác được gọi là vùng liên kết (binding site) và thường là những khe rãnh (depression) hoặc "túi" ("pocket") trên bề mặt phân từ. Khả năng liên kết này được thực hiện trung gian thông qua bởi cấu trúc bậc ba của protein, mà xác định vị trí túi liên kết, và bởi các tính chất hóa học của các chuỗi nhánh bên amino acid xung quanh. Liên kết protein có thể rất đặc hiệu và cực kỳ chặt; ví dụ,protein ức chế ribonuclease(ribonuclease inhibitor protein) liên kết với proteinangiogeneinở người vớihằng số phân lycỡ dưới femto mol (<10−15M) nhưng không liên kết với proteinonconasetương đồng ở động vật lưỡng cư (>1 M). Những sự thay đổi hóa học rất nhỏ như thêm vào một nhóm methyl ở phân tử liên kết đôi khi đủ làm gần như loại bỏ liên kết với protein; ví dụ,aminoacyl tRNA synthetaseđặc hiệu với amino acidvalinelại rất phân biệt vớiisoleucinemặc dù hai amino acid này có chuỗi bên rất tương đồng.[28]
Protein có thể liên kết với các protein khác cũng như với các cơ chấttiểu phân tử(small-molecule substrate). Khi protein liên kết đặc hiệu với những bản sao khác của cùng phân tử, chúng có thểoligomehóa để tạo thành những sợi nhỏ; quá trình này thường xuất hiện ở những protein cấu trúc mà chứa những monome dạng cầu mà tự tổ chức thành những sợi vững chắc.Tương tác protein–proteincũng điều hòa các hoạt động do enzyme, điều khiển xúc tiến toàn bộchu kỳ tế bào,và cho phép lắp ghép nhữngphức hợp proteinlớn mà chúng thực hiện những phản ứng liên quan mật thiết với nhau với một chức năng sinh học chung. Protein cũng có thể liên kết với, hay thậm chí tích hợp vào màng tế bào. Khả năng liên kết với các đối tác để cảm ứng sự thay đổi hình dáng trong các protein cho phép xây dựng lên một mạng lướitín hiệu tế bàorộng lớn và phức tạp.[29]Do tương tác giữa các protein là thuận nghịch, và phụ thuộc nhiều vào khả năng của các nhóm protein khác nhau để hình thành lên tổ hợp có khả năng thực hiện các chức năng riêng rẽ, lĩnh vực nghiên cứu tương tác giữa các protein đặc hiệu là chìa khóa nhằm hiểu biết những khía cạnh quan trọng của chức năng tế bào, và đi đến những tính chất giúp phân biệt giữa các loại tế bào đặc biệt.[30][31]
| Loại protein | Chức năng | Ví dụ |
|---|---|---|
| Cấu trúc | Cấu trúc, nâng đỡ | Collagenevàelastintạo nên cấu trúc sợi rất bền củamô liên kết,dây chằng,gân.Keratintạo nên cấu trúc chắc của da, lông, móng. Protein tơ nhện, tơ tằm tạo nên độ bền vững của tơ nhện, vỏ kén. |
| Enzyme | Xúc tác sinh học:tăng tốc độ phản ứng, chọn lọc các phản ứng sinh hóa | Cácenzymethủy phân trong dạ dày phân giải thức ăn, enzymeamylasetrong nước bọt phân giảitinh bộtchín, enzymepepsinphân giải protein, enzymelipasephân giảilipid. |
| Hormone | Điều hòa các hoạt động sinh lý | Hormoneinsulinvàglucagondotế bào đảo tụy(beta cell) thuộctuyến tụytiết ra có tác dụng điều hòa hàm lượng đườngglucosetrong máu động vật có xương sống. |
| Vận chuyển | Vận chuyển các chất | Huyết sắc tốhemoglobincó chứa trong hồng cầu động vật có xương sống có vai trò vận chuyểnoxytừphổitheomáuđi nuôi các tế bào. |
| Vận động | Tham gia vào chức năng vận động của tế bào và cơ thể | Actinin,myosincó vai trò vận động cơ.Tubulincó vai trò vận động lông, roi của các sinh vật đơn bào. |
| Bảo vệ | Bảo vệ cơ thể chống bệnh tật | Interferonchốngvirus.Kháng thể chốngvi khuẩngây bệnh. |
| Thụ quan | Cảm nhận, truyền tín hiệu, đáp ứng các kích thích của môi trường | Thụ quan màngcủa tế bào thần kinh khác tiết ra (chất trung gian thần kinh) và truyền tín hiệu. |
| Dự trữ | Dự trữ chất dinh dưỡng | Albuminlòng trắngtrứnglà nguồn cung cấpamino acidchophôiphát triển.Caseintrong sữa mẹ là nguồn cung cấp amino acid chothai nhi.Trong hạt cây có chứa nguồn protein dự trữ cần thiết cho hạt nảy mầm. |
Enzyme[sửa|sửa mã nguồn]
Vai trò được biết đến nhiều nhất của protein trong tế bào như là cácenzyme,khi chúng là yếu tố xúc tác cho các phản ứng sinh hóa. Enzyme có tính đặc hiệu cao và chỉ tham gia vào một hoặc một vài phản ứng hóa học. Enzyme tham gia nhiều nhất vào các phản ứng trongtrao đổi chất,cũng như tác động vào DNA trong những quá trình nhưnhân đôi DNA,sửa chữa DNA,vàphiên mã.Một số enzyme tác động lên những protein khác để gắn thêm vào hoặc loại bỏ nhóm chức hóa học trong quá trình gọi sửa đổi sau dịch mã (post-translational modification). Có khoảng 4.000 phản ứng sinh hóa đã biết được xúc tác bởi enzyme.[34]Sự gia tăng tốc độ phản ứng nhờ xúc tác có enzyme thường là rất lớn—tăng tới 1017lần trong phản ứng mà không có xúc tác như trong trường hợp củaorotate decarboxylase(xảy ra trong 78 triệu năm mà không có enzyme, 18 milli giây với enzyme).[35]
Các phân tử liên kết vào và bị tác động bởi enzyme được gọi là cáccơ chất(substrate). Mặc dù enzyme có thể chứa hàng trăm amino acid, thường chỉ có một số nhỏ các nhóm dư (residues) trên nó là tham gia tiếp xúc với cơ chất, và thậm chí một số nhỏ hơn—trung bình từ 3 đến 4 nhóm dư—là tham gia trực tiếp vào xúc tác.[36]Vùng của enzyme liên kết với cơ chất và chứa nhóm dư xúc tác được gọi là vị trí hoạt động (active site).
Dirigenet protein là những phần tử trong một lớp các protein chi phốihóa học lập thể(stereochemistry) của một hợp chất được tổng hợp bởi những enzyme khác.[37]
Tín hiệu tế bào và liên kết phối tử[sửa|sửa mã nguồn]
Nhiều protein tham gia vào các giai đoạn của quá trình truyềntín hiệu tế bàovàtải nạp tín hiệu.Một số protein, nhưinsulin,là những protein ngoại bào thực hiện truyền tín hiệu từ tế bào mà chúng được sinh tổng hợp đến những tế bào khác trongmôở xa. Những protein khác làprotein màng(membrane protein) hoạt động như là nhữngthụ thểmà chức năng chính là liên kết với một phân tử tín hiệu và cảm ứng một đáp ứng hóa sinh bên trong tế bào. Nhiều thụ thể có vị trí liên kết nằm bên trên bề mặt tế bào và miền tác dụng nằm bên trong tế bào, mà hoạt động chức năng enzyme có thể trải qua một sự thay đổi cấu dạng (conformational change) được phát hiện bởi những protein khác bên trong tế bào.[38]
Kháng thểlà những thành phần protein của mộthệ miễn dịch thu được(adaptive immune system) có chức năng chính là liên kết với cáckháng nguyên,hoặc những cơ chất lạ bên trong tế bào của cơ thể, và nhận diện đánh dấu chúng để tiêu hủy. Kháng thể có thể tiết vào môi trường ngoại bào hoặc bám vào màng của nhữngtế bào Bchuyên biệt (B cell) gọi làtế bào plasma.Trong khi các enzyme bị giới hạn ở ái lực liên kết với các chất nền bởi tính cần thiết cho việc điều khiển phản ứng mà chúng tham gia, các kháng thể lại không bị giới hạn này. Ái lực liên kết của các kháng thể với mục tiêu của nó là cực kỳ cao.[39]
Nhiềuphối tử(ligand) vận chuyển các protein gắn đặc hiệu cùng với các phân tử sinh học nhỏ và vận chuyển chúng đến những vị trí khác nhau trong cơ thể của một sinh vật đa bào. Những protein này phải có ái lực liên kết lớn khi các phối tử có mặt ở mức độ tập trung lớn, nhưng cũng giải phóng được phối tử khi sự có mặt của chúng ở mức độ thấp tại những mô đích đến. Ví dụ điển hình của protein liên kết phối tử làhaemoglobin,giúp vận chuyểnoxytừphổiđến các mô và các cơ quan khác ởđộng vật có xương sốngvà có sựtương đồnggần gũi trong mọigiới sinh học.[40]Lectinlà những protein liên kết với đường có chức năng đặc hiệu cao với phân tử đường của nó. Lectin đóng vai trò điển hình trong hiệu ứng nhận dạng phân tử ở tế bào và các protein.[41]Các thụ thể vàhormonelà những protein liên kết đặc hiệu cao.
Protein xuyên màng(transmembrane protein) cũng được coi như những protein chuyên chở phối tử mà làm thay đổi tính thấm của màng tế bào đối với những phân tử nhỏ và ion. Riêng ở màng có một lõi kị nước mà các phân tử phân cực hay mang điện không thểkhuếch tánqua nó. Protein màng chứa các kênh bên trong cho phép các phân tử như vậy đi vào và thoát ra khỏi tế bào. Nhiều proteinkênh ionlà chuyên biệt cho phép chỉ một ion đặc biệt đi qua; ví dụ, các kênhkalivànatrichỉ cho một loại ion tương ứng đi qua.[42]
Protein cấu trúc[sửa|sửa mã nguồn]

Các protein cấu trúc đem lại tính vững trãi và sự cứng chắc cho các thành phần sinh học không ở trạng thái lỏng khác. Hầu hết các protein cấu trúc là nhữngprotein dạng sợi;ví dụ,collagenvàelastinlà những thành phần quan trọng củamô liên kếtnhưsụn,vàkeratinđược tìm thấy trong các cấu trúc cứng hoặc có dạng sợi nhưlông,móng,lông vũ,móng guốc,vàvỏ giáp ngoài.[43]Một sốprotein dạng cầucũng đóng vai trò làm chức năng sinh học, ví dụ,sợi actinvàtubulincó dạng cầu và hòa tan được khi là các monome, nhưng khi bịpolymehóa tạo thành dạng sợi dài, cứng giúp cấu thành lênbộ xương tế bào,cho phép tế bào duy trì hình dạng và kích thước của nó.
Những protein khác phục vụ chức năng cấu trúc làprotein động cơnhưmyosin,kinesin,vàdynein,mà chúng có khả năng sinh ra lực cơ học. Những protein này đặc biệt quan trọng cho sự di chuyển (motility) của tế bào ở những sinh vật đơn bào và củatinh trùngở phần lớn sinh vật đa bào cho hoạt độngsinh sản.Chúng cũng sinh ra lực đẩy làmcơco lại[44]và đóng vai trò quan trọng ở quá trình vận chuyển bên trong tế bào.
Phương pháp nghiên cứu[sửa|sửa mã nguồn]
Các hoạt động và chức năng của protein có thể nghiên cứu trongống nghiệm(in vitro),in vivo,vàin silico.Phương phápin vitronghiên cứu các protein được sàng lọc trong những môi trường có kiểm soát giúp tìm hiểu một protein thực hiện chức năng của nó như thế nào: ví dụ, lĩnh vực nghiên cứuđộng học enzyme(enzyme kinetic) khám phácơ chế phản ứngcủa sự hoạt động xúc tác của một enzyme và ái lực của nó đối với nhiều phân tử cơ chất khác nhau. Ngược lại, phương pháp thực nghiệmin vivocung cấp thông tin về vai trò sinh lý của một protein bên trongtế bàohay thậm chí toàn bộsinh vật.Phương phápin silicosử dụng các phương pháp của tin sinh học để nghiên cứu protein.
Tinh sạch protein[sửa|sửa mã nguồn]

Để thực hiện phân tíchin vitro,một protein cần nghiên cứu phải được tinh sạch và sàng lọc (protein purification) khỏi những thành phần khác của tế bào. Quá trình này thường bắt đầu bằng cách phá tế bào (hay tiêu tế bào,cytolysis), khi ấy màng tế bào bị phá vỡ khi lượng nướcthẩm thấuquá nhiều vào trong tế bào và các thành phần bên trong được giải phóng vào một dung môi gọi là dung dịch thủy phân tế bào (crude lysate,haycytolysate). Hỗn hợp thu được được tinh sạch bằng phương pháp siêu ly tâm (ultracentrifugation), mà phân tách nhiều thành phần tế bào thành các phần chứa các protein hòa tan khác nhau; như mànglipidvà protein;bào quantế bào, vàacid nucleic.Hỗn hợp đượckết tinhbằng phương pháp tách tinh thể muối (salting out) cho phép tập trung protein từ dung dịch này. Sau đó sử dụng nhiều kỹ thuậtsắc kýđể cô lập một hoặc một vài protein cần nghiên cứu dựa trên những tính chất của chúng như trọng lượng phân tử, tổng điện tích và ái lực liên kết.[45]Mức độ sàng lọc được giám sát nhờ sử dụng nhiều kỹ thuậtđiện di trên gel(gel electrophoresis) nếu biết trọng lượng phân tử vàđiểm đẳng điện(isoelectric point) của protein cần nghiên cứu, hoặc bằng phân tíchphổnếu protein có những đặc trưng phổ dễ phân biệt, hoặc bằng thí nghiệm thử enzyme (enzyme assay) nếu protein có hoạt tính enzyme. Thêm vào đó, protein có thể được cô lập theo điện tích của chúng nhờ sử dụng phương pháp tập trung đẳng điện (isoelectric focusing).[46]
Đối với các protein tự nhiên, cần phải thực hiện một chuỗi các bước tinh sạch trước khi có thể thu được một lượng protein đủ thuần khiết cho mục đích sử dụng trong phòng thí nghiệm. Để làm đơn giản quá trình này, các nhà hóa sinh thường sử dụngkỹ thuật di truyềnđể thêm vào các đặc điểm cho protein giúp dễ dàng sàng lọc chúng hơn mà không làm ảnh hưởng đến cấu trúc hay hoạt động của chúng. Ở đây, một "chất đánh dấu" (tag) chứa một trình tự amino acid đặc hiệu, thường là một chuỗihistidine(chất "His-tag"), được gắn vào một đầu của protein. Kết quả là, khi đưa dung dịch hòa tan protein vào các ống nghiệm của máy sắc ký chứaniken,histidine liên kết phối tử với niken và đọng lại trong cột trong khi những thành phần không được đánh dấu trong dung dịch sẽ chảy qua không bị cản trở. Nhiều phương pháp đánh dấu đã được phát triển để giúp các nhà nghiên cứu sàng lọc các protein đặc biệt từ những hợp chất phức tạp.[47]
Khu trú tế bào[sửa|sửa mã nguồn]
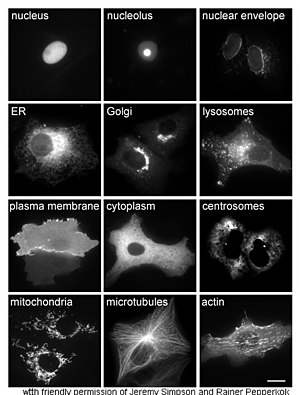
Phương pháp nghiên cứuin vivocho protein thường đề cập đến sự tổng hợp và sự định vị (khu trú,localization) protein bên trong tế bào. Mặc dù nhiều protein nội bào được sinh tổng hợp bên trongtế bào chấtvà ở các vị trí liên kết với màng tế bào hoặc protein được tiết ra từmạng lưới nội chất,chi tiết cụ thể bằng cách nào mà các protein được định hướng (protein targeting) đến những bào quan cụ thể hoặc các cấu trúc của tế bào vẫn chưa được hiểu rõ. Một kỹ thuật hữu ích để đánh giá sự khu trú tế bào bằng cách sử dụng kỹ thuật di truyền nhằm biểu hiện bên trong một tế bào một protein dung hợp (fusion protein,haychimera,một protein được tạo ra thông qua việc nối hai hoặc nhiều đoạn gene với nhau mà ban đầu mã hóa cho từng protein riêng biệt) chứa protein tự nhiên cần nghiên cứu mà nó liên kết với một "thành phần báo cáo" như protein huỳnh quang xanh (GFP).[48]Vị trí của protein dung hợp bên trong tế bào có thể dễ dàng nhận ra và chụp ảnh dướikính hiển vi,[49]như minh họa ở hình bên cạnh.
Những phương pháp khác nhằm lý giải vị trí của protein trong tế bào đòi hỏi sử dụng các ngăn nội bào chỉ thị đã biết cho từng vùng chuyên biệt như lưới nội chất ER,bộ máy Golgi,thực bào,không bào,ty thể,lục lạp,màng sinh chất,vv. Bằng cách sử dụng các phân tử đánh dấu huỳnh quang xanh cho những vùng chỉ thị này hoặc của những kháng thể cho những phân tử chỉ thị đã biết, người ta có thể dễ dàng nhận ra vị trí của protein cần nghiên cứu trong tế bào. Ví dụ, kỹ thuật hiển vi huỳnh quang miễn dịch gián tiếp (indirect immunofluorescence) sẽ cho phép huỳnh quang các vị trí và hiển thị chúng. Bột huỳnh quang được sử dụng để đánh dấu các ngăn của tế bào cho các mục đích tương tự.[50]
Có những kỹ thuật khác, ví dụ như kỹ thuậthóa mô miễn dịch(immunohistochemistry) thường lợi dụng một kháng thể của một hay nhiều protein cần nghiên cứu mà liên hợp với các enzyme để thu được hoặc là vị trí phát sáng hoặc là tín hiệu tạo sắc tố (chromogeneic) mà các nhà nghiên cứu có thể so sánh giữa các mẫu, cho phép họ thu thập thông tin về vị trí của protein. Một kỹ thuật ứng dụng khác làđồng cất phân đoạn(cofractionation) trong gradiensucrose(hoặc những vật liệu khác) sử dụng các bướclọc ly tâm phân đoạn(differential centrifugation).[51]Trong khi kỹ thuật này không cho biết sự đồng khu trú của một khoang của tỷ trọng đã biết và protein quan tâm, nó tăng tỷ lệ tinh khiết, và tuân theo các nghiên cứu trên quy mô lớn.
Cuối cùng, phương pháp tiêu chuẩn vàng để xác định sự khu trú tế bào là bằng kỹ thuật hiển vi điện tử miễn dịch (immunoelectron microscopy). Kỹ thuật này cũng sử dụng một kháng thể với protein cần nghiên cứu, và kết hợp với các kỹ thuật hiển vi điện tử cổ điển khác. Mẫu được chuẩn bị như đối với kiểm tra qua kính hiển vi điện từ thông thường, và sau đó được xử lý bằng một kháng thể với protein quan tâm mà liên hợp với vật liệu có mật độ electron dày đặc, mà thường là vàng. Kỹ thuật này cho phép xác định được chi tiết siêu cấu trúc cũng những vị trí của protein đang cần nghiên cứu.[52]
Thông qua các ứng dụng kỹ thuật di truyền khác được biết đến như gây đột biến định hướng điểm (site-directed mutagenesis), các nhà nghiên cứu có thể thay đổi được trình tự của protein và do đó đến cấu trúc của nó, sự khu trú tế bào, và tính nhạy cảm đối với sự điều hòa biểu hiện. Kỹ thuật này thậm chí cho phép đính những phân tử amino acid không có trong tự nhiên vào protein, bằng sử dụng các tRNA được sửa đổi,[53]và có thể cho phép đánh giá sự hợp lý trongthiết kếprotein mới với những tính chất nổi bật.[54]
Proteomic[sửa|sửa mã nguồn]
Tổng toàn bộ protein ở một thời điểm có trong một tế bào hoặc loại tế bào được gọi là bộ protein hayproteome,và ngành nghiên cứu tập hợp dữ liệu lớn như thế gọi làproteomic,được đặt tên tương tự như của ngànhgeneomic.Các kỹ thuật thực nghiệm quan trọng của proteomic bao gồm điện di trên keo hai chiều (2D gel electrophoresis),[55]cho phép tách số lượng lớn các protein,phương pháp khối phổ,[56]cho phép nhanh chóng nhận ra loại protein và trình tự các peptide (hầu hết sau khi tiêu hóa trên gel (in-gel digestion)),protein microarray,[57]cho phép xác định mức độ tương đối của một số lớn các protein có mặt trong một tế bào, và sàng lọc thể lai hai mảnh (two-hybrid screening), cho phép khám phá một cách có hệ thống tương tác protein-protein.[58]Tổng toàn bộ các tương tác sinh học khả dĩ như những tương tác này gọi làinteractome.[59]Nỗ lực có hệ thống nhằm xác định cấu trúc của protein biểu diễn cho mỗi hình dạng gập khả dĩ gọi là ngành nghiên cứu bộ gene cấu trúc (structural geneomics).[60]
Tin sinh học[sửa|sửa mã nguồn]
Rất nhiều phương pháp tính toán đã được phát triển để phân tích cấu trúc, chức năng, và sự tiến hóa của protein.
Nhờ sự phát triển của những công cụ này giúp đem lại lượng lớn dữ liệu thu thập được về bộ gene và bộ protein (proteomic) ở nhiều sinh vật, bao gồmbộ gene người.Không thể đơn giản chỉ nghiên cứu bằng thực nghiệm mọi protein được, do vậy chỉ có một vài phân tử được nghiên cứu trong phòng thí nghiệm trong khi các công cụ tính toán được sử dụng để ngoại suy ra những protein tương tự. Những proteintương đồngnày có thể nhận ra với độ chính xác cao ở những sinh vật có liên hệ xa bởi phương pháp bắt cặp trình tự (sequence alignment). Bộ gene và trình tự gene được tìm kiếm bằng nhiều công cụ khác nhau cho những tính chất nhất định. Các công cụ nhận diện trình tự (sequence profiling tools) có khả năng tìm ra những vị tríenzyme giới hạn,khung đọc mở(open reading frame) ở trình tựnucleotide,và dự đoán cấu trúc bậc 2.Cây phát sinh chủng loàicó thể xây dựng và các giả thuyết tiến hóa được phát triển nhờ sử dụng các phần mềm chuyên dụng nhưClustalWkhi xem xét tổ tiên của những sinh vật hiện đại và các gene mà chúng biểu hiện. Lĩnh vực tin sinh học hiện nay trở thành công cụ quý giá cho phân tích gene và protein.
Dự đoán cấu trúc và mô phỏng[sửa|sửa mã nguồn]

Chú thích:(a)Cấu trúc chính là trình tự các amino acid tạo nên chuỗi polypeptide(trên cùng bên trái).
(b)Cấu trúc bậc hai, có thể ở dạng chuỗi xoắn Alpha hoặc tấm xếp nếp beta, được duy trì bởi các liên kết hydro giữa các amino acid ở các vùng khác nhau của sợi polypeptide ban đầu(giữa bên trái và giữa bên phải).
(c)Cấu trúc bậc ba xảy ra do sự gấp nếp và liên kết của cấu trúc bậc hai.(dưới cùng bên trái)
(d)Cấu trúc bậc bốn xảy ra do tương tác giữa hai hoặc nhiều đơn vị con bậc ba. Ví dụ được hiển thị ở đây là hemoglobin, một protein trong tế bào hồng cầu vận chuyển oxy đến các mô cơ thể(dưới cùng bên phải).
Bổ sung cho ngành bộ gene cấu trúc (structural geneomic), lĩnh vựcdự đoán cấu trúc proteinphát triển các mô hình toán học hữu hiệu về protein để dự đoán lý thuyết dựa trên công cụ tính toán về cấu trúc của chúng, thay vì phát hiện cấu trúc protein trong phòng thí nghiệm.[61]Phương pháp dự đoán cấu trúc thành công nhất, gọi là mô hình đồng đẳng (homology modeling), dựa trên sự tồn tại của một cấu trúc "khuôn mẫu" với trình tự giống với của protein đang được xây dựng mô hình; mục đích của bộ gene cấu trúc là cung cấp hình ảnh biểu diễn thỏa đáng trong các cấu trúc đã biết để mô hình hóa nhiều nhất có thể các cấu trúc còn chưa được biết.[62]Mặc dù mục tiêu tạo ra những mô hình chính xác vẫn còn là thử thách khi chỉ những khuôn mẫu có liên hệ xa là mới có, người ta đã đề xuất rằng sựbắt cặp trình tựlà nút thắt cổ chai trong quá trình này, khi có thể tạo ra những mô hình khá chính xác nếu đã biết một trình tự bắt cặp "hoàn hảo".[63]Nhiều phương pháp dự đoán cấu trúc được ứng dụng trong lĩnh vựckỹ thuật protein,trong đó những protein gập lạ đã được thiết kế.[64]Một vấn đề tính toán phức tạp hợp đó là dự đoán tương tác liên phân tử, như trong sự cập bến của phân tử (molecular docking) và dự đoán tương tác protein–protein.[65]
Những mô hình toán học để mô phỏng tiến trình động lực của sựgập proteinvà liên kết protein bao gồmcơ học phân tử,và đặc biệt làđộng lực học phân tử.Kỹ thuật Monte Carlotrang bị cho các tính toán, mà dựa trênđiện toán phân tánvà tính toán song song tiên tiến (ví dụ như dự ánFolding@home[66]thực hiện mô phỏng cấu trúc phân tử dựa trênGPU). Mô phỏngin silicokhám phá ra sự gập của những miền nhỏ xoắn α trên protein như đầu củavillin[67]và protein phụ choHIV.[68]Các phương pháp lai kết hợp chuẩn động lực học phân tử với toán học củacơ học lượng tửđể khám phá các trạng thái điện tử củarhodopsin.[69]
Dự đoán protein mất trật tự và cấu trúc không cố định[sửa|sửa mã nguồn]
Nhiều protein (ở sinh vật nhân thực Eucaryota ~33%) chứa nhiều đoạn với cấu trúc không ổn định nhưng có chức năng sinh học và được phân loại thànhprotein mất trật tự nội tại(intrinsically disordered proteins).[70]Dự đoán và phân tích protein mất trật tự do đó là một mảng quan trọng của nghiên cứu cấu trúc protein.[71]
Dinh dưỡng[sửa|sửa mã nguồn]


Hầu hết cácvi sinh vậtvà thực vật có thể sinh tổng hợp tất cả 20 amino acid chính, trong khi động vật (bao gồm con người) phải lấy một số amino acid từthức ăn.[27]Các amino acid mà một sinh vật không thể tự tổng hợp được gọi là cácamino acid thiết yếu.Những enzyme quan trọng mà tham gia tổng hợp một số amino acid không có ở động vật — nhưaspartokinase,tham gia xúc tác ở phản ứng đầu tiên của quá trình tổng hợplysine,methionine,vàthreoninetừaspartate.Nếu các amino acid có mặt trong môi trường, vi sinh vật có thể bảo tồn được năng lượng bằng cách tiếp nhận amino acid từ môi trường xung quanh chúng và điều hòa giảm sinh tổng hợp các amino acid này trong chu trình sinh dưỡng của nó.
Ở động vật, amino acid nhận được thông qua tiêu thụ thức ăn chứa protein. Protein tiêu hóa sau đó bị phân tách thành các amino acid nhờ quá trìnhtiêu hóa,mà điển hình bao gồm sựbiến tínhcủa protein do tiếp xúc vớiacidvà bịthủy phânbởi enzyme xúc tácprotease.Một số amino acid tiêu thụ được sử dụng để sinh tổng hợp protein mới, trong khi những amino acid khác chuyển hóa thànhglucosenhờ quá trìnhtân tạo glucose(gluconeogenesis), hoặc tham gia vàochu trình acid citric.Việc sử dụng protein như là nhiên liệu rất quan trọng trong điều kiệnthiếu ănkhi nó cho phép chính protein trong cơ thể được sử dụng để hỗ trợ sự sống, đặc biệt như được tìm thấy ởcơ.[72]
Lịch sử và từ nguyên[sửa|sửa mã nguồn]
Protein được công nhận là một lớp các phân tử sinh học chuyên biệt bởiAntoine Fourcroyvà những người khác vào thế kỷ 18, phân biệt nhờ vào đặc tính của phân tử nhưđông đặchoặclên bông(flocculate) khi xử lý qua nhiệt hoặc acid.[73]Các mẫu được chú ý ở thời điểm đó bao gồm albumin từlòng trắngtrứng,serum albuminmáu,fibrin,vàglutenhạt lúa mì.
Nhà hóa học người Hà LanGerardus Johannes Mulderlà người đầu tiên miêu tả về protein và tên gọi này được nhà hóa học người Thụy ĐiểnJöns Jacob Berzeliusđặt vào năm 1838.[74][75]Mulder thực hiện các phân tích sơ cấp về những protein phổ biến và ông tìm thấy gần như mọi protein có cùng mộtcông thức thực nghiệm,C400H620N100O120P1S1.[76]Ông đi đến kết luận sai lầm rằng chúng phải là hỗn hợp của một loại phân tử rất lớn. Thuật ngữ "protein" dùng cho những phân tử này do Berzelius, một đồng nghiệp của Mulder, đề xuất; protein bắt nguồn trongtiếng Hy Lạpπρώτειος (proteios), nghĩa là "sơ cấp",[77]"dẫn đầu", hoặc "đứng phía trước",[78]+-in.Mulder nhận biết được các sản phẩm của sự thoái hóa protein nhưamino acidleucinmà ông tìm thấy (một cách gần đúng) trọng lượng phân tử bằng 131Da.[76]
Những nhà dinh dưỡng học thời đầu nhưCarl von Voitngười Đức tin rằng protein là thành phần dinh dưỡng quan trọng nhất để duy trì cấu trúc của cơ thể, bởi vì niềm tin phổ biến thời đấy cho rằng "máu tươi tạo máu tươi" ( "flesh makes flesh").[79]Karl Heinrich Ritthausenthêm vào các dạng protein đã biết bao gồmacid glutamic.Ở Trung tâm thí nghiệm nông nghiệp Connecticut (Connecticut Agricultural Experiment Station), nhà hóa họcThomas Burr Osborneđã thực hiện đánh giá chi tiết các loại protein có trong cây trồng. Nghiên cứu cùngLafayette Mendelvà áp dụngquy luật cực tiểu của Liebigkhi nuôichuột thí nghiệm,họ đã thiết lập lên danh mục các amino acid dinh dưỡng thiết yếu. Công trình này được tiếp tục nghiên cứu và trao đổi hợp tác vớiWilliam Cumming Rose.Hiểu biết protein là các chuỗipolypeptidethông qua nghiên cứu của hai nhà hóa họcngười ĐứcFranz HofmeistervàHermann Emil Fischervào năm 1902.[80][81]Vai trò trung tâm của protein làmenzymexúc tác trong sinh vật sống không được đánh giá đầy đủ cho đến tận năm 1926, khiJames B. Sumnerchỉ ra enzymeureasethực chất là một protein.[82]
Sự khó khăn trong quá trình tinh sạch protein thành lượng lớn khiến các nhà hóa sinh nghiên chúng rất khó khăn ở thời điểm đầu. Do vậy, những nghiên cứu ban đầu tập trung vào những protein được tinh lọc lượng lớn, ví dụ như củamáu,lòng trứng trắng, nhiềuđộc tốkhác nhau, và các enzyme tiêu hóa / trao đổi chất lấy từ cáclò sát sinh.Trong thập niên 1950, công ty Armour Hot Dog Co. đã lọc được khoảng 1 kg thuần khiếtribonuclease Atừ tuyến tụy của bò và cung cấp miễn phí cho các nhà khoa học; nhờ thế mà ribonuclease A trở thành mục tiêu nghiên cứu chính của hóa sinh trong hàng thập kỷ sau đó.[76]

Linus Paulingđược ghi nhận là đã đưa ra mô hình dự đoán thành công cấu trúc bậc 2 của các protein đối xứng đều dựa trên liên kết hiđrô, ý tưởng ông lấy từWilliam Astburyvào năm 1933.[83]Công trình nghiên cứu sau đó củaWalter Kauzmannvềsự biến tính,[84][85]dựa một phần trên nghiên cứu trước đây củaKaj Linderstrøm-Lang,[86]đóng góp vào hiểu biết quá trình gập protein (protein folding) và cấu trúc trung gian bởi tương táckị nước.
Protein đượcgiải trình tựđầu tiên làinsulin,doFrederick Sangerthực hiện vào năm 1949. Sanger đã xác định đúng trình tự các amino acid của insulin, vì thế chứng minh một cách thuyết phục rằng các protein là những polymer mạch thẳng chứa các amino acid hơn là các mạch nhánh,hệ keo,hoặccyclol.[87]Ông giành giải Nobel Hóa học cho thành tựu này vào năm 1958.[88]
Cấu trúc proteinlần đầu tiên được quan sát là củahemoglobinvàmyoglobin,doMax PerutzvàSir John Cowdery Kendrew,thực hiện một cách độc lập vào năm 1958.[89][90]Tính đến năm 2017[cập nhật],ngân hàng dữ liệu protein(Protein Data Bank) chứa hơn 126.060 protein có cấu trúc được quan sát ở cấp độ nguyên tử.[91]Trong thời gian gần đây, kỹ thuật hiển vi electron lạnh (cryo-electron microscopy) đối với quá trình lắp ráp đại phân tử[92]và tính toán dự đoán cấu trúc protein (computational protein structure prediction) của cácmiền proteinnhỏ (small protein domain)[93]là hai phương pháp tiếp cận chính trong nghiên cứu cấu trúc protein.
Tham khảo[sửa|sửa mã nguồn]
- ^Nelson DL, Cox MM (2005).Lehninger's Principles of Biochemistry(ấn bản 4). New York, New York: W. H. Freeman and Company.
- ^Gutteridge A, Thornton JM (2005).“Understanding nature's catalytic toolkit”.Trends in Biochemical Sciences.30(11): 622–29.doi:10.1016/j.tibs.2005.09.006.PMID16214343.
- ^Murrayet al., p. 19.
- ^Murrayet al., p. 31.
- ^abcLodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipurksy SL, Darnell J (2004).Molecular Cell Biology(ấn bản 5). New York, New York: WH Freeman and Company.
- ^Milo, Ron (ngày 1 tháng 12 năm 2013).“What is the total number of protein molecules per cell volume? A call to rethink some published values”.BioEssays(bằng tiếng Anh).35(12): 1050–1055.doi:10.1002/bies.201300066.ISSN1521-1878.PMC3910158.PMID24114984.
- ^Wu, Linfeng; Candille, Sophie I.; Choi, Yoonha; Xie, Dan; Jiang, Lihua; Li-Pook-Than, Jennifer; Tang, Hua; Snyder, Michael (2013).“Variation and genetic control of protein abundance in humans”.Nature.499(7456): 79–82.Bibcode:2013Natur.499...79W.doi:10.1038/nature12223.PMC3789121.PMID23676674.
- ^Kozlowski, Lukasz P. (2016). “Proteome-pI: proteome isoelectric point database”.Nucleic Acids Research.45(D1): D1112–D1116.doi:10.1093/nar/gkw978.ISSN0305-1048.
- ^abvan Holde and Mathews, pp. 1002–42.
- ^Dobson CM (2000). “The nature and significance of protein folding”. Trong Pain RH (biên tập).Mechanisms of Protein Folding.Oxford, Oxfordshire: Oxford University Press. tr. 1–28.ISBN0-19-963789-X.
- ^Fulton A, Isaacs W (1991).“Titin, a huge, elastic sarcomeric protein with a probable role in morphogenesis”.BioEssays.13(4): 157–61.doi:10.1002/bies.950130403.PMID1859393.
- ^Bruckdorfer T, Marder O, Albericio F (2004). “From production of peptides in milligram amounts for research to multi-tons quantities for drugs of the future”.Current Pharmaceutical Biotechnology.5(1): 29–43.doi:10.2174/1389201043489620.PMID14965208.
- ^Schwarzer D, Cole P (2005). “Protein semisynthesis and expressed protein ligation: chasing a protein's tail”.Current Opinion in Chemical Biology.9(6): 561–69.doi:10.1016/j.cbpa.2005.09.018.PMID16226484.
- ^Kent SB (2009).“Total chemical synthesis of proteins”.Chemical Society Reviews.38(2): 338–51.doi:10.1039/b700141j.PMID19169452.
- ^Murrayet al., p. 36.
- ^Murrayet al., p. 37.
- ^Murrayet al., pp. 30–34.
- ^van Holde and Mathews, pp. 368–75.
- ^abLodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipurksy SL, Darnell J (2016).Molecular Cell Biology(ấn bản 8). W.H Freeman. tr. 72.ISBN978-1464183393.Quản lý CS1: sử dụng tham số tác giả (liên kết)
- ^van Holde and Mathews, pp. 165–85.
- ^Fernández A, Scott R (2003).“Dehydron: a structurally encoded signal for protein interaction”.Biophysical Journal.85(3): 1914–28.Bibcode:2003BpJ....85.1914F.doi:10.1016/S0006-3495(03)74619-0.PMC1303363.PMID12944304.
- ^Branden and Tooze, pp. 340–41.
- ^Gonen T, Cheng Y, Sliz P, Hiroaki Y, Fujiyoshi Y, Harrison SC, Walz T (2005).“Lipid-protein interactions in double-layered two-dimensional AQP0 crystals”.Nature.438(7068): 633–38.Bibcode:2005Natur.438..633G.doi:10.1038/nature04321.PMC1350984.PMID16319884.
- ^Standley DM, Kinjo AR, Kinoshita K, Nakamura H (2008).“Protein structure databases with new web services for structural biology and biomedical research”.Briefings in Bioinformatics.9(4): 276–85.doi:10.1093/bib/bbn015.PMID18430752.
- ^Walian P, Cross TA, Jap BK (2004).“Structural genomics of membrane proteins”.Genome Biology.5(4): 215.doi:10.1186/gb-2004-5-4-215.PMC395774.PMID15059248.Bản gốclưu trữ ngày 2 tháng 1 năm 2016.Truy cập ngày 18 tháng 4 năm 2017.
- ^Sleator RD. (2012). “Prediction of protein functions”.Methods in Molecular Biology.Methods in Molecular Biology.815:15–24.doi:10.1007/978-1-61779-424-7_2.ISBN978-1-61779-423-0.PMID22130980.
- ^abVoet D, Voet JG. (2004).BiochemistryVol 1 3rd ed. Wiley: Hoboken, NJ.
- ^Sankaranarayanan R, Moras D (2001). “The fidelity of the translation of the genetic code”.Acta Biochimica Polonica.48(2): 323–35.PMID11732604.
- ^van Holde and Mathews, pp. 830–49.
- ^Copland JA, Sheffield-Moore M, Koldzic-Zivanovic N, Gentry S, Lamprou G, Tzortzatou-Stathopoulou F, Zoumpourlis V, Urban RJ, Vlahopoulos SA (2009). “Sex steroid receptors in skeletal differentiation and epithelial neoplasia: is tissue-specific intervention possible?”.BioEssays.31(6): 629–41.doi:10.1002/bies.200800138.PMID19382224.
- ^Samarin S, Nusrat A (2009). “Regulation of epithelial apical junctional complex by Rho family GTPases”.Frontiers in Bioscience.14(14): 1129–42.doi:10.2741/3298.PMID19273120.
- ^Bailey, Regina.“Protein function”.thoughtco.Bản gốclưu trữ ngày 19 tháng 4 năm 2017.Truy cập ngày 16 tháng 4 năm 2017.
- ^“What are proteins and what do they do?”.National Institutes of Health.Bản gốclưu trữ ngày 22 tháng 1 năm 2020.Truy cập ngày 16 tháng 4 năm 2017.
- ^Bairoch A (2000).“The ENZYME database in 2000”(PDF).Nucleic Acids Research.28(1): 304–305.doi:10.1093/nar/28.1.304.PMC102465.PMID10592255.Bản gốc(PDF)lưu trữ ngày 1 tháng 6 năm 2011.
- ^Radzicka A, Wolfenden R (1995). “A proficient enzyme”.Science.267(5194): 90–93.Bibcode:1995Sci...267...90R.doi:10.1126/science.7809611.PMID7809611.
- ^EBI External Services (ngày 20 tháng 1 năm 2010).“The Catalytic Site Atlas at The European Bioinformatics Institute”.Ebi.ac.uk.Bản gốclưu trữ ngày 3 tháng 8 năm 2013.Truy cập ngày 16 tháng 1 năm 2011.
- ^Pickel B, Schaller A (2013). “Dirigent proteins: molecular characteristics and potential biotechnological applications”.Applied Microbiology and Biotechnology.97(19): 8427–8438.doi:10.1007/s00253-013-5167-4.PMID23989917.
- ^Branden and Tooze, pp. 251–81.
- ^van Holde and Mathews, pp. 247–50.
- ^van Holde and Mathews, pp. 220–29.
- ^Rüdiger H, Siebert HC, Solís D, Jiménez-Barbero J, Romero A, von der Lieth CW, Diaz-Mariño T, Gabius HJ (2000). “Medicinal chemistry based on the sugar code: fundamentals of lectinology and experimental strategies with lectins as targets”.Current Medicinal Chemistry.7(4): 389–416.doi:10.2174/0929867003375164.PMID10702616.
- ^Branden and Tooze, pp. 232–34.
- ^van Holde and Mathews, pp. 178–81.
- ^van Holde and Mathews, pp. 258–64; 272.
- ^Murrayet al., pp. 21–24.
- ^Hey J, Posch A, Cohen A, Liu N, Harbers A (2008). “Fractionation of complex protein mixtures by liquid-phase isoelectric focusing”.Methods in Molecular Biology.Methods in Molecular Biology™.424:225–39.doi:10.1007/978-1-60327-064-9_19.ISBN978-1-58829-722-8.PMID18369866.
- ^Terpe K (2003). “Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems”.Applied Microbiology and Biotechnology.60(5): 523–33.doi:10.1007/s00253-002-1158-6.PMID12536251.
- ^Stepanenko OV, Verkhusha VV, Kuznetsova IM, Uversky VN, Turoverov KK (2008).“Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes”.Current Protein & Peptide Science.9(4): 338–69.doi:10.2174/138920308785132668.PMC2904242.PMID18691124.
- ^Yuste R (2005). “Fluorescence microscopy today”.Nature Methods.2(12): 902–904.doi:10.1038/nmeth1205-902.PMID16299474.
- ^Margolin W (2000).“Green fluorescent protein as a reporter for macromolecular localization in bacterial cells”.Methods (San Diego, Calif.).20(1): 62–72.doi:10.1006/meth.1999.0906.PMID10610805.
- ^Walker JH, Wilson K (2000).Principles and Techniques of Practical Biochemistry.Cambridge, UK: Cambridge University Press. tr.287–89.ISBN0-521-65873-X.
- ^Mayhew TM, Lucocq JM (2008).“Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review”.Histochemistry and Cell Biology.130(2): 299–313.doi:10.1007/s00418-008-0451-6.PMC2491712.PMID18553098.
- ^Hohsaka T, Sisido M (2002). “Incorporation of non-natural amino acids into proteins”.Current Opinion in Chemical Biology.6(6): 809–15.doi:10.1016/S1367-5931(02)00376-9.PMID12470735.
- ^Cedrone F, Ménez A, Quéméneur E (2000). “Tailoring new enzyme functions by rational redesign”.Current Opinion in Structural Biology.10(4): 405–10.doi:10.1016/S0959-440X(00)00106-8.PMID10981626.
- ^Görg A, Weiss W, Dunn MJ (2004). “Current two-dimensional electrophoresis technology for proteomics”.Proteomics.4(12): 3665–85.doi:10.1002/pmic.200401031.PMID15543535.
- ^Conrotto P, Souchelnytskyi S (2008). “Proteomic approaches in biological and medical sciences: principles and applications”.Experimental Oncology.30(3): 171–80.PMID18806738.
- ^Joos T, Bachmann J (2009).“Protein microarrays: potentials and limitations”.Frontiers in Bioscience.14(14): 4376–85.doi:10.2741/3534.PMID19273356.
- ^Koegl M, Uetz P (2007).“Improving yeast two-hybrid screening systems”.Briefings in Functional Genomics & Proteomics.6(4): 302–12.doi:10.1093/bfgp/elm035.PMID18218650.Bản gốclưu trữ ngày 15 tháng 4 năm 2013.Truy cập ngày 18 tháng 4 năm 2017.
- ^Plewczyński D, Ginalski K (2009). “The interactome: predicting the protein–protein interactions in cells”.Cellular & Molecular Biology Letters.14(1): 1–22.doi:10.2478/s11658-008-0024-7.PMID18839074.
- ^Zhang C, Kim SH (2003). “Overview of structural genomics: from structure to function”.Current Opinion in Chemical Biology.7(1): 28–32.doi:10.1016/S1367-5931(02)00015-7.PMID12547423.
- ^Zhang Y (2008).“Progress and challenges in protein structure prediction”.Current Opinion in Structural Biology.18(3): 342–48.doi:10.1016/j.sbi.2008.02.004.PMC2680823.PMID18436442.
- ^Xiang Z (2006).“Advances in homology protein structure modeling”.Current Protein and Peptide Science.7(3): 217–27.doi:10.2174/138920306777452312.PMC1839925.PMID16787261.
- ^Zhang Y, Skolnick J (2005).“The protein structure prediction problem could be solved using the current PDB library”.Proceedings of the National Academy of Sciences of the United States of America.102(4): 1029–34.Bibcode:2005PNAS..102.1029Z.doi:10.1073/pnas.0407152101.PMC545829.PMID15653774.
- ^Kuhlman B, Dantas G, Ireton GC, Varani G, Stoddard BL, Baker D (2003).“Design of a novel globular protein fold with atomic-level accuracy”.Science.302(5649): 1364–68.Bibcode:2003Sci...302.1364K.doi:10.1126/science.1089427.PMID14631033.
- ^Ritchie DW (2008). “Recent progress and future directions in protein–protein docking”.Current Protein and Peptide Science.9(1): 1–15.doi:10.2174/138920308783565741.PMID18336319.
- ^Scheraga HA, Khalili M, Liwo A (2007). “Protein-folding dynamics: overview of molecular simulation techniques”.Annual Review of Physical Chemistry.58:57–83.Bibcode:2007ARPC...58...57S.doi:10.1146/annurev.physchem.58.032806.104614.PMID17034338.
- ^Zagrovic B, Snow CD, Shirts MR, Pande VS (2002). “Simulation of folding of a small Alpha -helical protein in atomistic detail using worldwide-distributed computing”.Journal of Molecular Biology.323(5): 927–37.doi:10.1016/S0022-2836(02)00997-X.PMID12417204.
- ^Herges T, Wenzel W (2005). “In silicofolding of a three helix protein and characterization of its free-energy landscape in an all-atom force field”.Physical Review Letters.94(1): 018101.Bibcode:2005PhRvL..94a8101H.doi:10.1103/PhysRevLett.94.018101.PMID15698135.
- ^Hoffmann M, Wanko M, Strodel P, König PH, Frauenheim T, Schulten K, Thiel W, Tajkhorshid E, Elstner M (2006). “Color tuning in rhodopsins: the mechanism for the spectral shift between bacteriorhodopsin and sensory rhodopsin II”.Journal of the American Chemical Society.128(33): 10808–18.doi:10.1021/ja062082i.PMID16910676.
- ^Ward, J. J.; Sodhi, J. S.; McGuffin, L. J.; Buxton, B. F.; Jones, D. T. (2004). “Prediction and functional analysis of native disorder in proteins from the three kingdoms of life”.Journal of Molecular Biology.337(3): 635–45.doi:10.1016/j.jmb.2004.02.002.PMID15019783.
- ^Peter Tompa; Alan Fersht (ngày 18 tháng 11 năm 2009).Structure and Function of Intrinsically Disordered Proteins.CRC Press.ISBN978-1-4200-7893-0.
- ^Brosnan J (tháng 6 năm 2003).“Interorgan amino acid transport and its regulation”.Journal of Nutrition.133(6 Suppl 1): 2068S–72S.PMID12771367.
- ^Thomas Burr Osborne (1909):The Vegetable Proteins,History pp 1 to 6, from archive.org
- ^Bulletin des Sciences Physiques et Naturelles en Néerlande (1838). pg 104.SUR LA COMPOSITION DE QUELQUES SUBSTANCES ANIMALES
- ^Hartley Harold (1951). “Origin of the Word 'Protein.'”.Nature.168(4267): 244–244.Bibcode:1951Natur.168..244H.doi:10.1038/168244a0.
- ^abcPerrett D (2007). “From 'protein' to the beginnings of clinical proteomics”.Proteomics: Clinical Applications.1(8): 720–38.doi:10.1002/prca.200700525.PMID21136729.
- ^New Oxford Dictionary of English
- ^Reynolds JA, Tanford C (2003).Nature's Robots: A History of Proteins (Oxford Paperbacks).New York, New York: Oxford University Press. tr.15.ISBN0-19-860694-X.
- ^Bischoff TLW, Voit, C (1860).Die Gesetze der Ernaehrung des Pflanzenfressers durch neue Untersuchungen festgestellt(bằng tiếng Đức). Leipzig, Heidelberg.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- ^“Hofmeister, Franz”.encyclopedia.Truy cập ngày 4 tháng 4 năm 2017.
- ^“Protein, section: Classification of protein”.britannica.Truy cập ngày 4 tháng 4 năm 2017.
- ^Sumner JB (1926).“The isolation and crystallization of the enzyme urease. Preliminary paper”(PDF).Journal of Biological Chemistry.69(2): 435–41.
- ^Pauling L, Corey RB, Branson HR (1951).“The structure of proteins: two hydrogen-bonded helical configurations of the polypeptide chain”(PDF).Proceedings of the National Academy of Sciences of the United States of America.37(5): 235–40.Bibcode:1951PNAS...37..235P.doi:10.1073/pnas.37.5.235.PMC1063348.PMID14834145.
- ^Kauzmann W (1956). “Structural factors in protein denaturation”.Journal of Cellular Physiology. Supplement.47(Suppl 1): 113–31.doi:10.1002/jcp.1030470410.PMID13332017.
- ^Kauzmann W (1959). “Some factors in the interpretation of protein denaturation”.Advances in Protein Chemistry.Advances in Protein Chemistry.14:1–63.doi:10.1016/S0065-3233(08)60608-7.ISBN978-0-12-034214-3.PMID14404936.
- ^Kalman SM, Linderstrom-Lang K, Ottesen M, Richards FM (1955). “Degradation of ribonuclease by subtilisin”.Biochimica et Biophysica Acta.16(2): 297–99.doi:10.1016/0006-3002(55)90224-9.PMID14363272.
- ^Sanger F (1949).“The terminal peptides of insulin”.Biochemical Journal.45(5): 563–74.PMC1275055.PMID15396627.
- ^Sanger F. (1958),Nobel lecture: The chemistry of insulin(PDF),Nobelprize.org
- ^Muirhead H, Perutz M (1963). “Structure of hemoglobin. A three-dimensional fourier synthesis of reduced human hemoglobin at 5.5 Å resolution”.Nature.199(4894): 633–38.Bibcode:1963Natur.199..633M.doi:10.1038/199633a0.PMID14074546.
- ^Kendrew J, Bodo G, Dintzis H, Parrish R, Wyckoff H, Phillips D (1958). “A three-dimensional model of the myoglobin molecule obtained by X-ray analysis”.Nature.181(4610): 662–66.Bibcode:1958Natur.181..662K.doi:10.1038/181662a0.PMID13517261.
- ^“RCSB Protein Data Bank”.Bản gốclưu trữ ngày 18 tháng 4 năm 2015.Truy cập ngày 5 tháng 4 năm 2014.
- ^Zhou ZH (2008).“Towards atomic resolution structural determination by single-particle cryo-electron microscopy”.Current Opinion in Structural Biology.18(2): 218–28.doi:10.1016/j.sbi.2008.03.004.PMC2714865.PMID18403197.
- ^Keskin O, Tuncbag N, Gursoy A (2008). “Characterization and prediction of protein interfaces to infer protein-protein interaction networks”.Current Pharmaceutical Biotechnology.9(2): 67–76.doi:10.2174/138920108783955191.PMID18393863.
Sách[sửa|sửa mã nguồn]
- Branden C, Tooze J (1999).Introduction to Protein Structure.New York: Garland Pub.ISBN0-8153-2305-0.
- Murray RF, Harper HW, Granner DK, Mayes PA, Rodwell VW (2006).Harper's Illustrated Biochemistry.New York: Lange Medical Books/McGraw-Hill.ISBN0-07-146197-3.
- Van Holde KE, Mathews CK (1996).Biochemistry.Menlo Park, California: Benjamin/Cummings Pub. Co., Inc.ISBN0-8053-3931-0.
Liên kết ngoài[sửa|sửa mã nguồn]
| Wikimedia Commons có thêm hình ảnh và phương tiện truyền tải vềProtein. |
| Traproteintrong từ điển mở tiếng ViệtWiktionary |
Cơ sở dữ liệu và các dự án[sửa|sửa mã nguồn]
- The Protein Naming UtilityLưu trữ2012-12-21 tạiArchive.today
- Human Protein Atlas
- NCBI Entrez Protein database
- NCBI Protein Structure database
- Human Protein Reference DatabaseLưu trữ2006-04-24 tạiWayback Machine
- Human ProteinpediaLưu trữ2007-03-14 tạiWayback Machine
- Folding@Home (Stanford University)Lưu trữ2012-09-08 tạiWayback Machine
- Comparative Toxicogeneomics Databasecurates protein–chemical interactions, as well as gene/protein–disease relationships and chemical-disease relationships.
- Bioinformatic Harvester[liên kết hỏng]A Meta search engine (29 databases) for gene and protein information.
- Protein Databank in Europe(see alsoPDBeQuips[liên kết hỏng],short articles and tutorials on interesting PDB structures)
- Research Collaboratory for Structural Bioinformatics(see alsoMolecule of the MonthLưu trữ2020-07-24 tạiWayback Machine,presenting short accounts on selected proteins from the PDB)
- Proteopedia – Life in 3D:rotatable, zoomable 3D model with wiki annotations for every known protein molecular structure.
- UniProt the Universal Protein ResourceLưu trữ2008-06-08 tạiWayback Machine
- neXtProt – Exploring the universe of human proteins:human-centric protein knowledge resource
- Multi-Omics Profiling Expression Database: MOPEDLưu trữ2014-10-22 tạiWayback Machinehuman and model organism protein/gene knowledge and expression data
Các website giáo dục[sửa|sửa mã nguồn]
- "An Introduction to Proteins"from HOPES (Huntington's Disease Outreach Project for Education at Stanford)
- Proteins: Biogenesis to Degradation – The Virtual Library of Biochemistry and Cell BiologyLưu trữ2005-02-19 tạiWayback Machine
- Alphabet of Protein Structures